+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9192 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron microscopy structure of Plasmodium falciparum Rh5/CyRPA/Ripr invasion complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Invasion Ligand Protein complex / CELL INVASION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報microneme lumen / microneme / symbiont entry into host / apical part of cell / cytoplasmic vesicle / host extracellular space / host cell plasma membrane / protein-containing complex / extracellular region / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |    | |||||||||
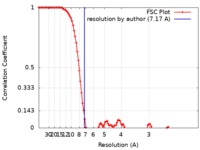
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.17 Å | |||||||||
 データ登録者 データ登録者 | Wilson W / Zhiheng Y | |||||||||
| 資金援助 |  オーストラリア, 1件 オーストラリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: Structure of Plasmodium falciparum Rh5-CyRPA-Ripr invasion complex. 著者: Wilson Wong / Rick Huang / Sebastien Menant / Chuan Hong / Jarrod J Sandow / Richard W Birkinshaw / Julie Healer / Anthony N Hodder / Usheer Kanjee / Christopher J Tonkin / Denise Heckmann / ...著者: Wilson Wong / Rick Huang / Sebastien Menant / Chuan Hong / Jarrod J Sandow / Richard W Birkinshaw / Julie Healer / Anthony N Hodder / Usheer Kanjee / Christopher J Tonkin / Denise Heckmann / Vladislav Soroka / Teit Max Moscote Søgaard / Thomas Jørgensen / Manoj T Duraisingh / Peter E Czabotar / Willem A de Jongh / Wai-Hong Tham / Andrew I Webb / Zhiheng Yu / Alan F Cowman /    要旨: Plasmodium falciparum causes the severe form of malaria that has high levels of mortality in humans. Blood-stage merozoites of P. falciparum invade erythrocytes, and this requires interactions ...Plasmodium falciparum causes the severe form of malaria that has high levels of mortality in humans. Blood-stage merozoites of P. falciparum invade erythrocytes, and this requires interactions between multiple ligands from the parasite and receptors in hosts. These interactions include the binding of the Rh5-CyRPA-Ripr complex with the erythrocyte receptor basigin, which is an essential step for entry into human erythrocytes. Here we show that the Rh5-CyRPA-Ripr complex binds the erythrocyte cell line JK-1 significantly better than does Rh5 alone, and that this binding occurs through the insertion of Rh5 and Ripr into host membranes as a complex with high molecular weight. We report a cryo-electron microscopy structure of the Rh5-CyRPA-Ripr complex at subnanometre resolution, which reveals the organization of this essential invasion complex and the mode of interactions between members of the complex, and shows that CyRPA is a critical mediator of complex assembly. Our structure identifies blades 4-6 of the β-propeller of CyRPA as contact sites for Rh5 and Ripr. The limited contacts between Rh5-CyRPA and CyRPA-Ripr are consistent with the dissociation of Rh5 and Ripr from CyRPA for membrane insertion. A comparision of the crystal structure of Rh5-basigin with the cryo-electron microscopy structure of Rh5-CyRPA-Ripr suggests that Rh5 and Ripr are positioned parallel to the erythrocyte membrane before membrane insertion. This provides information on the function of this complex, and thereby provides insights into invasion by P. falciparum. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9192.map.gz emd_9192.map.gz | 62.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9192-v30.xml emd-9192-v30.xml emd-9192.xml emd-9192.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9192_fsc.xml emd_9192_fsc.xml | 13.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9192.png emd_9192.png | 86.4 KB | ||
| Filedesc metadata |  emd-9192.cif.gz emd-9192.cif.gz | 6.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9192 http://ftp.pdbj.org/pub/emdb/structures/EMD-9192 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9192 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9192 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9192_validation.pdf.gz emd_9192_validation.pdf.gz | 407.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9192_full_validation.pdf.gz emd_9192_full_validation.pdf.gz | 407.2 KB | 表示 | |
| XML形式データ |  emd_9192_validation.xml.gz emd_9192_validation.xml.gz | 11.5 KB | 表示 | |
| CIF形式データ |  emd_9192_validation.cif.gz emd_9192_validation.cif.gz | 15.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9192 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9192 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9192 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9192 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9192.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9192.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : PfRh5/CyRPA/PfRipr complex
| 全体 | 名称: PfRh5/CyRPA/PfRipr complex |
|---|---|
| 要素 |
|
-超分子 #1: PfRh5/CyRPA/PfRipr complex
| 超分子 | 名称: PfRh5/CyRPA/PfRipr complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Cysteine-rich protective antigen
| 分子 | 名称: Cysteine-rich protective antigen / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: isolate 3D7 |
| 分子量 | 理論値: 39.270195 KDa |
| 組換発現 | 生物種: Insect cell expression vector pTIE1 (その他) |
| 配列 | 文字列: RHVFIRTELS FIKNNVPCIR DMFFIYKREL YNICLDDLKG EEDETHIYVQ KKVKDSWITL NDLFKETDLT GRPHIFAYVD VEEIIILLC EDEEFSNRKK DMTCHRFYSN DGKEYNNAEI TISDYILKDK LLSSYVSLPL KIENREYFLI CGVSPYKFKD D NKKDDILC ...文字列: RHVFIRTELS FIKNNVPCIR DMFFIYKREL YNICLDDLKG EEDETHIYVQ KKVKDSWITL NDLFKETDLT GRPHIFAYVD VEEIIILLC EDEEFSNRKK DMTCHRFYSN DGKEYNNAEI TISDYILKDK LLSSYVSLPL KIENREYFLI CGVSPYKFKD D NKKDDILC MASHDKGETW GTKIVIKYDN YKLGVQYFFL RPYISKNDLS FHFYVGDNIN NVKNVNFIEC THEKDLEFVC SN RDFLKDN KVLQDVSTLN DEYIVSYGND NNFAECYIFF NNENSILIKP EKYGNTTAGC YGGTFVKIDE NRALFIYSSS QGI YNIHTI YYANYE UniProtKB: Cysteine-rich protective antigen |
-分子 #2: Reticulocyte binding protein 5
| 分子 | 名称: Reticulocyte binding protein 5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 39.803371 KDa |
| 組換発現 | 生物種: Insect cell expression vector pTIE1 (その他) |
| 配列 | 文字列: IIPHYTFLDY YKHLSYNSIY HKSSTYGKCI AVDAFIKKIN ETYDKVKSKC NDIKNDLIAT IKKLEHPYDI NNKNDDSYRY DISEEIDDK SEETDDETEE VEDSIQDTDS NHTPSNKKKN DLMNRTFKKM MDEYNTKKKK LIKCIKNHEN DFNKICMDMK N YGTNLFEQ ...文字列: IIPHYTFLDY YKHLSYNSIY HKSSTYGKCI AVDAFIKKIN ETYDKVKSKC NDIKNDLIAT IKKLEHPYDI NNKNDDSYRY DISEEIDDK SEETDDETEE VEDSIQDTDS NHTPSNKKKN DLMNRTFKKM MDEYNTKKKK LIKCIKNHEN DFNKICMDMK N YGTNLFEQ LSCYNNNFCN TNGIRYHYDE YIHKLILSVK SKNLNKDLSD MTNILQQSEL LLTNLNKKMG SYIYIDTIKF IH KEMKHIF NRIEYHTKII NDKTKIIQDK IKLNIWRTFQ KDELLKRILD MSNEYSLFIT SDHLRQMLYN TFYSKEKHLN NIF HHLIYV LQMK UniProtKB: Reticulocyte binding protein 5 |
-分子 #3: PfRipr
| 分子 | 名称: PfRipr / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.379692 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #4: PfRipr
| 分子 | 名称: PfRipr / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 443.539 Da |
| 組換発現 | 生物種:  |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8.5 / 詳細: 20 mM Tris, pH 8.5, 150 mM NaCl |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 球面収差補正装置: Microscope was equipped with a Cs corrector with two hexapole elements エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 撮影したグリッド数: 4 / 実像数: 12974 / 平均露光時間: 10.0 sec. / 平均電子線量: 92.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 48077 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)