+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8218 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | MicroED structure of xylanase at 2.3 A resolution | |||||||||
 マップデータ マップデータ | Xylanase | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Hydrolase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報endo-1,4-beta-xylanase activity / endo-1,4-beta-xylanase / xylan catabolic process / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |  Trichoderma reesei (菌類) / Trichoderma reesei (菌類) /  Hypocrea jecorina (菌類) Hypocrea jecorina (菌類) | |||||||||
| 手法 | 電子線結晶学 / クライオ電子顕微鏡法 / 解像度: 2.3 Å | |||||||||
 データ登録者 データ登録者 | de la Cruz MJ / Hattne J | |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2017 ジャーナル: Nat Methods / 年: 2017タイトル: Atomic-resolution structures from fragmented protein crystals with the cryoEM method MicroED. 著者: M Jason de la Cruz / Johan Hattne / Dan Shi / Paul Seidler / Jose Rodriguez / Francis E Reyes / Michael R Sawaya / Duilio Cascio / Simon C Weiss / Sun Kyung Kim / Cynthia S Hinck / Andrew P ...著者: M Jason de la Cruz / Johan Hattne / Dan Shi / Paul Seidler / Jose Rodriguez / Francis E Reyes / Michael R Sawaya / Duilio Cascio / Simon C Weiss / Sun Kyung Kim / Cynthia S Hinck / Andrew P Hinck / Guillermo Calero / David Eisenberg / Tamir Gonen /  要旨: Traditionally, crystallographic analysis of macromolecules has depended on large, well-ordered crystals, which often require significant effort to obtain. Even sizable crystals sometimes suffer from ...Traditionally, crystallographic analysis of macromolecules has depended on large, well-ordered crystals, which often require significant effort to obtain. Even sizable crystals sometimes suffer from pathologies that render them inappropriate for high-resolution structure determination. Here we show that fragmentation of large, imperfect crystals into microcrystals or nanocrystals can provide a simple path for high-resolution structure determination by the cryoEM method MicroED and potentially by serial femtosecond crystallography. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8218.map.gz emd_8218.map.gz | 1010.5 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8218-v30.xml emd-8218-v30.xml emd-8218.xml emd-8218.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8218.png emd_8218.png | 193 KB | ||
| Filedesc metadata |  emd-8218.cif.gz emd-8218.cif.gz | 5.4 KB | ||
| Filedesc structureFactors |  emd_8218_sf.cif.gz emd_8218_sf.cif.gz | 683.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8218 http://ftp.pdbj.org/pub/emdb/structures/EMD-8218 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8218 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8218 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8218_validation.pdf.gz emd_8218_validation.pdf.gz | 557.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8218_full_validation.pdf.gz emd_8218_full_validation.pdf.gz | 556.6 KB | 表示 | |
| XML形式データ |  emd_8218_validation.xml.gz emd_8218_validation.xml.gz | 4.2 KB | 表示 | |
| CIF形式データ |  emd_8218_validation.cif.gz emd_8218_validation.cif.gz | 4.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8218 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8218 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8218 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8218 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5k7pMC  8216C  8217C  8219C  8220C  8221C  8222C  8472C  5k7nC  5k7oC  5k7qC  5k7rC  5k7sC  5k7tC  5ty4C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8218.map.gz / 形式: CCP4 / 大きさ: 1.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8218.map.gz / 形式: CCP4 / 大きさ: 1.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Xylanase | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 0.75252 Å / Y: 0.7469 Å / Z: 0.79332 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
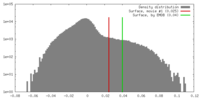
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 19 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Xylanase
| 全体 | 名称: Xylanase |
|---|---|
| 要素 |
|
-超分子 #1: Xylanase
| 超分子 | 名称: Xylanase / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Trichoderma reesei (菌類) Trichoderma reesei (菌類) |
| 分子量 | 理論値: 21.035 KDa |
-分子 #1: Endo-1,4-beta-xylanase 2
| 分子 | 名称: Endo-1,4-beta-xylanase 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: endo-1,4-beta-xylanase |
|---|---|
| 由来(天然) | 生物種:  Hypocrea jecorina (菌類) Hypocrea jecorina (菌類) |
| 分子量 | 理論値: 20.855467 KDa |
| 配列 | 文字列: QTIQPGTGYN NGYFYSYWND GHGGVTYTNG PGGQFSVNWS NSGNFVGGKG WQPGTKNKVI NFSGSYNPNG NSYLSVYGWS RNPLIEYYI VENFGTYNPS TGATKLGEVT SDGSVYDIYR TQRVNQPSII GTATFYQYWS VRRNHRSSGS VNTANHFNAW A QQGLTLGT ...文字列: QTIQPGTGYN NGYFYSYWND GHGGVTYTNG PGGQFSVNWS NSGNFVGGKG WQPGTKNKVI NFSGSYNPNG NSYLSVYGWS RNPLIEYYI VENFGTYNPS TGATKLGEVT SDGSVYDIYR TQRVNQPSII GTATFYQYWS VRRNHRSSGS VNTANHFNAW A QQGLTLGT MDYQIVAVEG YFSSGSASIT VS UniProtKB: Endo-1,4-beta-xylanase 2 |
-分子 #2: IODIDE ION
| 分子 | 名称: IODIDE ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: IOD |
|---|---|
| 分子量 | 理論値: 126.904 Da |
| Chemical component information |  ChemComp-IOD: |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 23 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線結晶学 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 緩衝液 | pH: 9 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) デジタル化 - サイズ - 横: 2048 pixel / デジタル化 - サイズ - 縦: 2048 pixel / 撮影したグリッド数: 1 / 実像数: 588 / 回折像の数: 588 / 平均露光時間: 4.1 sec. / 平均電子線量: 0.004 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / カメラ長: 1750 mm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)