[English] 日本語
 Yorodumi
Yorodumi- EMDB-8191: Cryo-EM structure of lactate dehydrogenase (LDH) in complex with ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-8191 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of lactate dehydrogenase (LDH) in complex with GSK2837808A | |||||||||
 Map data Map data | Lactate dehydrogenase (LDH) in complex with GSK2837808A | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | lactate dehydrogenase / small metabolic complex / small molecule inhibitor / OXIDOREDUCTASE | |||||||||
| Function / homology |  Function and homology information Function and homology informationPyruvate metabolism / Pyruvate metabolism / L-lactate dehydrogenase / oxidoreductase complex / pyruvate metabolic process / L-lactate dehydrogenase (NAD+) activity / lactate metabolic process / mitochondrial inner membrane / membrane raft / mitochondrion ...Pyruvate metabolism / Pyruvate metabolism / L-lactate dehydrogenase / oxidoreductase complex / pyruvate metabolic process / L-lactate dehydrogenase (NAD+) activity / lactate metabolic process / mitochondrial inner membrane / membrane raft / mitochondrion / identical protein binding / cytosol Similarity search - Function | |||||||||
| Biological species |  | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.8 Å | |||||||||
 Authors Authors | Merk A / Bartesaghi A | |||||||||
 Citation Citation |  Journal: Cell / Year: 2016 Journal: Cell / Year: 2016Title: Breaking Cryo-EM Resolution Barriers to Facilitate Drug Discovery. Authors: Alan Merk / Alberto Bartesaghi / Soojay Banerjee / Veronica Falconieri / Prashant Rao / Mindy I Davis / Rajan Pragani / Matthew B Boxer / Lesley A Earl / Jacqueline L S Milne / Sriram Subramaniam /  Abstract: Recent advances in single-particle cryoelecton microscopy (cryo-EM) are enabling generation of numerous near-atomic resolution structures for well-ordered protein complexes with sizes ≥ ∼200 kDa. ...Recent advances in single-particle cryoelecton microscopy (cryo-EM) are enabling generation of numerous near-atomic resolution structures for well-ordered protein complexes with sizes ≥ ∼200 kDa. Whether cryo-EM methods are equally useful for high-resolution structural analysis of smaller, dynamic protein complexes such as those involved in cellular metabolism remains an important question. Here, we present 3.8 Å resolution cryo-EM structures of the cancer target isocitrate dehydrogenase (93 kDa) and identify the nature of conformational changes induced by binding of the allosteric small-molecule inhibitor ML309. We also report 2.8-Å- and 1.8-Å-resolution structures of lactate dehydrogenase (145 kDa) and glutamate dehydrogenase (334 kDa), respectively. With these results, two perceived barriers in single-particle cryo-EM are overcome: (1) crossing 2 Å resolution and (2) obtaining structures of proteins with sizes < 100 kDa, demonstrating that cryo-EM can be used to investigate a broad spectrum of drug-target interactions and dynamic conformational states. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_8191.map.gz emd_8191.map.gz | 19.7 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-8191-v30.xml emd-8191-v30.xml emd-8191.xml emd-8191.xml | 16.6 KB 16.6 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_8191.png emd_8191.png | 169.7 KB | ||
| Filedesc metadata |  emd-8191.cif.gz emd-8191.cif.gz | 6.1 KB | ||
| Others |  emd_8191_additional.map.gz emd_8191_additional.map.gz | 19.9 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8191 http://ftp.pdbj.org/pub/emdb/structures/EMD-8191 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8191 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8191 | HTTPS FTP |
-Related structure data
| Related structure data |  5k0zMC  8192C  8193C  8194C  5k10C  5k11C  5k12C C: citing same article ( M: atomic model generated by this map |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_8191.map.gz / Format: CCP4 / Size: 22.2 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_8191.map.gz / Format: CCP4 / Size: 22.2 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Lactate dehydrogenase (LDH) in complex with GSK2837808A | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.495 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
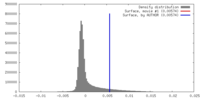
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
-Additional map: Map sharpened using a B-factor of -150
| File | emd_8191_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Map sharpened using a B-factor of -150 | ||||||||||||
| Projections & Slices |
| ||||||||||||
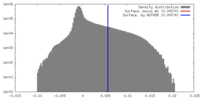
| Density Histograms |
- Sample components
Sample components
-Entire : Lactate dehydrogenase (LDH) in complex with GSK2837808A
| Entire | Name: Lactate dehydrogenase (LDH) in complex with GSK2837808A |
|---|---|
| Components |
|
-Supramolecule #1: Lactate dehydrogenase (LDH) in complex with GSK2837808A
| Supramolecule | Name: Lactate dehydrogenase (LDH) in complex with GSK2837808A type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1 |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 145 KDa |
-Macromolecule #1: L-lactate dehydrogenase B chain
| Macromolecule | Name: L-lactate dehydrogenase B chain / type: protein_or_peptide / ID: 1 / Number of copies: 4 / Enantiomer: LEVO / EC number: L-lactate dehydrogenase |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 36.115656 KDa |
| Recombinant expression | Organism: Bacteria (eubacteria) |
| Sequence | String: ATLKEKLITP VAAGSTVPSN KITVVGVGQV GMACAISILG KGLCDELALV DVLEDKLKGE MMDLQHGSLF LQTHKIVADK DYAVTANSK IVVVTAGVRQ QEGESRLNLV QRNVNVFKFI IPQIVKYSPN CTILVVSNPV DILTYVTWKL SGLPKHRVIG S GCNLDTAR ...String: ATLKEKLITP VAAGSTVPSN KITVVGVGQV GMACAISILG KGLCDELALV DVLEDKLKGE MMDLQHGSLF LQTHKIVADK DYAVTANSK IVVVTAGVRQ QEGESRLNLV QRNVNVFKFI IPQIVKYSPN CTILVVSNPV DILTYVTWKL SGLPKHRVIG S GCNLDTAR FRYLMAERLG IHPTSCHGWI LGEHGDSSVA VWSGVNVAGV SLQELNPAMG TDKDSENWKE VHKQVVESAY EV IRLKGYT NWAIGLSVAE LCETMLKNLY RVHSVSTLVK GTYGIENDVF LSLPCVLSAS GLTSVINQKL KDDEVAQLKK SAD TLWSIQ KDLKD UniProtKB: L-lactate dehydrogenase B chain |
-Macromolecule #2: water
| Macromolecule | Name: water / type: ligand / ID: 2 / Number of copies: 41 / Formula: HOH |
|---|---|
| Molecular weight | Theoretical: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 1.5 mg/mL |
|---|---|
| Buffer | pH: 7.4 / Component - Name: PBS / Details: Phosphate-buffered saline |
| Grid | Model: Quantifoil R1.2/1.3 / Material: COPPER / Mesh: 300 / Support film - Material: CARBON / Support film - topology: HOLEY / Pretreatment - Type: PLASMA CLEANING |
| Vitrification | Cryogen name: ETHANE / Instrument: LEICA EM GP / Details: Plunged into liquid ethane (LEICE EM GP). |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Temperature | Min: 79.6 K / Max: 79.8 K |
| Specialist optics | Energy filter - Name: GIF Quantum / Energy filter - Lower energy threshold: 0 eV / Energy filter - Upper energy threshold: 20 eV |
| Image recording | Film or detector model: GATAN K2 QUANTUM (4k x 4k) / Detector mode: SUPER-RESOLUTION / Digitization - Frames/image: 0-29 / Number real images: 1707 / Average exposure time: 0.2 sec. / Average electron dose: 60.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Calibrated magnification: 101000 / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.2 µm / Nominal defocus min: 0.8 µm / Nominal magnification: 270000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)