+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8013 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Head region of the yeast spliceosomal U4/U6.U5 tri-snRNP | |||||||||
 マップデータ マップデータ | Head Region of yeast spliceosomal U4/U6.U5 tri-snRNP. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Transcription / pre-mRNA splicing / snRNP / GTPase / spliceosome | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maturation of 5S rRNA / spliceosome conformational change to release U4 (or U4atac) and U1 (or U11) / U4/U6 snRNP / 7-methylguanosine cap hypermethylation / pICln-Sm protein complex / snRNP binding / small nuclear ribonucleoprotein complex / splicing factor binding / SMN-Sm protein complex / spliceosomal tri-snRNP complex ...maturation of 5S rRNA / spliceosome conformational change to release U4 (or U4atac) and U1 (or U11) / U4/U6 snRNP / 7-methylguanosine cap hypermethylation / pICln-Sm protein complex / snRNP binding / small nuclear ribonucleoprotein complex / splicing factor binding / SMN-Sm protein complex / spliceosomal tri-snRNP complex / mRNA cis splicing, via spliceosome / commitment complex / U2-type prespliceosome assembly / U2 snRNP / U1 snRNP / U4 snRNP / U2-type prespliceosome / poly(U) RNA binding / precatalytic spliceosome / generation of catalytic spliceosome for second transesterification step / mRNA 5'-splice site recognition / spliceosomal complex assembly / mRNA 3'-splice site recognition / spliceosomal tri-snRNP complex assembly / Prp19 complex / U5 snRNA binding / U5 snRNP / U2 snRNA binding / U6 snRNA binding / pre-mRNA intronic binding / spliceosomal snRNP assembly / U1 snRNA binding / U4/U6 x U5 tri-snRNP complex / catalytic step 2 spliceosome / spliceosomal complex / mRNA splicing, via spliceosome / metallopeptidase activity / nucleic acid binding / RNA helicase activity / RNA helicase / mRNA binding / ATP hydrolysis activity / RNA binding / ATP binding / identical protein binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
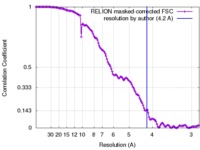
| 手法 | 単粒子再構成法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Nguyen THD / Galej WP | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2015 ジャーナル: Nature / 年: 2015タイトル: The architecture of the spliceosomal U4/U6.U5 tri-snRNP. 著者: Thi Hoang Duong Nguyen / Wojciech P Galej / Xiao-chen Bai / Christos G Savva / Andrew J Newman / Sjors H W Scheres / Kiyoshi Nagai /  要旨: U4/U6.U5 tri-snRNP is a 1.5-megadalton pre-assembled spliceosomal complex comprising U5 small nuclear RNA (snRNA), extensively base-paired U4/U6 snRNAs and more than 30 proteins, including the key ...U4/U6.U5 tri-snRNP is a 1.5-megadalton pre-assembled spliceosomal complex comprising U5 small nuclear RNA (snRNA), extensively base-paired U4/U6 snRNAs and more than 30 proteins, including the key components Prp8, Brr2 and Snu114. The tri-snRNP combines with a precursor messenger RNA substrate bound to U1 and U2 small nuclear ribonucleoprotein particles (snRNPs), and transforms into a catalytically active spliceosome after extensive compositional and conformational changes triggered by unwinding of the U4 and U6 (U4/U6) snRNAs. Here we use cryo-electron microscopy single-particle reconstruction of Saccharomyces cerevisiae tri-snRNP at 5.9 Å resolution to reveal the essentially complete organization of its RNA and protein components. The single-stranded region of U4 snRNA between its 3' stem-loop and the U4/U6 snRNA stem I is loaded into the Brr2 helicase active site ready for unwinding. Snu114 and the amino-terminal domain of Prp8 position U5 snRNA to insert its loop I, which aligns the exons for splicing, into the Prp8 active site cavity. The structure provides crucial insights into the activation process and the active site of the spliceosome. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
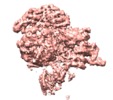
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8013.map.gz emd_8013.map.gz | 195.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8013-v30.xml emd-8013-v30.xml emd-8013.xml emd-8013.xml | 28.2 KB 28.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8013_fsc.xml emd_8013_fsc.xml | 13.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8013.png emd_8013.png | 179.8 KB | ||
| Filedesc metadata |  emd-8013.cif.gz emd-8013.cif.gz | 8.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8013 http://ftp.pdbj.org/pub/emdb/structures/EMD-8013 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8013 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8013 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5gaoMC  8006C  8007C  8008C  8009C  8010C  8011C  8012C  8014C  5gamC  5ganC  5gapC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8013.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8013.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Head Region of yeast spliceosomal U4/U6.U5 tri-snRNP. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.43 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Head region of the yeast spliceosomal U4/U6.U5 tri-snRNP
+超分子 #1: Head region of the yeast spliceosomal U4/U6.U5 tri-snRNP
+分子 #1: Small nuclear ribonucleoprotein-associated protein B
+分子 #2: Small nuclear ribonucleoprotein Sm D1
+分子 #3: Small nuclear ribonucleoprotein Sm D2
+分子 #4: Small nuclear ribonucleoprotein Sm D3
+分子 #5: Small nuclear ribonucleoprotein E
+分子 #6: Small nuclear ribonucleoprotein F
+分子 #7: Small nuclear ribonucleoprotein G
+分子 #8: Snu66
+分子 #9: Pre-mRNA-splicing helicase BRR2
+分子 #11: Pre-mRNA-splicing factor 8
+分子 #10: Saccharomyces cerevisiae strain UOA_M2 chromosome 5 sequence
-実験情報
-構造解析
 解析 解析 | 単粒子再構成法 |
|---|---|
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 / 構成要素 - 濃度: 1.0 mM / 構成要素 - 名称: DTT |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 6 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 70 sec. / 前処理 - 雰囲気: OTHER 詳細: Grids are made of holey carbon, carbon-coated and glow discharged in N-amylamine. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-20 / 実像数: 2477 / 平均露光時間: 16.0 sec. / 平均電子線量: 38.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 35714 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: RECIPROCAL / プロトコル: OTHER | ||||||
| 得られたモデル |  PDB-5gao: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)