+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7onb | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of the U2 5' module of the A3'-SSA complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | SPLICING / Spliceosome / spliceostatin A / splicing inhibitor | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報U11/U12 snRNP / B-WICH complex / splicing factor binding / U12-type spliceosomal complex / RNA splicing, via transesterification reactions / U2-type precatalytic spliceosome / U2-type spliceosomal complex / U2-type prespliceosome assembly / positive regulation of mRNA splicing, via spliceosome / U2 snRNP ...U11/U12 snRNP / B-WICH complex / splicing factor binding / U12-type spliceosomal complex / RNA splicing, via transesterification reactions / U2-type precatalytic spliceosome / U2-type spliceosomal complex / U2-type prespliceosome assembly / positive regulation of mRNA splicing, via spliceosome / U2 snRNP / SAGA complex / positive regulation of transcription by RNA polymerase III / U2-type prespliceosome / precatalytic spliceosome / positive regulation of transcription by RNA polymerase I / spliceosomal complex assembly / mRNA Splicing - Minor Pathway / regulation of RNA splicing / mRNA 3'-splice site recognition / U2 snRNA binding / regulation of DNA repair / catalytic step 2 spliceosome / mRNA Splicing - Major Pathway / RNA splicing / negative regulation of protein catabolic process / stem cell differentiation / spliceosomal complex / B-WICH complex positively regulates rRNA expression / positive regulation of neuron projection development / nuclear matrix / mRNA splicing, via spliceosome / mRNA processing / chromatin remodeling / nuclear speck / mRNA binding / positive regulation of DNA-templated transcription / protein-containing complex binding / nucleolus / positive regulation of transcription by RNA polymerase II / DNA binding / RNA binding / zinc ion binding / nucleoplasm / nucleus 類似検索 - 分子機能 | ||||||
| 生物種 |  unidentified adenovirus (ウイルス) unidentified adenovirus (ウイルス) Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | ||||||
 データ登録者 データ登録者 | Cretu, C. / Pena, V. | ||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structural basis of intron selection by U2 snRNP in the presence of covalent inhibitors. 著者: Constantin Cretu / Patricia Gee / Xiang Liu / Anant Agrawal / Tuong-Vi Nguyen / Arun K Ghosh / Andrew Cook / Melissa Jurica / Nicholas A Larsen / Vladimir Pena /    要旨: Intron selection during the formation of prespliceosomes is a critical event in pre-mRNA splicing. Chemical modulation of intron selection has emerged as a route for cancer therapy. Splicing ...Intron selection during the formation of prespliceosomes is a critical event in pre-mRNA splicing. Chemical modulation of intron selection has emerged as a route for cancer therapy. Splicing modulators alter the splicing patterns in cells by binding to the U2 snRNP (small nuclear ribonucleoprotein)-a complex chaperoning the selection of branch and 3' splice sites. Here we report crystal structures of the SF3B module of the U2 snRNP in complex with spliceostatin and sudemycin FR901464 analogs, and the cryo-electron microscopy structure of a cross-exon prespliceosome-like complex arrested with spliceostatin A. The structures reveal how modulators inactivate the branch site in a sequence-dependent manner and stall an E-to-A prespliceosome intermediate by covalent coupling to a nucleophilic zinc finger belonging to the SF3B subunit PHF5A. These findings support a mechanism of intron recognition by the U2 snRNP as a toehold-mediated strand invasion and advance an unanticipated drug targeting concept. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7onb.cif.gz 7onb.cif.gz | 533.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7onb.ent.gz pdb7onb.ent.gz | 390.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7onb.json.gz 7onb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7onb_validation.pdf.gz 7onb_validation.pdf.gz | 823.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7onb_full_validation.pdf.gz 7onb_full_validation.pdf.gz | 841.2 KB | 表示 | |
| XML形式データ |  7onb_validation.xml.gz 7onb_validation.xml.gz | 78 KB | 表示 | |
| CIF形式データ |  7onb_validation.cif.gz 7onb_validation.cif.gz | 113.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/on/7onb https://data.pdbj.org/pub/pdb/validation_reports/on/7onb ftp://data.pdbj.org/pub/pdb/validation_reports/on/7onb ftp://data.pdbj.org/pub/pdb/validation_reports/on/7onb | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Splicing factor 3B subunit ... , 5種, 5分子 ABCIK
| #1: タンパク質 | 分子量: 135718.844 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q15393 Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q15393 |
|---|---|
| #2: タンパク質 | 分子量: 10149.369 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q9BWJ5 Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q9BWJ5 |
| #3: タンパク質 | 分子量: 146024.938 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: O75533 Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: O75533 |
| #8: タンパク質 | 分子量: 100377.812 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q13435 Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q13435 |
| #11: タンパク質 | 分子量: 44436.570 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q15427 Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q15427 |
-RNA鎖 , 2種, 2分子 GH
| #6: RNA鎖 | 分子量: 73393.234 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  unidentified adenovirus (ウイルス) unidentified adenovirus (ウイルス)発現宿主: synthetic construct (人工物) |
|---|---|
| #7: RNA鎖 | 分子量: 60186.445 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: GenBank: 36516 Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: GenBank: 36516 |
-Splicing factor 3A subunit ... , 2種, 2分子 MN
| #9: タンパク質 | 分子量: 49327.355 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q15428 Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q15428 |
|---|---|
| #10: タンパク質 | 分子量: 58934.844 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q12874 Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q12874 |
-タンパク質 / タンパク質・ペプチド , 2種, 2分子 DF
| #4: タンパク質 | 分子量: 12427.524 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q7RTV0 Homo sapiens (ヒト) / 細胞株: HeLa S3 / 参照: UniProt: Q7RTV0 |
|---|---|
| #5: タンパク質・ペプチド | 分子量: 1999.216 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HeLa S3 Homo sapiens (ヒト) / 細胞株: HeLa S3 |
-非ポリマー , 2種, 6分子 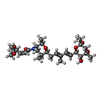


| #12: 化合物 | ChemComp-SJT / |
|---|---|
| #13: 化合物 | ChemComp-ZN / |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | ||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||
| 由来(組換発現) | 生物種: synthetic construct (人工物) | ||||||||||||||||||||||||
| 緩衝液 | pH: 7.9 | ||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||
| 試料 | 濃度: 0.24 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのタイプ: UltrAuFoil R1.2/1.3 | ||||||||||||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277.15 K 詳細: The A3'-SSA complex was frozen in vitreous ice by applying ~2.2 uL crosslinked sample to both sides of a glow-discharged UltrAufoil R1.2/1.3 gold grid. The excess sample was blotted away for ...詳細: The A3'-SSA complex was frozen in vitreous ice by applying ~2.2 uL crosslinked sample to both sides of a glow-discharged UltrAufoil R1.2/1.3 gold grid. The excess sample was blotted away for 2s using a blot force of 7 and the grid was snap frozen in liquid ethane cooled by liquid nitrogen. |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 81000 X / 倍率(補正後): 81000 X / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 1000 nm / Cs: 2.7 mm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 2 sec. / 電子線照射量: 41.41 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 10494 |
| 電子光学装置 | エネルギーフィルター名称: GIF Quantum LS / エネルギーフィルタースリット幅: 20 eV |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1052001 | ||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 78262 / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: OTHER / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
| ||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 交差検証法: NONE 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 127.46 Å2 | ||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj





































