+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6487 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron microscopy structure of RAG SEC (C2 symmetry) | |||||||||
 マップデータ マップデータ | Reconstruction of RAG SEC with C2 symmetry | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RAG1 / RAG2 / V(D)J recombination / Signal end complex / Antigen receptor recombination / T and B cell development | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報somatic diversification of immune receptors via germline recombination within a single locus / hematopoietic or lymphoid organ development / protein binding / protein-DNA complex assembly / DNA recombinase complex / endodeoxyribonuclease complex / immunoglobulin V(D)J recombination / lymphocyte differentiation / V(D)J recombination / phosphatidylinositol-3,4-bisphosphate binding ...somatic diversification of immune receptors via germline recombination within a single locus / hematopoietic or lymphoid organ development / protein binding / protein-DNA complex assembly / DNA recombinase complex / endodeoxyribonuclease complex / immunoglobulin V(D)J recombination / lymphocyte differentiation / V(D)J recombination / phosphatidylinositol-3,4-bisphosphate binding / phosphatidylinositol-3,5-bisphosphate binding / phosphatidylinositol-3,4,5-trisphosphate binding / T cell differentiation / phosphatidylinositol-4,5-bisphosphate binding / methylated histone binding / B cell differentiation / phosphatidylinositol binding / thymus development / RING-type E3 ubiquitin transferase / ubiquitin-protein transferase activity / ubiquitin protein ligase activity / chromatin organization / histone binding / T cell differentiation in thymus / endonuclease activity / DNA recombination / sequence-specific DNA binding / adaptive immune response / 加水分解酵素; エステル加水分解酵素 / chromatin binding / magnesium ion binding / protein homodimerization activity / DNA binding / zinc ion binding / nucleus / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Ru H / Chambers MG / Fu T / Tong AB / Liao M / Wu H | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2015 ジャーナル: Cell / 年: 2015タイトル: Molecular Mechanism of V(D)J Recombination from Synaptic RAG1-RAG2 Complex Structures. 著者: Heng Ru / Melissa G Chambers / Tian-Min Fu / Alexander B Tong / Maofu Liao / Hao Wu /  要旨: Diverse repertoires of antigen-receptor genes that result from combinatorial splicing of coding segments by V(D)J recombination are hallmarks of vertebrate immunity. The (RAG1-RAG2)2 recombinase (RAG) ...Diverse repertoires of antigen-receptor genes that result from combinatorial splicing of coding segments by V(D)J recombination are hallmarks of vertebrate immunity. The (RAG1-RAG2)2 recombinase (RAG) recognizes recombination signal sequences (RSSs) containing a heptamer, a spacer of 12 or 23 base pairs, and a nonamer (12-RSS or 23-RSS) and introduces precise breaks at RSS-coding segment junctions. RAG forms synaptic complexes only with one 12-RSS and one 23-RSS, a dogma known as the 12/23 rule that governs the recombination fidelity. We report cryo-electron microscopy structures of synaptic RAG complexes at up to 3.4 Å resolution, which reveal a closed conformation with base flipping and base-specific recognition of RSSs. Distortion at RSS-coding segment junctions and base flipping in coding segments uncover the two-metal-ion catalytic mechanism. Induced asymmetry involving tilting of the nonamer-binding domain dimer of RAG1 upon binding of HMGB1-bent 12-RSS or 23-RSS underlies the molecular mechanism for the 12/23 rule. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6487.map.gz emd_6487.map.gz | 25.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6487-v30.xml emd-6487-v30.xml emd-6487.xml emd-6487.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6487_fsc.xml emd_6487_fsc.xml | 6.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6487.png emd_6487.png | 570.7 KB | ||
| マスクデータ |  emd_6487_msk_1.map emd_6487_msk_1.map | 27 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6487 http://ftp.pdbj.org/pub/emdb/structures/EMD-6487 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6487 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6487 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6487_validation.pdf.gz emd_6487_validation.pdf.gz | 438.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6487_full_validation.pdf.gz emd_6487_full_validation.pdf.gz | 438.1 KB | 表示 | |
| XML形式データ |  emd_6487_validation.xml.gz emd_6487_validation.xml.gz | 9.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6487 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6487 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6487 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6487 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3jbxMC  6488C  6489C  6490C  6491C  3jbwC  3jbyC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10049 (タイトル: Cryo-EM Structures of Synaptic RAG1-RAG2 Complex EMPIAR-10049 (タイトル: Cryo-EM Structures of Synaptic RAG1-RAG2 ComplexData size: 65.9 Data #1: RAG SEC particle stack [picked particles - multiframe - processed] Data #2: Summed micrographs from drift-corrected multi-frame microsgraphs of RAG SEC (1st data set) [micrographs - single frame] Data #3: Summed micrographs from drift-corrected multi-frame microsgraphs of RAG SEC (2nd data set) [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6487.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6487.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of RAG SEC with C2 symmetry | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.23 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
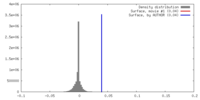
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-セグメンテーションマップ: This mask represent the whole active site region.
| 注釈 | This mask represent the whole active site region. | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_6487_msk_1.map emd_6487_msk_1.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
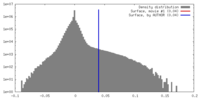
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Signal End Complex of RAG1-RAG2
| 全体 | 名称: Signal End Complex of RAG1-RAG2 |
|---|---|
| 要素 |
|
-超分子 #1000: Signal End Complex of RAG1-RAG2
| 超分子 | 名称: Signal End Complex of RAG1-RAG2 / タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse. 集合状態: Dimer of RAG1-RAG2 binds to 12-RSS and 23-RSS products with HMGB1 Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 380 KDa / 理論値: 380 KDa / 手法: Size exclusion chromatography |
-分子 #1: Recombination Activating Gene 1 and 2
| 分子 | 名称: Recombination Activating Gene 1 and 2 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: RAG1 and RAG2 / コピー数: 2 / 集合状態: Dimer of RAG1-RAG2 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 380 KDa / 理論値: 380 KDa |
| 組換発現 | 生物種:  組換細胞: Sf9 / 組換プラスミド: pFastBac1 |
| 配列 | UniProtKB: V(D)J recombination-activating protein 1 GO: DNA binding, endonuclease activity, ubiquitin-protein transferase activity, protein binding, zinc ion binding, DNA binding, chromatin binding, protein binding, phosphatidylinositol-4,5- ...GO: DNA binding, endonuclease activity, ubiquitin-protein transferase activity, protein binding, zinc ion binding, DNA binding, chromatin binding, protein binding, phosphatidylinositol-4,5-bisphosphate binding, phosphatidylinositol-3,4,5-trisphosphate binding InterPro: V(D)J recombination-activating protein 1, RAG nonamer-binding domain, Zinc finger, C3HC4 RING-type, Zinc finger, RING-type, Zinc finger, RING/FYVE/PHD-type, Zinc finger, RING-type, ...InterPro: V(D)J recombination-activating protein 1, RAG nonamer-binding domain, Zinc finger, C3HC4 RING-type, Zinc finger, RING-type, Zinc finger, RING/FYVE/PHD-type, Zinc finger, RING-type, conserved site, Galactose oxidase/kelch, beta-propeller, Kelch-type beta propeller, V-D-J recombination activating protein 2, Recombination activating protein 2, PHD domain, Zinc finger, FYVE/PHD-type |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 150 mM NaCl, 20 mM HEPES, 5 mM MgCl2, 1 mM TCEP |
| グリッド | 詳細: 400 mesh Quantifoil holey carbon grid, glow discharged |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 120 K / 装置: GATAN CRYOPLUNGE 3 / 手法: Blot for 2.5 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 80 K / 最高: 105 K / 平均: 100 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 150,000 times magnification. |
| 日付 | 2015年3月9日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / 実像数: 650 / 平均電子線量: 41 e/Å2 詳細: Every image is the average of 30 frames recorded by the direct electron detector. ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 40607 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 31000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)