+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5693 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the SecY protein translocation channel in action | |||||||||
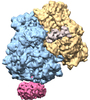
 マップデータ マップデータ | ribosome-nascent chain-SecYEG complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ribosome-channel complex / active SecYEG channel / nascent chain / E. coli 70S ribosome | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / cell envelope Sec protein transport complex / protein transport by the Sec complex / intracellular protein transmembrane transport / protein-transporting ATPase activity / SRP-dependent cotranslational protein targeting to membrane, translocation / signal sequence binding / protein secretion / protein transmembrane transporter activity / negative regulation of translational initiation ...: / cell envelope Sec protein transport complex / protein transport by the Sec complex / intracellular protein transmembrane transport / protein-transporting ATPase activity / SRP-dependent cotranslational protein targeting to membrane, translocation / signal sequence binding / protein secretion / protein transmembrane transporter activity / negative regulation of translational initiation / intracellular protein transport / ribosomal large subunit assembly / large ribosomal subunit rRNA binding / cytosolic large ribosomal subunit / cytoplasmic translation / tRNA binding / rRNA binding / structural constituent of ribosome / translation / membrane / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 10.1 Å | |||||||||
 データ登録者 データ登録者 | Park E / Menetret JF / Gumbart JC / Ludtke SJ / Li W / Whynot A / Rapoport TA / Akey CW | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2014 ジャーナル: Nature / 年: 2014タイトル: Structure of the SecY channel during initiation of protein translocation. 著者: Eunyong Park / Jean-François Ménétret / James C Gumbart / Steven J Ludtke / Weikai Li / Andrew Whynot / Tom A Rapoport / Christopher W Akey /  要旨: Many secretory proteins are targeted by signal sequences to a protein-conducting channel, formed by prokaryotic SecY or eukaryotic Sec61 complexes, and are translocated across the membrane during ...Many secretory proteins are targeted by signal sequences to a protein-conducting channel, formed by prokaryotic SecY or eukaryotic Sec61 complexes, and are translocated across the membrane during their synthesis. Crystal structures of the inactive channel show that the SecY subunit of the heterotrimeric complex consists of two halves that form an hourglass-shaped pore with a constriction in the middle of the membrane and a lateral gate that faces the lipid phase. The closed channel has an empty cytoplasmic funnel and an extracellular funnel that is filled with a small helical domain, called the plug. During initiation of translocation, a ribosome-nascent chain complex binds to the SecY (or Sec61) complex, resulting in insertion of the nascent chain. However, the mechanism of channel opening during translocation is unclear. Here we have addressed this question by determining structures of inactive and active ribosome-channel complexes with cryo-electron microscopy. Non-translating ribosome-SecY channel complexes derived from Methanocaldococcus jannaschii or Escherichia coli show the channel in its closed state, and indicate that ribosome binding per se causes only minor changes. The structure of an active E. coli ribosome-channel complex demonstrates that the nascent chain opens the channel, causing mostly rigid body movements of the amino- and carboxy-terminal halves of SecY. In this early translocation intermediate, the polypeptide inserts as a loop into the SecY channel with the hydrophobic signal sequence intercalated into the open lateral gate. The nascent chain also forms a loop on the cytoplasmic surface of SecY rather than entering the channel directly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5693.map.gz emd_5693.map.gz | 3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5693-v30.xml emd-5693-v30.xml emd-5693.xml emd-5693.xml | 35.5 KB 35.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5693_1.jpg emd_5693_1.jpg | 182.5 KB | ||
| マスクデータ |  emd_5693_msk_1.map emd_5693_msk_1.map emd_5693_msk_2.map emd_5693_msk_2.map emd_5693_msk_3.map emd_5693_msk_3.map emd_5693_msk_4.map emd_5693_msk_4.map emd_5693_msk_5.map emd_5693_msk_5.map emd_5693_msk_6.map emd_5693_msk_6.map emd_5693_msk_7.map emd_5693_msk_7.map emd_5693_msk_8.map emd_5693_msk_8.map emd_5693_msk_9.map emd_5693_msk_9.map | 27 MB 601 KB 27 MB 263.3 KB 27 MB 27 MB 27 MB 27 MB 4.3 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5693 http://ftp.pdbj.org/pub/emdb/structures/EMD-5693 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5693 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5693 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5693.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5693.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ribosome-nascent chain-SecYEG complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.12 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-セグメンテーションマップ: segmented large ribosomal subunit
| 注釈 | segmented large ribosomal subunit | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_5693_msk_1.map emd_5693_msk_1.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-セグメンテーションマップ: segmented A and P-site tRNAs
| 注釈 | segmented A and P-site tRNAs | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_5693_msk_2.map emd_5693_msk_2.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-セグメンテーションマップ: micelle map from channel density
| 注釈 | micelle map from channel density | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_5693_msk_3.map emd_5693_msk_3.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-セグメンテーションマップ: zoned SecYEG channel
| 注釈 | zoned SecYEG channel | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_5693_msk_4.map emd_5693_msk_4.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-セグメンテーションマップ: full channel density with micelle and nascent chain
| 注釈 | full channel density with micelle and nascent chain | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_5693_msk_5.map emd_5693_msk_5.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-セグメンテーションマップ: full nascent chain density, excluding fragments in the tunnel
| 注釈 | full nascent chain density, excluding fragments in the tunnel | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_5693_msk_6.map emd_5693_msk_6.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-セグメンテーションマップ: nascent chain density with signal sequence helix in the channel
| 注釈 | nascent chain density with signal sequence helix in the channel | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_5693_msk_7.map emd_5693_msk_7.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-セグメンテーションマップ: segmented S1 density, excluding flexible fragments
| 注釈 | segmented S1 density, excluding flexible fragments | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_5693_msk_8.map emd_5693_msk_8.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-セグメンテーションマップ: segmented small subunit
| 注釈 | segmented small subunit | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_5693_msk_9.map emd_5693_msk_9.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ribosome-nascent chain-SecYEG complex
| 全体 | 名称: ribosome-nascent chain-SecYEG complex |
|---|---|
| 要素 |
|
-超分子 #1000: ribosome-nascent chain-SecYEG complex
| 超分子 | 名称: ribosome-nascent chain-SecYEG complex / タイプ: sample / ID: 1000 詳細: Sample was prepared by crosslinking the nascent chain within stalled membrane associated ribosomes in bacteria, then purified for cryo-EM. 集合状態: monomer / Number unique components: 5 |
|---|---|
| 分子量 | 理論値: 2.5 MDa |
-超分子 #1: membrane-bound 70S ribosome
| 超分子 | 名称: membrane-bound 70S ribosome / タイプ: complex / ID: 1 / 組換発現: No / データベース: NCBI / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 2.5 MDa |
-分子 #1: preprotein translocase
| 分子 | 名称: preprotein translocase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: SecYEG channel / 詳細: SecY: P0AGA2, SecE: P0AG96, SecG: P0AG99 / コピー数: 1 / 集合状態: heterotrimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 73.4 KDa |
| 組換発現 | 生物種:  |
-分子 #2: S1P
| 分子 | 名称: S1P / タイプ: protein_or_peptide / ID: 2 Name.synonym: endogenous E. coli small ribosomal subunit protein コピー数: 1 / 集合状態: monomer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 61 KDa |
-分子 #3: transfer RNA
| 分子 | 名称: transfer RNA / タイプ: rna / ID: 3 / Name.synonym: tRNA 詳細: Both A- and P-site tRNAs present in purified ribosome-nascent chain-SecYEG complex, stalled with SecM sequence 分類: TRANSFER / Structure: DOUBLE HELIX / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 詳細: 50 mM Tris-acetate, 10 mM Mg(OAc)2, 80 mM KOAc, 0.06% DDM |
| グリッド | 詳細: 400 mesh Quantifoil holey grids with 2/1 or 1.2/1.2 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 77 K / 装置: FEI VITROBOT MARK III 詳細: A homemade freezing device with N2 gas driven plunger was also used to prepare grids in a cabinet to maintain humidity. 手法: Blot 1-2 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 最低: 94 K |
| アライメント法 | Legacy - 非点収差: Carbon grain was imaged at ~175000 times magnification and Thon rings were optimized manually, as visualized on a Fourier transform of ccd images. |
| 詳細 | Low dose imaging: automated single particle data collection program from TVIPS was used. |
| 日付 | 2012年2月10日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GENERIC TVIPS (4k x 4k) 実像数: 4900 / 平均電子線量: 20 e/Å2 詳細: CCD 4k x 4k image frames: best 4900 from ~5900 images used for data processing. ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 160 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 42000 |
| 試料ステージ | 試料ホルダー: Oxford cold holder / 試料ホルダーモデル: OTHER |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | ~450000 particles were selected with e2boxer, used for per ccd frame CTF correction, and then subjected to unsupervised classification to remove aggregates to give 167000 particles. These particles were subjected to supervised classification in 2 steps to give a data set enriched in channels. Particles with the best signal to noise ratio were identified using the FRC comparator from the refinemulti run and classified with e2ligandclassify.py. |
|---|---|
| CTF補正 | 詳細: per micrograph |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 10.1 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2 詳細: The structure was solved twice: first with a model starting from a 25-Angstrom filtered E. coli ribosome map generated in house, and then a second time using a filtered ribosome model (EMD- ...詳細: The structure was solved twice: first with a model starting from a 25-Angstrom filtered E. coli ribosome map generated in house, and then a second time using a filtered ribosome model (EMD-5036). In each case, after convergence, maps from two EMAN2 refinements with different parameters were averaged after alignment in Chimera. Four maps in total were averaged to reduce the noise. 使用した粒子像数: 53000 |
| 最終 2次元分類 | クラス数: 5300 |
-原子モデル構築 1
| 初期モデル | PDB ID:  2i2p |
|---|---|
| ソフトウェア | 名称: Chimera, MDFF |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-3j46: |
-原子モデル構築 2
| 初期モデル | PDB ID:  3j01 |
|---|---|
| ソフトウェア | 名称: Chimera, MDFF |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-3j46: |
-原子モデル構築 3
| 初期モデル | PDB ID:  3i8g Chain - #0 - Chain ID: B / Chain - #1 - Chain ID: C |
|---|---|
| ソフトウェア | 名称: Chimera, MDFF |
| 詳細 | A- and P-site tRNAs from T. thermophilus ribosome structure. mRNA from this structure also used for some modeling steps. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-3j46: |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)