+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4423 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Helical part of the influenza A virus ribonucleoprotein. Conformation 3. | |||||||||
 マップデータ マップデータ | Inuenza A nucleoprotein docked into 3D helical structure of the wild type ribonucleoprotein complex obtained using cryoEM. Conformation 3. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Influenza A virus / Ribonucleoprotein / RNA binding protein / Viral protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative stranded viral RNA replication / helical viral capsid / viral penetration into host nucleus / host cell / viral nucleocapsid / ribonucleoprotein complex / symbiont entry into host cell / host cell nucleus / structural molecule activity / RNA binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Influenza A virus (A/Wilson-Smith/1933(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Wilson-Smith/1933(H1N1)) (A型インフルエンザウイルス) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 10.0 Å | |||||||||
 データ登録者 データ登録者 | Coloma R / Arranz R | |||||||||
| 資金援助 |  スペイン, 2件 スペイン, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2020 ジャーナル: Nat Microbiol / 年: 2020タイトル: Structural insights into influenza A virus ribonucleoproteins reveal a processive helical track as transcription mechanism. 著者: Rocío Coloma / Rocío Arranz / José M de la Rosa-Trevín / Carlos O S Sorzano / Sandie Munier / Diego Carlero / Nadia Naffakh / Juan Ortín / Jaime Martín-Benito /    要旨: The influenza virus genome consists of eight viral ribonucleoproteins (vRNPs), each consisting of a copy of the polymerase, one of the genomic RNA segments and multiple copies of the nucleoprotein ...The influenza virus genome consists of eight viral ribonucleoproteins (vRNPs), each consisting of a copy of the polymerase, one of the genomic RNA segments and multiple copies of the nucleoprotein arranged in a double helical conformation. vRNPs are macromolecular machines responsible for messenger RNA synthesis and genome replication, that is, the formation of progeny vRNPs. Here, we describe the structural basis of the transcription process. The mechanism, which we call the 'processive helical track', is based on the extreme flexibility of the helical part of the vRNP that permits a sliding movement between both antiparallel nucleoprotein-RNA strands, thereby allowing the polymerase to move over the genome while bound to both RNA ends. Accordingly, we demonstrate that blocking this movement leads to inhibition of vRNP transcriptional activity. This mechanism also reveals a critical role of the nucleoprotein in maintaining the double helical structure throughout the copying process to make the RNA template accessible to the polymerase. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4423.map.gz emd_4423.map.gz | 6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4423-v30.xml emd-4423-v30.xml emd-4423.xml emd-4423.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_4423.png emd_4423.png | 150.5 KB | ||
| Filedesc metadata |  emd-4423.cif.gz emd-4423.cif.gz | 6 KB | ||
| その他 |  emd_4423_additional.map.gz emd_4423_additional.map.gz | 2.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4423 http://ftp.pdbj.org/pub/emdb/structures/EMD-4423 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4423 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4423 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4423_validation.pdf.gz emd_4423_validation.pdf.gz | 465.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4423_full_validation.pdf.gz emd_4423_full_validation.pdf.gz | 465.1 KB | 表示 | |
| XML形式データ |  emd_4423_validation.xml.gz emd_4423_validation.xml.gz | 5.3 KB | 表示 | |
| CIF形式データ |  emd_4423_validation.cif.gz emd_4423_validation.cif.gz | 6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4423 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4423 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4423 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4423 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4423.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4423.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Inuenza A nucleoprotein docked into 3D helical structure of the wild type ribonucleoprotein complex obtained using cryoEM. Conformation 3. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.26 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Local resolution map calculated using MonoRes software
| ファイル | emd_4423_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
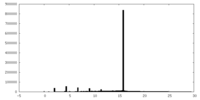
| 注釈 | Local resolution map calculated using MonoRes software | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
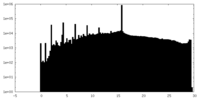
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Influenza A virus ribonucleoprotein
| 全体 | 名称: Influenza A virus ribonucleoprotein |
|---|---|
| 要素 |
|
-超分子 #1: Influenza A virus ribonucleoprotein
| 超分子 | 名称: Influenza A virus ribonucleoprotein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/Wilson-Smith/1933(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Wilson-Smith/1933(H1N1)) (A型インフルエンザウイルス) |
-分子 #1: Nucleoprotein
| 分子 | 名称: Nucleoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/Wilson-Smith/1933(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Wilson-Smith/1933(H1N1)) (A型インフルエンザウイルス) |
| 分子量 | 理論値: 53.114328 KDa |
| 配列 | 文字列: NATEIRASVG KMIDGIGRFY IQMCTELKLS DYEGRLIQNS LTIERMVLSA FDERRNKYLE EHPSAGKDPK KTGGPIYRRV DGKWRRELI LYDKEEIRRI WRQANNGDDA TAGLTHMMIW HSNLNDATYQ RTRALVRTGM DPRMCSLMQG STLPRRSGAA G AAVKGVGT ...文字列: NATEIRASVG KMIDGIGRFY IQMCTELKLS DYEGRLIQNS LTIERMVLSA FDERRNKYLE EHPSAGKDPK KTGGPIYRRV DGKWRRELI LYDKEEIRRI WRQANNGDDA TAGLTHMMIW HSNLNDATYQ RTRALVRTGM DPRMCSLMQG STLPRRSGAA G AAVKGVGT MVMELIRMIK RGINDRNFWR GENGRRTRIA YERMCNILKG KFQTAAQRTM VDQVRESRNP GNAEFEDLIF LA RSALILR GSVAHKSCLP ACVYGSAVAS GYDFEREGYS LVGIDPFRLL QNSQVYSLIR PNENPAHKSQ LVWMACHSAA FED LRVSSF IRGTKVVPRG KLSTRGVQIA SNENMETMES STLELRSRYW AIRTRSGGNT NQQRASSGQI SIQPTFSVQR NLPF DRPTI MAAFTGNTEG RTSDMRTEII RLMESARPED VSFQGRGVFE LSDEKATSPI VPSFDMSNEG SYFF UniProtKB: Nucleoprotein |
-分子 #2: Nucleoprotein
| 分子 | 名称: Nucleoprotein / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/Wilson-Smith/1933(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Wilson-Smith/1933(H1N1)) (A型インフルエンザウイルス) |
| 分子量 | 理論値: 2.107346 KDa |
| 配列 | 文字列: SSGQISIQPT FSVQRNLPF UniProtKB: Nucleoprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: TN buffer (50 nM Tris-HCl, 150 mM KCl) |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER/RHODIUM / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.01 kPa |
| 凍結 | 凍結剤: ETHANE / 装置: LEICA EM CPC |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - 画像ごとのフレーム数: 3-68 / 実像数: 420 / 平均電子線量: 2.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 34.16 Å 想定した対称性 - らせんパラメータ - ΔΦ: -65.95 ° 想定した対称性 - らせんパラメータ - 軸対称性: D1 (2回x1回 2面回転対称) アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 10.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: RELION (ver. 2.0) / 詳細: MonoRes software in Xmipp/Scipion / 使用した粒子像数: 2867 |
|---|---|
| Segment selection | 選択した数: 137461 / ソフトウェア - 名称: Xmipp / 詳細: Manual picking |
| 初期モデル | モデルのタイプ: OTHER 詳細: Initial model created using Iterative Helical Space Reconstruction protocols. |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: RELION (ver. 2.0) |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X