+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 Omicron-EG.5.1 one S1 disordered Spike Protein Trimer (S-GSAS-Omicron-EG.5.1) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SARS-COV-2 / Glycoprotein / Trimer / VIRAL PROTEIN | |||||||||
| 生物種 |  | |||||||||
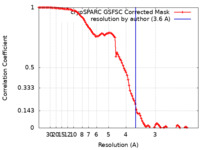
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Zhang QE / Acharya P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2024 ジャーナル: Mol Cell / 年: 2024タイトル: SARS-CoV-2 Omicron XBB lineage spike structures, conformations, antigenicity, and receptor recognition. 著者: Qianyi E Zhang / Jared Lindenberger / Ruth J Parsons / Bhishem Thakur / Rob Parks / Chan Soo Park / Xiao Huang / Salam Sammour / Katarzyna Janowska / Taylor N Spence / Robert J Edwards / ...著者: Qianyi E Zhang / Jared Lindenberger / Ruth J Parsons / Bhishem Thakur / Rob Parks / Chan Soo Park / Xiao Huang / Salam Sammour / Katarzyna Janowska / Taylor N Spence / Robert J Edwards / Mitchell Martin / Wilton B Williams / Sophie Gobeil / David C Montefiori / Bette Korber / Kevin O Saunders / Barton F Haynes / Rory Henderson / Priyamvada Acharya /   要旨: A recombinant lineage of the severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) Omicron variant, named XBB, appeared in late 2022 and evolved descendants that successively swept local and ...A recombinant lineage of the severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) Omicron variant, named XBB, appeared in late 2022 and evolved descendants that successively swept local and global populations. XBB lineage members were noted for their improved immune evasion and transmissibility. Here, we determine cryoelectron microscopy (cryo-EM) structures of XBB.1.5, XBB.1.16, EG.5, and EG.5.1 spike (S) ectodomains to reveal reinforced 3-receptor binding domain (RBD)-down receptor-inaccessible closed states mediated by interprotomer RBD interactions previously observed in BA.1 and BA.2. Improved XBB.1.5 and XBB.1.16 RBD stability compensated for stability loss caused by early Omicron mutations, while the F456L substitution reduced EG.5 RBD stability. S1 subunit mutations had long-range impacts on conformation and epitope presentation in the S2 subunit. Our results reveal continued S protein evolution via simultaneous optimization of multiple parameters, including stability, receptor binding, and immune evasion, and the dramatic effects of relatively few residue substitutions in altering the S protein conformational landscape. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44006.map.gz emd_44006.map.gz | 118.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44006-v30.xml emd-44006-v30.xml emd-44006.xml emd-44006.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44006_fsc.xml emd_44006_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44006.png emd_44006.png | 89.9 KB | ||
| Filedesc metadata |  emd-44006.cif.gz emd-44006.cif.gz | 5.9 KB | ||
| その他 |  emd_44006_half_map_1.map.gz emd_44006_half_map_1.map.gz emd_44006_half_map_2.map.gz emd_44006_half_map_2.map.gz | 116 MB 116 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44006 http://ftp.pdbj.org/pub/emdb/structures/EMD-44006 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44006 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44006 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44006_validation.pdf.gz emd_44006_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44006_full_validation.pdf.gz emd_44006_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_44006_validation.xml.gz emd_44006_validation.xml.gz | 19.1 KB | 表示 | |
| CIF形式データ |  emd_44006_validation.cif.gz emd_44006_validation.cif.gz | 24.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44006 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44006 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44006 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44006 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8uirC  8uk1C  8ukdC  8ukfC  8v0lC  8v0mC  8v0nC  8v0oC  8v0pC  8v0qC  8v0rC  8v0sC  8v0tC  8v0uC  8v0vC  8v0wC  8v0xC  9aywC  9ayxC  9ayyC C: 同じ文献を引用 ( |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44006.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44006.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
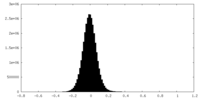
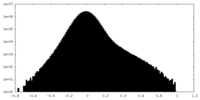
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_44006_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
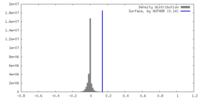
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_44006_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
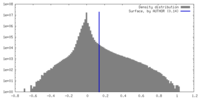
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GSAS-EG.5.1
| 全体 | 名称: GSAS-EG.5.1 |
|---|---|
| 要素 |
|
-超分子 #1: GSAS-EG.5.1
| 超分子 | 名称: GSAS-EG.5.1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLIT RTQSYTNSFT RGVYYPDKVF RSSVLHSTHD LFLPFFSNVT WFHAIHVSGT NGTKRFDNPA LPFNDGVYF ASTEKSNIIR GWIFGTTLDS KTQSLLIVNN ATNVVIKVCE FQFCNDPFLD VYQKNNKSWM ESEFRVYSSA N NCTFEYVS ...文字列: MFVFLVLLPL VSSQCVNLIT RTQSYTNSFT RGVYYPDKVF RSSVLHSTHD LFLPFFSNVT WFHAIHVSGT NGTKRFDNPA LPFNDGVYF ASTEKSNIIR GWIFGTTLDS KTQSLLIVNN ATNVVIKVCE FQFCNDPFLD VYQKNNKSWM ESEFRVYSSA N NCTFEYVS QPFLMDLEGK EGNFKNLREF VFKNIDGYFK IYSKHTPINL ERDLPQGFSA LEPLVDLPIG INITRFQTLL AL HRSYLTP VDSSSGWTAG AAAYYVGYLQ PRTFLLKYNE NGTITDAVDC ALDPLSETKC TLKSFTVEKG IYQTSNFRVQ PTE SIVRFP NITNLCPFHE VFNATTFASV YAWNRKRISN CVADYSVIYN FAPFFAFKCY GVSPTKLNDL CFTNVYADSF VIRG NEVSQ IAPGQTGNIA DYNYKLPDDF TGCVIAWNSN KLDSKPSGNY NYLYRLLRKS KLKPFERDIS TEIYQAGNKP CNGVA GPNC YSPLQSYGFR PTYGVGHQPY RVVVLSFELL HAPATVCGPK KSTNLVKNKC VNFNFNGLTG TGVLTESNKK FLPFQQ FGR DIADTTDAVR DPQTLEILDI TPCSFGGVSV ITPGTNTSNQ VAVLYQGVNC TEVPVAIHAD QLTPTWRVYS TGSNVFQ TR AGCLIGAEYV NNSYECDIPI GAGICASYQT QTKSHGSASS VASQSIIAYT MSLGAENSVA YSNNSIAIPT NFTISVTT E ILPVSMTKTS VDCTMYICGD STECSNLLLQ YGSFCTQLKR ALTGIAVEQD KNTQEVFAQV KQIYKTPPIK YFGGFNFSQ ILPDPSKPSK RSFIEDLLFN KVTLADAGFI KQYGDCLGDI AARDLICAQK FNGLTVLPPL LTDEMIAQYT SALLAGTITS GWTFGAGAA LQIPFAMQMA YRFNGIGVTQ NVLYENQKLI ANQFNSAIGK IQDSLSSTAS ALGKLQDVVN HNAQALNTLV K QLSSKFGA ISSVLNDILS RLDKVEAEVQ IDRLITGRLQ SLQTYVTQQL IRAAEIRASA NLAATKMSEC VLGQSKRVDF CG KGYHLMS FPQSAPHGVV FLHVTYVPAQ EKNFTTAPAI CHDGKAHFPR EGVFVSNGTH WFVTQRNFYE PQIITTDNTF VSG NCDVVI GIVNNTVYDP LQPELDSFKE ELDKYFKNHT SPDVDLGDIS GINASVVNIQ KEIDRLNEVA KNLNESLIDL QELG KYEQG SGYIPEAPRD GQAYVRKDGE WVLLSTFLGR SLEVLFQGPG HHHHHHHHSA WSHPQFEKGG GSGGGGSGGS AWSHP QFEK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 295.15 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 実像数: 11757 / 平均電子線量: 57.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm 最小 デフォーカス(公称値): 0.35000000000000003 µm 倍率(公称値): 81000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー








































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)