+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4179 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Combined solid-state NMR, solution-state NMR and EM data for structure determination of the tetrahedral aminopeptidase TET2 from P. horikoshii | ||||||||||||
 マップデータ マップデータ | CryoEM map of the H78C/H248C mutant tetrahedral aminopeptidase from P. horikoshii | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | peptidase / protein quality control / oligomer / aminopeptidase / PEPTIDE BINDING PROTEIN | ||||||||||||
| 機能・相同性 | Peptidase M42, domain 2 / M42 glutamyl aminopeptidase / Peptidase M42 / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アミノペプチターゼ / aminopeptidase activity / metallopeptidase activity / proteolysis / metal ion binding / Tetrahedral aminopeptidase 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |   Pyrococcus horikoshii (古細菌) / Pyrococcus horikoshii (古細菌) /   Pyrococcus horikoshii (strain ATCC 700860 / DSM 12428 / JCM 9974 / NBRC 100139 / OT-3) (古細菌) Pyrococcus horikoshii (strain ATCC 700860 / DSM 12428 / JCM 9974 / NBRC 100139 / OT-3) (古細菌) | ||||||||||||
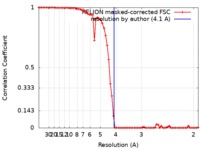
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | ||||||||||||
 データ登録者 データ登録者 | Gauto DF / Estrozi LF / Schwieters CD | ||||||||||||
| 資金援助 |  フランス, 3件 フランス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Integrated NMR and cryo-EM atomic-resolution structure determination of a half-megadalton enzyme complex. 著者: Diego F Gauto / Leandro F Estrozi / Charles D Schwieters / Gregory Effantin / Pavel Macek / Remy Sounier / Astrid C Sivertsen / Elena Schmidt / Rime Kerfah / Guillaume Mas / Jacques-Philippe ...著者: Diego F Gauto / Leandro F Estrozi / Charles D Schwieters / Gregory Effantin / Pavel Macek / Remy Sounier / Astrid C Sivertsen / Elena Schmidt / Rime Kerfah / Guillaume Mas / Jacques-Philippe Colletier / Peter Güntert / Adrien Favier / Guy Schoehn / Paul Schanda / Jerome Boisbouvier /      要旨: Atomic-resolution structure determination is crucial for understanding protein function. Cryo-EM and NMR spectroscopy both provide structural information, but currently cryo-EM does not routinely ...Atomic-resolution structure determination is crucial for understanding protein function. Cryo-EM and NMR spectroscopy both provide structural information, but currently cryo-EM does not routinely give access to atomic-level structural data, and, generally, NMR structure determination is restricted to small (<30 kDa) proteins. We introduce an integrated structure determination approach that simultaneously uses NMR and EM data to overcome the limits of each of these methods. The approach enables structure determination of the 468 kDa large dodecameric aminopeptidase TET2 to a precision and accuracy below 1 Å by combining secondary-structure information obtained from near-complete magic-angle-spinning NMR assignments of the 39 kDa-large subunits, distance restraints from backbone amides and ILV methyl groups, and a 4.1 Å resolution EM map. The resulting structure exceeds current standards of NMR and EM structure determination in terms of molecular weight and precision. Importantly, the approach is successful even in cases where only medium-resolution cryo-EM data are available. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4179.map.gz emd_4179.map.gz | 12.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4179-v30.xml emd-4179-v30.xml emd-4179.xml emd-4179.xml | 14.5 KB 14.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4179_fsc.xml emd_4179_fsc.xml | 6.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4179_1.png emd_4179_1.png emd_4179_2.png emd_4179_2.png | 111.3 KB 86.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4179 http://ftp.pdbj.org/pub/emdb/structures/EMD-4179 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4179 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4179 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4179_validation.pdf.gz emd_4179_validation.pdf.gz | 597.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4179_full_validation.pdf.gz emd_4179_full_validation.pdf.gz | 597.4 KB | 表示 | |
| XML形式データ |  emd_4179_validation.xml.gz emd_4179_validation.xml.gz | 9.6 KB | 表示 | |
| CIF形式データ |  emd_4179_validation.cif.gz emd_4179_validation.cif.gz | 12.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4179 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4179 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4179 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4179 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4179.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4179.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of the H78C/H248C mutant tetrahedral aminopeptidase from P. horikoshii | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.97 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : tetrahedral amino-peptidase from P. horikoshii
| 全体 | 名称: tetrahedral amino-peptidase from P. horikoshii |
|---|---|
| 要素 |
|
-超分子 #1: tetrahedral amino-peptidase from P. horikoshii
| 超分子 | 名称: tetrahedral amino-peptidase from P. horikoshii / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: H78C/H248C mutant |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus horikoshii (古細菌) Pyrococcus horikoshii (古細菌) |
| 分子量 | 理論値: 490 KDa |
-分子 #1: Tetrahedral aminopeptidase
| 分子 | 名称: Tetrahedral aminopeptidase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アミノペプチターゼ |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus horikoshii (strain ATCC 700860 / DSM 12428 / JCM 9974 / NBRC 100139 / OT-3) (古細菌) Pyrococcus horikoshii (strain ATCC 700860 / DSM 12428 / JCM 9974 / NBRC 100139 / OT-3) (古細菌)株: ATCC 700860 / DSM 12428 / JCM 9974 / NBRC 100139 / OT-3 |
| 分子量 | 理論値: 39.071027 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEVRNMVDYE LLKKVVEAPG VSGYEFLGIR DVVIEEIKDY VDEVKVDKLG NVIAHKKGEG PKVMIAAHMD QIGLMVTHIE KNGFLRVAP IGGVDPKTLI AQRFKVWIDK GKFIYGVGAS VPPHIQKPED RKKAPDWDQI FIDIGAESKE EAEDMGVKIG T VITWDGRL ...文字列: MEVRNMVDYE LLKKVVEAPG VSGYEFLGIR DVVIEEIKDY VDEVKVDKLG NVIAHKKGEG PKVMIAAHMD QIGLMVTHIE KNGFLRVAP IGGVDPKTLI AQRFKVWIDK GKFIYGVGAS VPPHIQKPED RKKAPDWDQI FIDIGAESKE EAEDMGVKIG T VITWDGRL ERLGKHRFVS IAFDDRIAVY TILEVAKQLK DAKADVYFVA TVQEEVGLRG ARTSAFGIEP DYGFAIDVTI AA DIPGTPE HKQVTHLGKG TAIKIMDRSV ICHPTIVRWL EELAKKHEIP YQLEILLGGG TDAGAIHLTK AGVPTGALSV PAR YIHSNT EVVDERDVDA TVELMTKALE NIHELKI UniProtKB: Tetrahedral aminopeptidase |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting time 2s, force 1, drain time 0.. |
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 90 / 平均露光時間: 4.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.5 µm / 最小 デフォーカス(補正後): 0.8 µm / 倍率(補正後): 25773 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 倍率(公称値): 20000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-6f3k:  PDB-6r8n: |
 ムービー
ムービー コントローラー
コントローラー