登録情報 データベース : EMDB / ID : EMD-41767タイトル Structure of human Wnt3a bound to WLS and CALR 複合体 : Wnt3a-WLS-CALR Complexタンパク質・ペプチド : Protein Wnt-3aタンパク質・ペプチド : Protein wntless homologタンパク質・ペプチド : Calreticulinリガンド : PALMITOLEIC ACIDリガンド : (2S)-3-(hexadecanoyloxy)-2-[(9Z)-octadec-9-enoyloxy]propyl 2-(trimethylammonio)ethyl phosphate機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 3.5 Å Qi X / Hu Q / Li X 資金援助 Organization Grant number 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) GM135343 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) HL160487 Welch Foundation I-1957
ジャーナル : Cell / 年 : 2023タイトル : Molecular basis of Wnt biogenesis, secretion, and Wnt7-specific signaling.著者 : Xiaofeng Qi / Qinli Hu / Nadia Elghobashi-Meinhardt / Tao Long / Hongwen Chen / Xiaochun Li / 要旨 : Wnt proteins are enzymatically lipidated by Porcupine (PORCN) in the ER and bind to Wntless (WLS) for intracellular transport and secretion. Mechanisms governing the transfer of these low-solubility ... Wnt proteins are enzymatically lipidated by Porcupine (PORCN) in the ER and bind to Wntless (WLS) for intracellular transport and secretion. Mechanisms governing the transfer of these low-solubility Wnts from the ER to the extracellular space remain unclear. Through structural and functional analyses of Wnt7a, a crucial Wnt involved in central nervous system angiogenesis and blood-brain barrier maintenance, we have elucidated the principles of Wnt biogenesis and Wnt7-specific signaling. The Wnt7a-WLS complex binds to calreticulin (CALR), revealing that CALR functions as a chaperone to facilitate Wnt transfer from PORCN to WLS during Wnt biogenesis. Our structures, functional analyses, and molecular dynamics simulations demonstrate that a phospholipid in the core of Wnt-bound WLS regulates the association and dissociation between Wnt and WLS, suggesting a lipid-mediated Wnt secretion mechanism. Finally, the structure of Wnt7a bound to RECK, a cell-surface Wnt7 co-receptor, reveals how RECK engages the N-terminal domain of Wnt7a to activate Wnt7-specific signaling. 履歴 登録 2023年8月27日 - ヘッダ(付随情報) 公開 2023年10月18日 - マップ公開 2023年10月18日 - 更新 2024年10月30日 - 現状 2024年10月30日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: Cell / 年: 2023
ジャーナル: Cell / 年: 2023

 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_41767.map.gz
emd_41767.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-41767-v30.xml
emd-41767-v30.xml emd-41767.xml
emd-41767.xml EMDBヘッダ
EMDBヘッダ emd_41767.png
emd_41767.png emd-41767.cif.gz
emd-41767.cif.gz emd_41767_half_map_1.map.gz
emd_41767_half_map_1.map.gz emd_41767_half_map_2.map.gz
emd_41767_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-41767
http://ftp.pdbj.org/pub/emdb/structures/EMD-41767 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41767
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41767 emd_41767_validation.pdf.gz
emd_41767_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_41767_full_validation.pdf.gz
emd_41767_full_validation.pdf.gz emd_41767_validation.xml.gz
emd_41767_validation.xml.gz emd_41767_validation.cif.gz
emd_41767_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41767
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41767 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41767
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41767 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_41767.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_41767.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)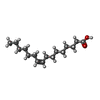

 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)