+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3bmq | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of Pteridine Reductase 1 (PTR1) from Trypanosoma brucei in ternary complex with cofactor (NADP+) and inhibitor (Compound AX5) | ||||||
 要素 要素 | (Pteridine reductase) x 2 | ||||||
 キーワード キーワード | OXIDOREDUCTASE / pteridine reductase / ptr1 / trypanosoma brucei / short chain dehydrogenase / inhibitor | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.7 Å 分子置換 / 解像度: 1.7 Å | ||||||
 データ登録者 データ登録者 | Martini, V.P. / Iulek, J. / Hunter, W.N. | ||||||
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2010 ジャーナル: J.Med.Chem. / 年: 2010タイトル: Structure-based design of pteridine reductase inhibitors targeting african sleeping sickness and the leishmaniases. 著者: Tulloch, L.B. / Martini, V.P. / Iulek, J. / Huggan, J.K. / Lee, J.H. / Gibson, C.L. / Smith, T.K. / Suckling, C.J. / Hunter, W.N. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3bmq.cif.gz 3bmq.cif.gz | 237.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3bmq.ent.gz pdb3bmq.ent.gz | 199 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3bmq.json.gz 3bmq.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3bmq_validation.pdf.gz 3bmq_validation.pdf.gz | 1.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3bmq_full_validation.pdf.gz 3bmq_full_validation.pdf.gz | 1.7 MB | 表示 | |
| XML形式データ |  3bmq_validation.xml.gz 3bmq_validation.xml.gz | 56.8 KB | 表示 | |
| CIF形式データ |  3bmq_validation.cif.gz 3bmq_validation.cif.gz | 84.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bm/3bmq https://data.pdbj.org/pub/pdb/validation_reports/bm/3bmq ftp://data.pdbj.org/pub/pdb/validation_reports/bm/3bmq ftp://data.pdbj.org/pub/pdb/validation_reports/bm/3bmq | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3bmcC  3bmnC  3bmoC  3jq6C  3jq7C  3jq8C  3jq9C  3jqaC  3jqbC  3jqcC  3jqdC  3jqeC  3jqfC  3jqgC  3bms  3bmt C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 2種, 4分子 ADBC
| #1: タンパク質 | 分子量: 30685.787 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: PTR1 / プラスミド: pET15b / 発現宿主:  #2: タンパク質 | 分子量: 30669.791 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: PTR1 / プラスミド: pET15b / 発現宿主:  |
|---|
-非ポリマー , 6種, 1376分子 
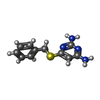









| #3: 化合物 | ChemComp-NAP / #4: 化合物 | ChemComp-AX5 / #5: 化合物 | #6: 化合物 | #7: 化合物 | #8: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.07 Å3/Da / 溶媒含有率: 40.62 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.5 詳細: 2-3M sodium acetate, 10-100mM sodium citrate, pH 4.5, vapor diffusion, hanging drop, temperature 293K |
-データ収集
| 回折 | 平均測定温度: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SRS SRS  / ビームライン: PX14.1 / ビームライン: PX14.1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 相対比: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 1.7→74.625 Å / Num. obs: 94013 / % possible obs: 85.8 % / 冗長度: 5.6 % / Rmerge(I) obs: 0.055 / Rsym value: 0.055 / Net I/σ(I): 8.4 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
-位相決定
| 位相決定 | 手法:  分子置換 分子置換 |
|---|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 1.7→74.54 Å / Cor.coef. Fo:Fc: 0.974 / Cor.coef. Fo:Fc free: 0.95 / SU B: 3.182 / SU ML: 0.059 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.102 / ESU R Free: 0.106 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 分子置換 / 解像度: 1.7→74.54 Å / Cor.coef. Fo:Fc: 0.974 / Cor.coef. Fo:Fc free: 0.95 / SU B: 3.182 / SU ML: 0.059 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.102 / ESU R Free: 0.106 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS All of the 17 related structures are expressed from the same DNA construct, which encodes CYS at positions 59 and 168. Crystals harvested ...詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS All of the 17 related structures are expressed from the same DNA construct, which encodes CYS at positions 59 and 168. Crystals harvested within a couple of days of formation contain CYS at positions 59 and 168. However these two residues appear quite reactive and over time become oxidised to CSX, as determined by the emergence in older crystals of electron density for the OD atom. Sometimes CYS168 reacts with DTT in the crystallisation buffer, covalently linking the two molecules by an S-S bond.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 9.163 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.7→74.54 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.7→1.744 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー























 PDBj
PDBj












