+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3715 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | T=4 HBV virus-like particle. | |||||||||
 マップデータ マップデータ | T=4 Hepatitis B virus virus-like particle | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Hepatitis B virus (B 型肝炎ウイルス) Hepatitis B virus (B 型肝炎ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Patel N / White SJ / Thompson RF / Bingham R / Weiss EU / Maskell DP / Zlotnick A / Dykeman E / Tuma R / Twarock R ...Patel N / White SJ / Thompson RF / Bingham R / Weiss EU / Maskell DP / Zlotnick A / Dykeman E / Tuma R / Twarock R / Ranson NA / Stockley PG | |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2017 ジャーナル: Nat Microbiol / 年: 2017タイトル: HBV RNA pre-genome encodes specific motifs that mediate interactions with the viral core protein that promote nucleocapsid assembly. 著者: Nikesh Patel / Simon J White / Rebecca F Thompson / Richard Bingham / Eva U Weiß / Daniel P Maskell / Adam Zlotnick / Eric Dykeman / Roman Tuma / Reidun Twarock / Neil A Ranson / Peter G Stockley /   要旨: Formation of the hepatitis B virus nucleocapsid is an essential step in the viral lifecycle, but its assembly is not fully understood. We report the discovery of sequence-specific interactions ...Formation of the hepatitis B virus nucleocapsid is an essential step in the viral lifecycle, but its assembly is not fully understood. We report the discovery of sequence-specific interactions between the viral pre-genome and the hepatitis B core protein that play roles in defining the nucleocapsid assembly pathway. Using RNA SELEX and bioinformatics, we identified multiple regions in the pre-genomic RNA with high affinity for core protein dimers. These RNAs form stem-loops with a conserved loop motif that trigger sequence-specific assembly of virus-like particles (VLPs) at much higher fidelity and yield than in the absence of RNA. The RNA oligos do not interact with preformed RNA-free VLPs, so their effects must occur during particle assembly. Asymmetric cryo-electron microscopy reconstruction of the T = 4 VLPs assembled in the presence of one of the RNAs reveals a unique internal feature connected to the main core protein shell via lobes of density. Biophysical assays suggest that this is a complex involving several RNA oligos interacting with the C-terminal arginine-rich domains of core protein. These core protein-RNA contacts may play one or more roles in regulating the organization of the pre-genome during nucleocapsid assembly, facilitating subsequent reverse transcription and acting as a nucleation complex for nucleocapsid assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3715.map.gz emd_3715.map.gz | 78.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3715-v30.xml emd-3715-v30.xml emd-3715.xml emd-3715.xml | 12.9 KB 12.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3715_fsc.xml emd_3715_fsc.xml | 13.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3715.png emd_3715.png | 204.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3715 http://ftp.pdbj.org/pub/emdb/structures/EMD-3715 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3715 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3715 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3715_validation.pdf.gz emd_3715_validation.pdf.gz | 267 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3715_full_validation.pdf.gz emd_3715_full_validation.pdf.gz | 266.1 KB | 表示 | |
| XML形式データ |  emd_3715_validation.xml.gz emd_3715_validation.xml.gz | 13.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3715 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3715 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3715 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3715 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3715.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3715.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T=4 Hepatitis B virus virus-like particle | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
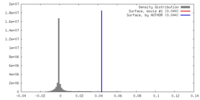
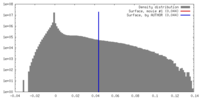
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Hepatitis B virus
| 全体 | 名称:   Hepatitis B virus (B 型肝炎ウイルス) Hepatitis B virus (B 型肝炎ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Hepatitis B virus
| 超分子 | 名称: Hepatitis B virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Is adw strain but with some mutations from deposited sequence. NCBI-ID: 10407 / 生物種: Hepatitis B virus / Sci species strain: adw / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| Host system | 生物種:  |
| ウイルス殻 | Shell ID: 1 / T番号(三角分割数): 4 |
-分子 #1: Hepatitis B virus core antigen
| 分子 | 名称: Hepatitis B virus core antigen / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Hepatitis B virus (B 型肝炎ウイルス) / 株: adw Hepatitis B virus (B 型肝炎ウイルス) / 株: adw |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDIDPYKEFG ATVELLSFL P SDFFPSVR DL LDTASAL YRE ALESPE HCSP HHTAL RQAIL CWGE LMTLAT WVG NNLEDPA SR DLVVNYVN T NMGLKIRQL LWFHISCLTF GRETVLEYL V SFGVWIRT PP AYRPPNA PIL STLPET TVVR RRDRG ...文字列: MDIDPYKEFG ATVELLSFL P SDFFPSVR DL LDTASAL YRE ALESPE HCSP HHTAL RQAIL CWGE LMTLAT WVG NNLEDPA SR DLVVNYVN T NMGLKIRQL LWFHISCLTF GRETVLEYL V SFGVWIRT PP AYRPPNA PIL STLPET TVVR RRDRG RSPRR RTPS PRRRRS QSP RRRRSQS RE SQC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 5.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 281 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 2397 / 平均露光時間: 2.5 sec. / 平均電子線量: 67.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)