+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31906 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
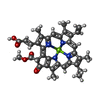
| タイトル | Structure of S1M1-type FCPII complex from diatom | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Photosystem / PSII / ELECTRON TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報light-harvesting complex / photosynthesis, light harvesting in photosystem I / plastid / chlorophyll binding / response to light stimulus / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) | |||||||||
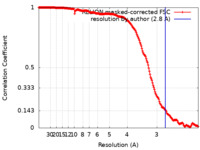
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Nagao R / Kato K | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural basis for different types of hetero-tetrameric light-harvesting complexes in a diatom PSII-FCPII supercomplex 著者: Nagao R / Kato K / Kumazawa M / Ifuku K / Yokono M / Suzuki T / Dohmae N / Akita F / Akimoto S / Miyazaki N / Shen JR | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31906.map.gz emd_31906.map.gz | 29.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31906-v30.xml emd-31906-v30.xml emd-31906.xml emd-31906.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31906_fsc.xml emd_31906_fsc.xml | 18 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31906.png emd_31906.png | 111.7 KB | ||
| Filedesc metadata |  emd-31906.cif.gz emd-31906.cif.gz | 6.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31906 http://ftp.pdbj.org/pub/emdb/structures/EMD-31906 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31906 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31906 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31906_validation.pdf.gz emd_31906_validation.pdf.gz | 356 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31906_full_validation.pdf.gz emd_31906_full_validation.pdf.gz | 355.5 KB | 表示 | |
| XML形式データ |  emd_31906_validation.xml.gz emd_31906_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_31906_validation.cif.gz emd_31906_validation.cif.gz | 22.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31906 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31906 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31906 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31906 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31906.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31906.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.113 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : S1M1-type FCPII complex
+超分子 #1: S1M1-type FCPII complex
+分子 #1: Chlorophyll a/b-binding protein
+分子 #2: Fcpb2, Fucoxanthin chlorophyll a/c-binding protein
+分子 #3: Fcpb3, Fucoxanthin chlorophyll a/c-binding protein
+分子 #4: Fcpb4, Fucoxanthin chlorophyll a/c-binding protein
+分子 #5: Fcpb5, Fucoxanthin chlorophyll a/c-binding protein
+分子 #6: Fcpb6, Fucoxanthin chlorophyll a/c-binding protein
+分子 #7: Fcpb7, Fucoxanthin chlorophyll a/c-binding protein
+分子 #8: CHLOROPHYLL A
+分子 #9: Chlorophyll c1
+分子 #10: (3S,3'S,5R,5'R,6S,6'R,8'R)-3,5'-dihydroxy-8-oxo-6',7'-didehydro-5...
+分子 #11: 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE
+分子 #12: UNKNOWN LIGAND
+分子 #13: (3S,3'R,5R,6S,7cis)-7',8'-didehydro-5,6-dihydro-5,6-epoxy-beta,be...
+分子 #14: 1,2-DI-O-ACYL-3-O-[6-DEOXY-6-SULFO-ALPHA-D-GLUCOPYRANOSYL]-SN-GLYCEROL
+分子 #15: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #16: DODECYL-ALPHA-D-MALTOSIDE
+分子 #17: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.256 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.5 / 構成要素:
| ||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)