+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30327 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mycobacterium smegmatis SugABC complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ABC transporter / Lipoprotein / Mycobacteria / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報carbohydrate transport / ABC-type transporter activity / ATP-binding cassette (ABC) transporter complex / transmembrane transport / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Mycolicibacterium smegmatis MC2 155 (バクテリア) / Mycolicibacterium smegmatis MC2 155 (バクテリア) /  Mycolicibacterium smegmatis (strain ATCC 700084 / mc(2)155) (バクテリア) Mycolicibacterium smegmatis (strain ATCC 700084 / mc(2)155) (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.41 Å | |||||||||
 データ登録者 データ登録者 | Liu F / Liang J | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2020 ジャーナル: Sci Adv / 年: 2020タイトル: Structural basis of trehalose recycling by the ABC transporter LpqY-SugABC. 著者: Fengjiang Liu / Jingxi Liang / Bing Zhang / Yan Gao / Xiuna Yang / Tianyu Hu / Haitao Yang / Wenqing Xu / Luke W Guddat / Zihe Rao /   要旨: In bacteria, adenosine 5'-triphosphate (ATP)-binding cassette (ABC) importers are essential for the uptake of nutrients including the nonreducing disaccharide trehalose, a metabolite that is crucial ...In bacteria, adenosine 5'-triphosphate (ATP)-binding cassette (ABC) importers are essential for the uptake of nutrients including the nonreducing disaccharide trehalose, a metabolite that is crucial for the survival and virulence of several human pathogens including SugABC is an ABC transporter that translocates trehalose from the periplasmic lipoprotein LpqY into the cytoplasm of mycobacteria. Here, we report four high-resolution cryo-electron microscopy structures of the mycobacterial LpqY-SugABC complex to reveal how it binds and passes trehalose through the membrane to the cytoplasm. A unique feature observed in this system is the initial mode of capture of the trehalose at the LpqY interface. Uptake is achieved by a pivotal rotation of LpqY relative to SugABC, moving from an open and accessible conformation to a clamped conformation upon trehalose binding. These findings enrich our understanding as to how ABC transporters facilitate substrate transport across the membrane in Gram-positive bacteria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30327.map.gz emd_30327.map.gz | 200.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30327-v30.xml emd-30327-v30.xml emd-30327.xml emd-30327.xml | 12.4 KB 12.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_30327.png emd_30327.png | 52.8 KB | ||
| Filedesc metadata |  emd-30327.cif.gz emd-30327.cif.gz | 5.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30327 http://ftp.pdbj.org/pub/emdb/structures/EMD-30327 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30327 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30327 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_30327_validation.pdf.gz emd_30327_validation.pdf.gz | 546.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_30327_full_validation.pdf.gz emd_30327_full_validation.pdf.gz | 545.9 KB | 表示 | |
| XML形式データ |  emd_30327_validation.xml.gz emd_30327_validation.xml.gz | 7 KB | 表示 | |
| CIF形式データ |  emd_30327_validation.cif.gz emd_30327_validation.cif.gz | 8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30327 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30327 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30327 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30327 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30327.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30327.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
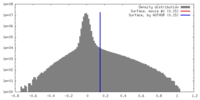
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Mycobacterium smegmatis SugABC complex
| 全体 | 名称: Mycobacterium smegmatis SugABC complex |
|---|---|
| 要素 |
|
-超分子 #1: Mycobacterium smegmatis SugABC complex
| 超分子 | 名称: Mycobacterium smegmatis SugABC complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
-分子 #1: ABC transporter, ATP-binding protein SugC
| 分子 | 名称: ABC transporter, ATP-binding protein SugC / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis (strain ATCC 700084 / mc(2)155) (バクテリア) Mycolicibacterium smegmatis (strain ATCC 700084 / mc(2)155) (バクテリア)株: ATCC 700084 / mc(2)155 |
| 分子量 | 理論値: 43.703609 KDa |
| 組換発現 | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 配列 | 文字列: MAEIVLDRVT KSYPDGAGGV RAAVKEFSMT IADGEFIILV GPSGCGKSTT LNMIAGLEEI TSGELRIGGE RVNEKAPKDR DIAMVFQSY ALYPHMTVRQ NIAFPLTLAK VPKAEIAAKV EETAKILDLS ELLDRKPGQL SGGQRQRVAM GRAIVRSPKA F LMDEPLSN ...文字列: MAEIVLDRVT KSYPDGAGGV RAAVKEFSMT IADGEFIILV GPSGCGKSTT LNMIAGLEEI TSGELRIGGE RVNEKAPKDR DIAMVFQSY ALYPHMTVRQ NIAFPLTLAK VPKAEIAAKV EETAKILDLS ELLDRKPGQL SGGQRQRVAM GRAIVRSPKA F LMDEPLSN LDAKLRVQMR AEISRLQDRL GTTTVYVTHD QTEAMTLGDR VVVMLAGEVQ QIGTPDELYS SPANLFVAGF IG SPAMNFF PATRTDVGVR LPFGEVTLTP HMLDLLDKQA RPENIIVGIR PEHIEDSALL DGYARIRALT FSVRADIVES LGA DKYVHF TTEGAGAESA QLAELAADSG AGTNQFIARV SADSRVRTGE QIELAIDTTK LSIFDAATGL NLTRDITPTD PTEA AGPDA G UniProtKB: ABC transporter, ATP-binding protein SugC |
-分子 #2: ABC sugar transporter, permease component
| 分子 | 名称: ABC sugar transporter, permease component / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis (strain ATCC 700084 / mc(2)155) (バクテリア) Mycolicibacterium smegmatis (strain ATCC 700084 / mc(2)155) (バクテリア)株: ATCC 700084 / mc(2)155 |
| 分子量 | 理論値: 32.73925 KDa |
| 組換発現 | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 配列 | 文字列: MTAAVTPSAS AVASDDKKSE RRLAFWLIAP AVLLMLAVTA YPIGYAVWLS LQRYNLAEPH DTEFIGLANY VTVLTDGYWW TAFAVTLGI TVVSVAIEFA LGLALALVMH RTIFGKGAVR TAILIPYGIV TVAASYSWYY AWTPGTGYLA NLLPEGSAPL T DQLPSLAI ...文字列: MTAAVTPSAS AVASDDKKSE RRLAFWLIAP AVLLMLAVTA YPIGYAVWLS LQRYNLAEPH DTEFIGLANY VTVLTDGYWW TAFAVTLGI TVVSVAIEFA LGLALALVMH RTIFGKGAVR TAILIPYGIV TVAASYSWYY AWTPGTGYLA NLLPEGSAPL T DQLPSLAI VVLAEVWKTT PFMALLLLAG LALVPQDLLN AAQVDGAGPW KRLTKVILPM IKPAILVALL FRTLDAFRIF DN IYILTGG SNDTGSVSIL GYDNLFKAFN VGLGSAISVL IFLSVAIIAF IYIKIFGAAA PGSDEEVR UniProtKB: ABC sugar transporter, permease component |
-分子 #3: ABC transporter, permease protein SugB
| 分子 | 名称: ABC transporter, permease protein SugB / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis (strain ATCC 700084 / mc(2)155) (バクテリア) Mycolicibacterium smegmatis (strain ATCC 700084 / mc(2)155) (バクテリア)株: ATCC 700084 / mc(2)155 |
| 分子量 | 理論値: 29.839441 KDa |
| 組換発現 | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 配列 | 文字列: MADRVDARRA TWWSVVNILV IVYALIPVLW ILSLSLKPTS SVKDGKLIPT EITFANYKAI FSGDAFTSAL FNSIGIGLIT TIIAVVIGG MAAYAVARLQ FPGKQLLIGV ALLIAMFPHI SLVTPIFNMW RGIGLFDTWP GLIIPYITFA LPLAIYTLSA F FREIPWDL ...文字列: MADRVDARRA TWWSVVNILV IVYALIPVLW ILSLSLKPTS SVKDGKLIPT EITFANYKAI FSGDAFTSAL FNSIGIGLIT TIIAVVIGG MAAYAVARLQ FPGKQLLIGV ALLIAMFPHI SLVTPIFNMW RGIGLFDTWP GLIIPYITFA LPLAIYTLSA F FREIPWDL EKAAKMDGAT PAQAFRKVIA PLAAPGIVTA AILVFIFAWN DLLLALSLTA TQRAITAPVA IANFTGSSQF EE PTGSIAA GAMVITIPII IFVLIFQRRI VAGLTSGAVK G UniProtKB: ABC transporter, permease protein SugB |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.41 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 197191 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)