+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | XRCC4-XLF-XRCC4 subcomplex of PAXX-LR complex | |||||||||
 マップデータ マップデータ | XXL in PAXX-LR | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / Kinase / NHEJ / DNA BINDING PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 14.0 Å | |||||||||
 データ登録者 データ登録者 | Chen S / He Y | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Cryo-EM visualization of DNA-PKcs structural intermediates in NHEJ. 著者: Siyu Chen / Alex Vogt / Linda Lee / Tasmin Naila / Ryan McKeown / Alan E Tomkinson / Susan P Lees-Miller / Yuan He /   要旨: DNA double-strand breaks (DSBs), one of the most cytotoxic forms of DNA damage, can be repaired by the tightly regulated nonhomologous end joining (NHEJ) machinery (Stinson and Loparo and Zhao ). ...DNA double-strand breaks (DSBs), one of the most cytotoxic forms of DNA damage, can be repaired by the tightly regulated nonhomologous end joining (NHEJ) machinery (Stinson and Loparo and Zhao ). Core NHEJ factors form an initial long-range (LR) synaptic complex that transitions into a DNA-PKcs (DNA-dependent protein kinase, catalytic subunit)-free, short-range state to align the DSB ends (Chen ). Using single-particle cryo-electron microscopy, we have visualized three additional key NHEJ complexes representing different transition states, with DNA-PKcs adopting distinct dimeric conformations within each of them. Upon DNA-PKcs autophosphorylation, the LR complex undergoes a substantial conformational change, with both Ku and DNA-PKcs rotating outward to promote DNA break exposure and DNA-PKcs dissociation. We also captured a dimeric state of catalytically inactive DNA-PKcs, which resembles structures of other PIKK (Phosphatidylinositol 3-kinase-related kinase) family kinases, revealing a model of the full regulatory cycle of DNA-PKcs during NHEJ. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28736.map.gz emd_28736.map.gz | 39.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28736-v30.xml emd-28736-v30.xml emd-28736.xml emd-28736.xml | 18.9 KB 18.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28736.png emd_28736.png | 19.2 KB | ||
| その他 |  emd_28736_half_map_1.map.gz emd_28736_half_map_1.map.gz emd_28736_half_map_2.map.gz emd_28736_half_map_2.map.gz | 32.5 MB 32.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28736 http://ftp.pdbj.org/pub/emdb/structures/EMD-28736 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28736 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28736 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28736_validation.pdf.gz emd_28736_validation.pdf.gz | 754.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28736_full_validation.pdf.gz emd_28736_full_validation.pdf.gz | 753.7 KB | 表示 | |
| XML形式データ |  emd_28736_validation.xml.gz emd_28736_validation.xml.gz | 11 KB | 表示 | |
| CIF形式データ |  emd_28736_validation.cif.gz emd_28736_validation.cif.gz | 12.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28736 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28736 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28736 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28736 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28736.map.gz / 形式: CCP4 / 大きさ: 43.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28736.map.gz / 形式: CCP4 / 大きさ: 43.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | XXL in PAXX-LR | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.158 Å | ||||||||||||||||||||||||||||||||||||
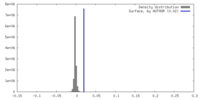
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half1
| ファイル | emd_28736_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
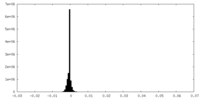
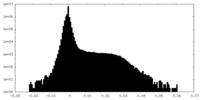
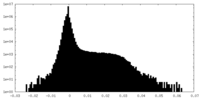
| 密度ヒストグラム |
-ハーフマップ: half2
| ファイル | emd_28736_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
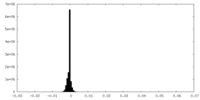
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NHEJ Long-range complex with PAXX
| 全体 | 名称: NHEJ Long-range complex with PAXX |
|---|---|
| 要素 |
|
-超分子 #1: NHEJ Long-range complex with PAXX
| 超分子 | 名称: NHEJ Long-range complex with PAXX / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.75 MDa |
-分子 #1: XRCC4_HUMAN DNA repair protein XRCC4
| 分子 | 名称: XRCC4_HUMAN DNA repair protein XRCC4 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MERKISRIHL VSEPSITHFL QVSWEKTLES GFVITLTDGH SAWTGTVSES EISQEADDMA MEKGKYVGEL RKALLSGAGP ADVYTFNFSK ESCYFFFEKN LKDVSFRLGS FNLEKVENPA EVIRELICYC LDTIAENQAK NEHLQKENER LLRDWNDVQG RFEKCVSAKE ...文字列: MERKISRIHL VSEPSITHFL QVSWEKTLES GFVITLTDGH SAWTGTVSES EISQEADDMA MEKGKYVGEL RKALLSGAGP ADVYTFNFSK ESCYFFFEKN LKDVSFRLGS FNLEKVENPA EVIRELICYC LDTIAENQAK NEHLQKENER LLRDWNDVQG RFEKCVSAKE ALETDLYKRF ILVLNEKKTK IRSLHNKLLN AAQEREKDIK QEGETAICSE MTADRDPVYD ESTDEESENQ TDLSGLASAA VSKDDSIISS LDVTDIAPSR KRRQRMQRNL GTEPKMAPQE NQLQEKENSR PDSSLPETSK KEHISAENMS LETLRNSSPE DLFDEI |
-分子 #2: NHEJ1_HUMAN Non-homologous end-joining factor 1
| 分子 | 名称: NHEJ1_HUMAN Non-homologous end-joining factor 1 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEELEQGLLM QPWAWLQLAE NSLLAKVFIT KQGYALLVSD LQQVWHEQVD TSVVSQRAKE LNKRLTAPPA AFLCHLDNLL RPLLKDAAHP SEATFSCDCV ADALILRVRS ELSGLPFYWN FHCMLASPSL VSQHLIRPLM GMSLALQCQV RELATLLHMK DLEIQDYQES ...文字列: MEELEQGLLM QPWAWLQLAE NSLLAKVFIT KQGYALLVSD LQQVWHEQVD TSVVSQRAKE LNKRLTAPPA AFLCHLDNLL RPLLKDAAHP SEATFSCDCV ADALILRVRS ELSGLPFYWN FHCMLASPSL VSQHLIRPLM GMSLALQCQV RELATLLHMK DLEIQDYQES GATLIRDRLK TEPFEENSFL EQFMIEKLPE ACSIGDGKPF VMNLQDLYMA VTTQEVQVGQ KHQGAGDPHT SNSASLQGID SQCVNQPEQL VSSAPTLSAP EKESTGTSGP LQRPQLSKVK RKKPRGLFS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 302 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 11006 / 平均露光時間: 4.0 sec. / 平均電子線量: 65.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 4.0 µm / 最小 デフォーカス(補正後): 2.0 µm / 倍率(補正後): 60000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT 当てはまり具合の基準: Cross-correlation coefficient |
|---|
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)