+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the Dsl1 complex bound to SNAREs Sec20 and Use1 | |||||||||
 マップデータ マップデータ | Half map B | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Tether / SNARE / Complex / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Golgi to ER transport vesicle membrane / vesicle fusion with endoplasmic reticulum / ER-dependent peroxisome organization / RZZ complex / Dsl1/NZR complex / COPI-dependent Golgi-to-ER retrograde traffic / regulation of ER to Golgi vesicle-mediated transport / SNARE complex / SNAP receptor activity / retrograde vesicle-mediated transport, Golgi to endoplasmic reticulum ...Golgi to ER transport vesicle membrane / vesicle fusion with endoplasmic reticulum / ER-dependent peroxisome organization / RZZ complex / Dsl1/NZR complex / COPI-dependent Golgi-to-ER retrograde traffic / regulation of ER to Golgi vesicle-mediated transport / SNARE complex / SNAP receptor activity / retrograde vesicle-mediated transport, Golgi to endoplasmic reticulum / mitotic spindle assembly checkpoint signaling / cytoplasmic side of endoplasmic reticulum membrane / endoplasmic reticulum to Golgi vesicle-mediated transport / vesicle-mediated transport / autophagy / protein transport / nuclear envelope / endoplasmic reticulum membrane / endoplasmic reticulum / membrane / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||
 データ登録者 データ登録者 | DAmico KA / Jeffrey PD / Hughson FM | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structure of a membrane tethering complex incorporating multiple SNAREs. 著者: Kevin A DAmico / Abigail E Stanton / Jaden D Shirkey / Sophie M Travis / Philip D Jeffrey / Frederick M Hughson /  要旨: Most membrane fusion reactions in eukaryotic cells are mediated by multisubunit tethering complexes (MTCs) and SNARE proteins. MTCs are much larger than SNAREs and are thought to mediate the initial ...Most membrane fusion reactions in eukaryotic cells are mediated by multisubunit tethering complexes (MTCs) and SNARE proteins. MTCs are much larger than SNAREs and are thought to mediate the initial attachment of two membranes. Complementary SNAREs then form membrane-bridging complexes whose assembly draws the membranes together for fusion. Here we present a cryo-electron microscopy structure of the simplest known MTC, the 255-kDa Dsl1 complex of Saccharomyces cerevisiae, bound to the two SNAREs that anchor it to the endoplasmic reticulum. N-terminal domains of the SNAREs form an integral part of the structure, stabilizing a Dsl1 complex configuration with unexpected similarities to the 850-kDa exocyst MTC. The structure of the SNARE-anchored Dsl1 complex and its comparison with exocyst reveal what are likely to be common principles underlying MTC function. Our structure also implies that tethers and SNAREs can work together as a single integrated machine. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28204.map.gz emd_28204.map.gz | 200 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28204-v30.xml emd-28204-v30.xml emd-28204.xml emd-28204.xml | 27.2 KB 27.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28204_fsc.xml emd_28204_fsc.xml emd_28204_fsc_2.xml emd_28204_fsc_2.xml emd_28204_fsc_3.xml emd_28204_fsc_3.xml emd_28204_fsc_4.xml emd_28204_fsc_4.xml | 13.4 KB 13.4 KB 13.4 KB 13.3 KB | 表示 表示 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28204.png emd_28204.png | 57.4 KB | ||
| Filedesc metadata |  emd-28204.cif.gz emd-28204.cif.gz | 8.3 KB | ||
| その他 |  emd_28204_half_map_1.map.gz emd_28204_half_map_1.map.gz emd_28204_half_map_2.map.gz emd_28204_half_map_2.map.gz | 198.5 MB 198.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28204 http://ftp.pdbj.org/pub/emdb/structures/EMD-28204 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28204 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28204 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28204_validation.pdf.gz emd_28204_validation.pdf.gz | 986.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28204_full_validation.pdf.gz emd_28204_full_validation.pdf.gz | 985.8 KB | 表示 | |
| XML形式データ |  emd_28204_validation.xml.gz emd_28204_validation.xml.gz | 22.3 KB | 表示 | |
| CIF形式データ |  emd_28204_validation.cif.gz emd_28204_validation.cif.gz | 28.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28204 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28204 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28204 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28204 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ekiMC  8ftuC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28204.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28204.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.114 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map A
| ファイル | emd_28204_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
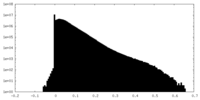
| 投影像・断面図 |
| ||||||||||||
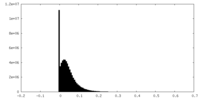
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_28204_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
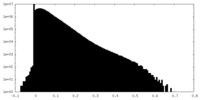
| 投影像・断面図 |
| ||||||||||||
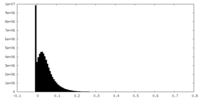
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dsl1 complex bound to SNARE proteins Sec20 and Use1
| 全体 | 名称: Dsl1 complex bound to SNARE proteins Sec20 and Use1 |
|---|---|
| 要素 |
|
-超分子 #1: Dsl1 complex bound to SNARE proteins Sec20 and Use1
| 超分子 | 名称: Dsl1 complex bound to SNARE proteins Sec20 and Use1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 255.41261 KDa |
-超分子 #2: Dsl1 Complex
| 超分子 | 名称: Dsl1 Complex / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #3-#5 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Protein transport protein SEC20
| 分子 | 名称: Protein transport protein SEC20 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 32.244254 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVVTFLQDLE VLQDALLNNL QKLSAISRRK ESGESKHDNK DSFAAIANEH NDEEEEIEFE DLVNIIESKV SDFESVLKCS IVEMTYKYP ELKLQWEKSP RYDQCDKLHI VKLDKQMNED IYAQLVEELD FVLQFVDWFY CYRLKVKEIL RQHHKRDLAW N DEKRDRAI ...文字列: MVVTFLQDLE VLQDALLNNL QKLSAISRRK ESGESKHDNK DSFAAIANEH NDEEEEIEFE DLVNIIESKV SDFESVLKCS IVEMTYKYP ELKLQWEKSP RYDQCDKLHI VKLDKQMNED IYAQLVEELD FVLQFVDWFY CYRLKVKEIL RQHHKRDLAW N DEKRDRAI KFHAVDYDKL HQGTSSSSSL TSTSMEKAST REKLLSKTKQ LTNNLVRGNQ ILQSGILQSD LNLDELRAQT NS LTQIDDK YTQFETVFKK TADLVKVLEN ASHQEKRD UniProtKB: Protein transport protein SEC20 |
-分子 #2: Protein transport protein USE1
| 分子 | 名称: Protein transport protein USE1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 24.254781 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAETSNDPFL SYVLSSKQLT NLNRLRRKAV TKQLGSSDDN KVSEEFLRYQ HTYQREAFEY LQTKHDAHKI MESQYEQYQS SSKTRRYSI DLDSVDAVDT ESQTEYPNEE FIDRNEDSEA VMELRKRLLG KGQNKGLGYE TTKSVDRQIE DQDTLQQDLI Q DMSKLVGS ...文字列: MAETSNDPFL SYVLSSKQLT NLNRLRRKAV TKQLGSSDDN KVSEEFLRYQ HTYQREAFEY LQTKHDAHKI MESQYEQYQS SSKTRRYSI DLDSVDAVDT ESQTEYPNEE FIDRNEDSEA VMELRKRLLG KGQNKGLGYE TTKSVDRQIE DQDTLQQDLI Q DMSKLVGS LKQGAVAFQS ALDEDKQVLG AAEIGIQVAS QGLMDVSGKL RKYD UniProtKB: Protein transport protein USE1 |
-分子 #3: Protein transport protein TIP20
| 分子 | 名称: Protein transport protein TIP20 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 81.253062 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNGIDDLLNI NDRIKQVQNE RNELASKLQN LKQSLASNDT EVALSEVIAQ DIIEVGASVE GLEQLRAKYG DLQILNKLEK VAVQQTQMQ AGVDKLDSFE RQLDELAEQP PDQFTLDDVK ALHSKLTSVF ATVPQINNID SQYAAYNKLK SKVTGKYNDV I IQRLATNW ...文字列: MNGIDDLLNI NDRIKQVQNE RNELASKLQN LKQSLASNDT EVALSEVIAQ DIIEVGASVE GLEQLRAKYG DLQILNKLEK VAVQQTQMQ AGVDKLDSFE RQLDELAEQP PDQFTLDDVK ALHSKLTSVF ATVPQINNID SQYAAYNKLK SKVTGKYNDV I IQRLATNW SNTFDQKLLE AQWDTQKFAS TSVGLVKCLR ENSTKLYQLS LLYLPLEEET QNGDSERPLS RSNNNQEPVL WN FKSLANN FNVRFTYHFH ATSSSSKIET YFQFLNDYLA ENLYKCINIF HDDCNGLTKP VIHEQFINYV LQPIRDKVRS TLF QNDLKT LIVLISQILA TDKNLLNSFH YHGLGLVSLI SDEVWEKWIN YEVEMANRQF INITKNPEDF PKSSQNFVKL INKI YDYLE PFYDLDFDLL VRYKLMTCSL IFMNLTSSYL DYILTVDSLN ETRTKEQELY QTMAKLQHVN FVYRKIKSLS SNFIF IQLT DIVNSTESKK YNSLFQNVEN DYEKAMSTDM QNSIVHRIQK LLKETLRNYF KISTWSTLEM SVDENIGPSS VPSAEL VNS INVLRRLINK LDSMDIPLAI SLKVKNELLN VIVNYFTESI LKLNKFNQNG LNQFLHDFKS LSSILSLPSH ATNYKCM SL HELVKILKLK YDPNNQQFLN PEYIKTGNFT SLKEAYSIKY LKDTKIQDAL YRIIYGNIL UniProtKB: Protein transport protein TIP20 |
-分子 #4: Protein transport protein SEC39
| 分子 | 名称: Protein transport protein SEC39 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 82.483797 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLEEQLYLLA CIFASRADTR NIKKLSTRLG SQSKYLEILC VLWPELDDPK NLLFLRELEE EVQSPEGEET TDEDVIVELL ESDSSLIPL IESDTTTRSN RYHELQEFIS KKLNNKTLEN FEEWLRERIL ICNEMIPETP LLYSVLWETA KSKVLSTKFI G WVEGVLKP ...文字列: MLEEQLYLLA CIFASRADTR NIKKLSTRLG SQSKYLEILC VLWPELDDPK NLLFLRELEE EVQSPEGEET TDEDVIVELL ESDSSLIPL IESDTTTRSN RYHELQEFIS KKLNNKTLEN FEEWLRERIL ICNEMIPETP LLYSVLWETA KSKVLSTKFI G WVEGVLKP LDHLNKRLHL IFKINEWEKM PDSELFKIIF DGVEDMQGYI GIADVIEDEL APTLSYGKKW ETFITEFFNK QQ FSLKSDT NYQLFIKLYY SLEKGVKDNS EASRKLQSNV VDILFHNSEN LFNLSSLTHK LDELWSILSG FPDEITIEEQ KTI TALEMK QFMEFFIKCS TKFSFKEIFA ITQEEESAQL AHFSSLCHEE FNKANEISSF LQAMYETVLD ISKDDKIFTR ISMD EKLYS ILEILLQMNE FAYIEAIIER FDYSNNTQIY ELLVKFFWHF FNNASNGLRK EPEMKKASQT LQIIQKHMSQ RAGTN LTKL EVLLEISDKL SHYSINLNKS HNGARDTAFK PSNILEYRDC PLDIISNLLE LNPRLYKDLP TTKSLLFGIY DSLSIN REG QTGKVEVDLM VLHIDYALVN LDFGTAYELG KQVFEICQEA GQHMMKALGD EHWLTFYQMG KFVDPNWVDN EIPTEII VL QMSILGRLLE VCPLEEVEIV TSQWSTLELE LSARDLVKDK YALDGQNDNK SKVGGIAREI FHNVTNF UniProtKB: Protein transport protein SEC39 |
-分子 #5: Protein transport protein DSL1
| 分子 | 名称: Protein transport protein DSL1 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 91.445133 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKHHHHHHHG AAGTSLYKKA GENLYFQGSM ESLFPNKGEI IRELLKDPLI LKNDSKRSNG SELELDSSDL LQREAILANE LNILDNLKT FLNLIKEVKT NLNILELENC YYSLQSLRKK MRNNAAYLKQ SFNFQQSIST YVDTLHLELV STLYKILTNG F WKITENSI ...文字列: MKHHHHHHHG AAGTSLYKKA GENLYFQGSM ESLFPNKGEI IRELLKDPLI LKNDSKRSNG SELELDSSDL LQREAILANE LNILDNLKT FLNLIKEVKT NLNILELENC YYSLQSLRKK MRNNAAYLKQ SFNFQQSIST YVDTLHLELV STLYKILTNG F WKITENSI QFTPTVEWGK DKVHIEYDTF MDFVAQQYFP KGSLDNQAWF ILDMTSADSQ EQVRAKLNTI MKEYMNLSRI VS MIKNSIF ISGKEISYEN EKNILVFSKS SSHGQHCVST VLTSFEAVCD FMLDGLAFRD RKTLSYELGP LFNTEFTKFV KNN ASIILE SLDSPLKNLV SVINNKLTRL VAKSEVTNWT HSGKEIQDLL MNKQLYYNLL LDKVLESHIS EIRSIFEDPK KSWQ NLEVV ELTTSNTNTM SEKIGKNDSD VQNEKELHNA VSKDDDWNWE VEDDDADAWG DEIDVNIDDE EEKTNQEKEK EPEEE ENAW DEAWAIDENI DDASLENGKE HLKAHDVGSL DKDHIEVTQL PKLFLAISQN FKSSFADSHV DEQYFAYKYN LLQTSY MAM CTANFSHNWC QLYVDMRYLI ERDEKLYRIK ELTRNLLETK LNMKYRIVCQ LIRHQLTEFR ENERNPSWDA TIEKLLP YI LKEIVRPLQK IRGEEGSRYL LSFLNFLYND CVTKEILKWQ IISEVNSENL GELVSLLVNN TDIQLLAKEP SYKKMREK F ATMGKFLPLH LKEIMEMFYN GDFYLFATDE LIQWIELLFA DTPLRRNAID DIYEIRGTAL DD UniProtKB: Protein transport protein DSL1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Buffer was made fresh from concentrated components and sterile filtered. NP40 was not present during protein purification but was an additive during the grid preparation. | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 10 | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV / 詳細: Force=0 Wait Time=0 Blot Time=6s Drain Time=0. | |||||||||||||||
| 詳細 | Sample was consistently in the thickest regions of ice only, often close to the edges of the carbon hole |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 5857 / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.25 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X