+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
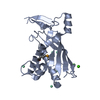
| タイトル | Staphylococcus epidermidis RP62a CRISPR effector subcomplex | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Type IIIA CRISPR / effector complex / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA complex | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報exonuclease activity / defense response to virus / endonuclease activity / RNA binding / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.55 Å | |||||||||||||||
 データ登録者 データ登録者 | Smith EM / Ferrell SH / Tokars VL / Mondragon A | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Structures of an active type III-A CRISPR effector complex. 著者: Eric M Smith / Sé Ferrell / Valerie L Tokars / Alfonso Mondragón /  要旨: Clustered regularly interspaced short palindromic repeats (CRISPR) and their CRISPR-associated proteins (Cas) provide many prokaryotes with an adaptive immune system against invading genetic material. ...Clustered regularly interspaced short palindromic repeats (CRISPR) and their CRISPR-associated proteins (Cas) provide many prokaryotes with an adaptive immune system against invading genetic material. Type III CRISPR systems are unique in that they can degrade both RNA and DNA. In response to invading nucleic acids, they produce cyclic oligoadenylates that act as secondary messengers, activating cellular nucleases that aid in the immune response. Here, we present seven single-particle cryo-EM structures of the type III-A Staphylococcus epidermidis CRISPR effector complex. The structures reveal the intact S. epidermidis effector complex in an apo, ATP-bound, cognate target RNA-bound, and non-cognate target RNA-bound states and illustrate how the effector complex binds and presents crRNA. The complexes bound to target RNA capture the type III-A effector complex in a post-RNA cleavage state. The ATP-bound structures give details about how ATP binds to Cas10 to facilitate cyclic oligoadenylate production. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26920.map.gz emd_26920.map.gz | 141.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26920-v30.xml emd-26920-v30.xml emd-26920.xml emd-26920.xml | 23.3 KB 23.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26920_fsc.xml emd_26920_fsc.xml | 14.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26920.png emd_26920.png | 64.3 KB | ||
| マスクデータ |  emd_26920_msk_1.map emd_26920_msk_1.map | 282.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-26920.cif.gz emd-26920.cif.gz | 7 KB | ||
| その他 |  emd_26920_half_map_1.map.gz emd_26920_half_map_1.map.gz emd_26920_half_map_2.map.gz emd_26920_half_map_2.map.gz | 262.5 MB 262.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26920 http://ftp.pdbj.org/pub/emdb/structures/EMD-26920 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26920 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26920 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26920_validation.pdf.gz emd_26920_validation.pdf.gz | 992.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26920_full_validation.pdf.gz emd_26920_full_validation.pdf.gz | 992.4 KB | 表示 | |
| XML形式データ |  emd_26920_validation.xml.gz emd_26920_validation.xml.gz | 23.1 KB | 表示 | |
| CIF形式データ |  emd_26920_validation.cif.gz emd_26920_validation.cif.gz | 29.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26920 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26920 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26920 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26920 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7uzwMC  7uzxC  7uzyC  7uzzC  7v00C  7v01C  7v02C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26920.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26920.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26920_msk_1.map emd_26920_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_26920_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26920_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Staphylococcus epidermidis RP62a CRISPR effector subcomplex
| 全体 | 名称: Staphylococcus epidermidis RP62a CRISPR effector subcomplex |
|---|---|
| 要素 |
|
-超分子 #1: Staphylococcus epidermidis RP62a CRISPR effector subcomplex
| 超分子 | 名称: Staphylococcus epidermidis RP62a CRISPR effector subcomplex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: RP62a |
-分子 #1: CRISPR system Cms endoribonuclease Csm3
| 分子 | 名称: CRISPR system Cms endoribonuclease Csm3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A |
| 分子量 | 理論値: 24.033975 KDa |
| 配列 | 文字列: MYSKIKISGT IEVVTGLHIG GGGESSMIGA IDSPVVRDLQ TKLPIIPGSS IKGKMRNLLA KHFGLKMKQE SHNQDDERVL RLFGSSEKG NIQRARLQIS DAFFSEKTKE HFAQNDIAYT ETKFENTINR LTAVANPRQI ERVTRGSEFD FVFIYNVDEE S QVEDDFEN ...文字列: MYSKIKISGT IEVVTGLHIG GGGESSMIGA IDSPVVRDLQ TKLPIIPGSS IKGKMRNLLA KHFGLKMKQE SHNQDDERVL RLFGSSEKG NIQRARLQIS DAFFSEKTKE HFAQNDIAYT ETKFENTINR LTAVANPRQI ERVTRGSEFD FVFIYNVDEE S QVEDDFEN IEKAIHLLEN DYLGGGGTRG NGRIQFKDTN IETVVGEYDS TNLKIK UniProtKB: CRISPR system Cms endoribonuclease Csm3 |
-分子 #2: CRISPR system Cms protein Csm4
| 分子 | 名称: CRISPR system Cms protein Csm4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A |
| 分子量 | 理論値: 34.551938 KDa |
| 配列 | 文字列: MTLATKVFKL SFKTPVHFGK KRLSDGEMTI TADTLFSALF IETLQLGKDT DWLLNDLIIS DTFPYENELY YLPKPLIKID SKEEDNHKA FKKLKYVPVH HYNQYLNGEL SAEDATDLND IFNIGYFSLQ TKVSLIAQET DSSADSEPYS VGTFTFEPEA G LYFIAKGS ...文字列: MTLATKVFKL SFKTPVHFGK KRLSDGEMTI TADTLFSALF IETLQLGKDT DWLLNDLIIS DTFPYENELY YLPKPLIKID SKEEDNHKA FKKLKYVPVH HYNQYLNGEL SAEDATDLND IFNIGYFSLQ TKVSLIAQET DSSADSEPYS VGTFTFEPEA G LYFIAKGS EETLDHLNNI MTALQYSGLG GKRNAGYGQF EYEIINNQQL SKLLNQNGKH SILLSTAMAK KEEIESALKE AR YILTKRS GFVQSTNYSE MLVKKSDFYS FSSGSVFKNI FNGDIFNVGH NGKHPVYRYA KPLWLEV UniProtKB: CRISPR system Cms protein Csm4 |
-分子 #3: CRISPR system single-strand-specific deoxyribonuclease Cas10/Csm1...
| 分子 | 名称: CRISPR system single-strand-specific deoxyribonuclease Cas10/Csm1 (subtype III-A) タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A |
| 分子量 | 理論値: 87.685781 KDa |
| 配列 | 文字列: MNKKNILMYG SLLHDIGKII YRSGDHTFSR GTHSKLGHQF LSQFSEFKDN EVLDNVAYHH YKELAKANLD NDNTAYITYI ADNIASGID RRDIIEEGDE EYEKQLFNFD KYTPLYSVFN IVNSEKLKQT NGKFKFSNES NIEYPKTENI QYSSGNYTTL M KDMSHDLE ...文字列: MNKKNILMYG SLLHDIGKII YRSGDHTFSR GTHSKLGHQF LSQFSEFKDN EVLDNVAYHH YKELAKANLD NDNTAYITYI ADNIASGID RRDIIEEGDE EYEKQLFNFD KYTPLYSVFN IVNSEKLKQT NGKFKFSNES NIEYPKTENI QYSSGNYTTL M KDMSHDLE HKLSIKEGTF PSLLQWTESL WQYVPSSTNK NQLIDISLYD HSRITCAIAS CIFDYLNENN IHNYKDELFS KY ENTKSFY QKEAFLLLSM DMSGIQDFIY NISGSKALKS LRSRSFYLEL MLEVIVDQLL ERLELARANL LYTGGGHAYL LVS NTDKVK KKITQFNNEL KKWFMSEFTT DLSLSMAFEK CSGDDLMNTS GNYRTIWRNV SSKLSDIKAH KYSAEDILKL NHFH SYGDR ECKECLRSDI DINDDGLCSI CEGIINISND LRDKSFFVLS ETGKLKMPFN KFISVIDYEE AEMLVQNNNQ VRIYS KNKP YIGIGISTNL WMCDYDYASQ NQDMREKGIG SYVDREEGVK RLGVVRADID NLGATFISGI PEKYNSISRT ATLSRQ LSL FFKYELNHLL ENYQITAIYS GGDDLFLIGA WDDIIEASIY INDKFKEFTL DKLTLSAGVG MFSGKYPVSK MAFETGR LE EAAKTGEKNQ ISLWLQEKVY NWDEFKKNIL EEKLLVLQQG FSQTDEHGKA FIYKMLALLR NNEAINIARL AYLLARSK M NEDFTSKIFN WAQNDKDKNQ LITALEYYIY QIREAD UniProtKB: CRISPR system single-strand-specific deoxyribonuclease Cas10/Csm1 (subtype III-A) |
-分子 #4: crRNA
| 分子 | 名称: crRNA / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A |
| 分子量 | 理論値: 13.862377 KDa |
| 配列 | 文字列: ACGAGAACAC GUAUGCCGAA GUAUAUAAAU CAUCAGUACA AAG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. 詳細: cryo-EM grids were prepared by glow discharging for 10 seconds at 15 mA in a Pelco easiGlow glow discharger. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: 3 ul of the protein-RNA complex were added to the grid and allowed to incubate for 10 seconds. After incubation the grid was blotted in a Vitrobot Mark IV (FEI Thermo Fischer) (humidity 95% ...詳細: 3 ul of the protein-RNA complex were added to the grid and allowed to incubate for 10 seconds. After incubation the grid was blotted in a Vitrobot Mark IV (FEI Thermo Fischer) (humidity 95% and temperature 4 C) for 3 seconds with a force of 0 before plunge freezing in liquid ethane.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 2455 / 平均電子線量: 49.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 44454 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER | ||||||||||||
| 得られたモデル |  PDB-7uzw: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)