+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22431 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human HUWE1 (non-crosslinked) | ||||||||||||||||||
 マップデータ マップデータ | Cryo-EM structure of human HUWE1 (non-crosslinked) | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of mitochondrial fusion / positive regulation of mitophagy in response to mitochondrial depolarization / histone ubiquitin ligase activity / HECT-type E3 ubiquitin transferase / positive regulation of protein targeting to mitochondrion / Golgi organization / protein monoubiquitination / positive regulation of protein ubiquitination / circadian regulation of gene expression / base-excision repair ...negative regulation of mitochondrial fusion / positive regulation of mitophagy in response to mitochondrial depolarization / histone ubiquitin ligase activity / HECT-type E3 ubiquitin transferase / positive regulation of protein targeting to mitochondrion / Golgi organization / protein monoubiquitination / positive regulation of protein ubiquitination / circadian regulation of gene expression / base-excision repair / protein polyubiquitination / ubiquitin-protein transferase activity / ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / ubiquitin-dependent protein catabolic process / secretory granule lumen / ficolin-1-rich granule lumen / membrane fusion / cell differentiation / Golgi membrane / Neutrophil degranulation / mitochondrion / DNA binding / RNA binding / extracellular exosome / extracellular region / nucleoplasm / membrane / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Hunkeler M / Fischer ES | ||||||||||||||||||
| 資金援助 |  米国, 米国,  スイス, 5件 スイス, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2021 ジャーナル: Mol Cell / 年: 2021タイトル: Solenoid architecture of HUWE1 contributes to ligase activity and substrate recognition. 著者: Moritz Hunkeler / Cyrus Y Jin / Michelle W Ma / Julie K Monda / Daan Overwijn / Eric J Bennett / Eric S Fischer /  要旨: HECT ubiquitin ligases play essential roles in metazoan development and physiology. The HECT ligase HUWE1 is central to the cellular stress response by mediating degradation of key death or survival ...HECT ubiquitin ligases play essential roles in metazoan development and physiology. The HECT ligase HUWE1 is central to the cellular stress response by mediating degradation of key death or survival factors, including Mcl1, p53, DDIT4, and Myc. Although mutations in HUWE1 and related HECT ligases are widely implicated in human disease, our molecular understanding remains limited. Here we present a comprehensive investigation of full-length HUWE1, deepening our understanding of this class of enzymes. The N-terminal ∼3,900 amino acids of HUWE1 are indispensable for proper ligase function, and our cryo-EM structures of HUWE1 offer a complete molecular picture of this large HECT ubiquitin ligase. HUWE1 forms an alpha solenoid-shaped assembly with a central pore decorated with protein interaction modules. Structures of HUWE1 variants linked to neurodevelopmental disorders as well as of HUWE1 bound to a model substrate link the functions of this essential enzyme to its three-dimensional organization. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22431.map.gz emd_22431.map.gz | 86 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22431-v30.xml emd-22431-v30.xml emd-22431.xml emd-22431.xml | 27.7 KB 27.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22431_fsc.xml emd_22431_fsc.xml | 10.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22431.png emd_22431.png | 42.4 KB | ||
| マスクデータ |  emd_22431_msk_1.map emd_22431_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_22431_additional_1.map.gz emd_22431_additional_1.map.gz emd_22431_half_map_1.map.gz emd_22431_half_map_1.map.gz emd_22431_half_map_2.map.gz emd_22431_half_map_2.map.gz | 47 MB 84.7 MB 84.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22431 http://ftp.pdbj.org/pub/emdb/structures/EMD-22431 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22431 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22431 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22431_validation.pdf.gz emd_22431_validation.pdf.gz | 558.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22431_full_validation.pdf.gz emd_22431_full_validation.pdf.gz | 557.8 KB | 表示 | |
| XML形式データ |  emd_22431_validation.xml.gz emd_22431_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  emd_22431_validation.cif.gz emd_22431_validation.cif.gz | 23 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22431 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22431 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22431 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22431 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22431.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22431.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of human HUWE1 (non-crosslinked) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0588 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22431_msk_1.map emd_22431_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
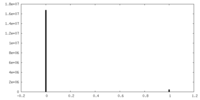
| 投影像・断面図 |
| ||||||||||||
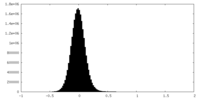
| 密度ヒストグラム |
-追加マップ: main map blurred to B=10A2
| ファイル | emd_22431_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map blurred to B=10A2 | ||||||||||||
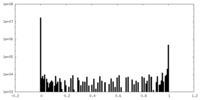
| 投影像・断面図 |
| ||||||||||||
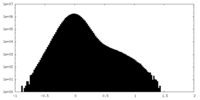
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_22431_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
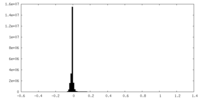
| 投影像・断面図 |
| ||||||||||||
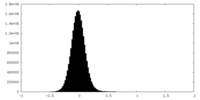
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_22431_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
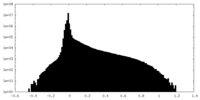
| 投影像・断面図 |
| ||||||||||||
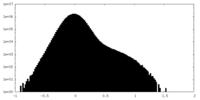
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : E3 ubiquitin-protein ligase HUWE1
| 全体 | 名称: E3 ubiquitin-protein ligase HUWE1 |
|---|---|
| 要素 |
|
-超分子 #1: E3 ubiquitin-protein ligase HUWE1
| 超分子 | 名称: E3 ubiquitin-protein ligase HUWE1 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: full length, non-crosslinked |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 480 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換株: Expi293 / 組換プラスミド: pDEST Homo sapiens (ヒト) / 組換株: Expi293 / 組換プラスミド: pDEST |
-分子 #1: E3 ubiquitin-protein ligase HUWE1
| 分子 | 名称: E3 ubiquitin-protein ligase HUWE1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: HECT-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDYKDDDDKL AAANSSIDLI STSLYKKAGF KGTNSVDMKV DRTKLKKTPT EAPADCRALI DKLKVCNDEQ LLLELQQIKT WNIGKCELYH WVDLLDRFDG ILADAGQTVE NMSWMLVCDR PEREQLKMLL LAVLNFTALL IEYSFSRHLY SSIEHLTTLL ASSDMQVVLA ...文字列: MDYKDDDDKL AAANSSIDLI STSLYKKAGF KGTNSVDMKV DRTKLKKTPT EAPADCRALI DKLKVCNDEQ LLLELQQIKT WNIGKCELYH WVDLLDRFDG ILADAGQTVE NMSWMLVCDR PEREQLKMLL LAVLNFTALL IEYSFSRHLY SSIEHLTTLL ASSDMQVVLA VLNLLYVFSK RSNYITRLGS DKRTPLLTRL QHLAESWGGK ENGFGLAECC RDLHMMKYPP SATTLHFEFY ADPGAEVKIE KRTTSNTLHY IHIEQLDKIS ESPSEIMESL TKMYSIPKDK QMLLFTHIRL AHGFSNHRKR LQAVQARLHA ISILVYSNAL QESANSILYN GLIEELVDVL QITDKQLMEI KAASLRTLTS IVHLERTPKL SSIIDCTGTA SYHGFLPVLV RNCIQAMIDP SMDPYPHQFA TALFSFLYHL ASYDAGGEAL VSCGMMEALL KVIKFLGDEQ DQITFVTRAV RVVDLITNLD MAAFQSHSGL SIFIYRLEHE VDLCRKECPF VIKPKIQRPN TTQEGEEMET DMDGVQCIPQ RAALLKSMLN FLKKAIQDPA FSDGIRHVMD GSLPTSLKHI ISNAEYYGPS LFLLATEVVT VFVFQEPSLL SSLQDNGLTD VMLHALLIKD VPATREVLGS LPNVFSALCL NARGLQSFVQ CQPFERLFKV LLSPDYLPAM RRRRSSDPLG DTASNLGSAV DELMRHQPTL KTDATTAIIK LLEEICNLGR DPKYICQKPS IQKADGTATA PPPRSNHAAE EASSEDEEEE EVQAMQSFNS TQQNETEPNQ QVVGTEERIP IPLMDYILNV MKFVESILSN NTTDDHCQEF VNQKGLLPLV TILGLPNLPI DFPTSAACQA VAGVCKSILT LSHEPKVLQE GLLQLDSILS SLEPLHRPIE SPGGSVLLRE LACAGNVADA TLSAQATPLL HALTAAHAYI MMFVHTCRVG QSEIRSISVN QWGSQLGLSV LSKLSQLYCS LVWESTVLLS LCTPNSLPSG CEFGQADMQK LVPKDEKAGT TQGGKRSDGE QDGAAGSMDA STQGLLEGIG LDGDTLAPME TDEPTASDSK GKSKITPAMA ARIKQIKPLL SASSRLGRAL AELFGLLVKL CVGSPVRQRR SHHAASTTTA PTPAARSTAS ALTKLLTKGL SWQPPPYTPT PRFRLTFFIC SVGFTSPMLF DERKYPYHLM LQKFLCSGGH NALFETFNWA LSMGGKVPVS EGLEHSDLPD GTGEFLDAWL MLVEKMVNPT TVLESPHSLP AKLPGGVQNF PQFSALRFLV VTQKAAFTCI KNLWNRKPLK VYGGRMAESM LAILCHILRG EPVIRERLSK EKEGSRGEED TGQEEGGSRR EPQVNQQQLQ QLMDMGFTRE HAMEALLNTS TMEQATEYLL THPPPIMGGV VRDLSMSEED QMMRAIAMSL GQDIPMDQRA ESPEEVACRK EEEERKAREK QEEEEAKCLE KFQDADPLEQ DELHTFTDTM LPGCFHLLDE LPDTVYRVCD LIMTAIKRNG ADYRDMILKQ VVNQVWEAAD VLIKAALPLT TSDTKTVSEW ISQMATLPQA SNLATRILLL TLLFEELKLP CAWVVESSGI LNVLIKLLEV VQPCLQAAKE QKEVQTPKWI TPVLLLIDFY EKTAISSKRR AQMTKYLQSN SNNWRWFDDR SGRWCSYSAS NNSTIDSAWK SGETSVRFTA GRRRYTVQFT TMVQVNEETG NRRPVMLTLL RVPRLNKNSK NSNGQELEKT LEESKEMDIK RKENKGNDTP LALESTNTEK ETSLEETKIG EILIQGLTED MVTVLIRACV SMLGVPVDPD TLHATLRLCL RLTRDHKYAM MFAELKSTRM ILNLTQSSGF NGFTPLVTLL LRHIIEDPCT LRHTMEKVVR SAATSGAGST TSGVVSGSLG SREINYILRV LGPAACRNPD IFTEVANCCI RIALPAPRGS GTASDDEFEN LRIKGPNAVQ LVKTTPLKPS PLPVIPDTIK EVIYDMLNAL AAYHAPEEAD KSDPKPGVMT QEVGQLLQDM GDDVYQQYRS LTRQSSDFDT QSGFSINSQV FAADGASTET SASGTSQGEA STPEESRDGK KDKEGDRASE EGKQKGKGSK PLMPTSTILR LLAELVRSYV GIATLIANYS YTVGQSELIK EDCSVLAFVL DHLLPHTQNA EDKDTPALAR LFLASLAAAG SGTDAQVALV NEVKAALGRA LAMAESTEKH ARLQAVMCII STIMESCPST SSFYSSATAK TQHNGMNNII RLFLKKGLVN DLARVPHSLD LSSPNMANTV NAALKPLETL SRIVNQPSSL FGSKSASSKN KSEQDAQGAS QDSSSNQQDP GEPGEAEVQE EDHDVTQTEV ADGDIMDGEA ETDSVVIAGQ PEVLSSQEMQ VENELEDLID ELLERDGGSG NSTIIVSRSG EDESQEDVLM DEAPSNLSQA STLQANREDS MNILDPEDEE EHTQEEDSSG SNEDEDDSQD EEEEEEEDEE DDQEDDEGEE GDEDDDDDGS EMELDEDYPD MNASPLVRFE RFDREDDLII EFDNMFSSAT DIPPSPGNIP TTHPLMVRHA DHSSLTLGSG SSTTRLTQGI GRSQRTLRQL TANTGHTIHV HYPGNRQPNP PLILQRLLGP SAAADILQLS SSLPLQSRGR ARLLVGNDDV HIIARSDDEL LDDFFHDQST ATSQAGTLSS IPTALTRWTE ECKVLDAESM HDCVSVVKVS IVNHLEFLRD EELEERREKR RKQLAEEETK ITDKGKEDKE NRDQSAQCTA SKSNDSTEQN LSDGTPMPDS YPTTPSSTDA ATSESKETLG TLQSSQQQPT LPTPPALGEV PQELQSPAGE GGSSTQLLMP VEPEELGPTR PSGEAETTQM ELSPAPTITS LSPERAEDSD ALTAVSSQLE GSPMDTSSLA SCTLEEAVGD TSAAGSSEQP RAGSSTPGDA PPAVAEVQGR SDGSGESAQP PEDSSPPASS ESSSTRDSAV AISGADSRGI LEEPLPSTSS EEEDPLAGIS LPEGVDPSFL AALPDDIRRE VLQNQLGIRP PTRTAPSTNS SAPAVVGNPG VTEVSPEFLA ALPPAIQEEV LAQQRAEQQR RELAQNASSD TPMDPVTFIQ TLPSDLRRSV LEDMEDSVLA VMPPDIAAEA QALRREQEAR QRQLMHERLF GHSSTSALSA ILRSPAFTSR LSGNRGVQYT RLAVQRGGTF QMGGSSSHNR PSGSNVDTLL RLRGRLLLDH EALSCLLVLL FVDEPKLNTS RLHRVLRNLC YHAQTRHWVI RSLLSILQRS SESELCIETP KLTTSEEKGK KSSKSCGSSS HENRPLDLLH KMESKSSNQL SWLSVSMDAA LGCRTNIFQI QRSGGRKHTE KHASGGSTVH IHPQAAPVVC RHVLDTLIQL AKVFPSHFTQ QRTKETNCES DRERGNKACS PCSSQSSSSG ICTDFWDLLV KLDNMNVSRK GKNSVKSVPV SAGGEGETSP YSLEASPLGQ LMNMLSHPVI RRSSLLTEKL LRLLSLISIA LPENKVSEAQ ANSGSGASST TTATSTTSTT TTTAASTTPT PPTAPTPVTS APALVAATAI STIVVAASTT VTTPTTATTT VSISPTTKGS KSPAKVSDGG SSSTDFKMVS SGLTENQLQL SVEVLTSHSC SEEGLEDAAN VLLQLSRGDS GTRDTVLKLL LNGARHLGYT LCKQIGTLLA ELREYNLEQQ RRAQCETLSP DGLPEEQPQT TKLKGKMQSR FDMAENVVIV ASQKRPLGGR ELQLPSMSML TSKTSTQKFF LRVLQVIIQL RDDTRRANKK AKQTGRLGSS GLGSASSIQA AVRQLEAEAD AIIQMVREGQ RARRQQQAAT SESSQSEASV RREESPMDVD QPSPSAQDTQ SIASDGTPQG EKEKEERPPE LPLLSEQLSL DELWDMLGEC LKELEESHDQ HAVLVLQPAV EAFFLVHATE RESKPPVRDT RESQLAHIKD EPPPLSPAPL TPATPSSLDP FFSREPSSMH ISSSLPPDTQ KFLRFAETHR TVLNQILRQS TTHLADGPFA VLVDYIRVLD FDVKRKYFRQ ELERLDEGLR KEDMAVHVRR DHVFEDSYRE LHRKSPEEMK NRLYIVFEGE EGQDAGGLLR EWYMIISREM FNPMYALFRT SPGDRVTYTI NPSSHCNPNH LSYFKFVGRI VAKAVYDNRL LECYFTRSFY KHILGKSVRY TDMESEDYHF YQGLVYLLEN DVSTLGYDLT FSTEVQEFGV CEVRDLKPNG ANILVTEENK KEYVHLVCQM RMTGAIRKQL AAFLEGFYEI IPKRLISIFT EQELELLISG LPTIDIDDLK SNTEYHKYQS NSIQIQWFWR ALRSFDQADR AKFLQFVTGT SKVPLQGFAA LEGMNGIQKF QIHRDDRSTD RLPSAHTCFN QLDLPAYESF EKLRHMLLLA IQECSEGFGL A |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 12.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283 K / 装置: LEICA EM GP 詳細: CHAPSO detergent added to final conc. of 0.8 mM. Sample applied twice.. | |||||||||
| 詳細 | Monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | Data colleciton in counting mode, using multi-shot scheme (4 holes per stage position, 3 movies per hole) |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 撮影したグリッド数: 1 / 実像数: 9225 / 平均露光時間: 7.0 sec. / 平均電子線量: 49.37 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): -2.3000000000000003 µm 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X