+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22200 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the G protein-gated inward rectifier K+ channel GIRK2 (Kir3.2) in complex with PIP2 | |||||||||
 マップデータ マップデータ | Cryo-EM structure of the G protein-gated inward rectifier K+ channel GIRK2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | G protein-coupled inwardly rectifying potassium channels / PIP2 / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報G-protein activated inward rectifier potassium channel activity / Activation of G protein gated Potassium channels / Inhibition of voltage gated Ca2+ channels via Gbeta/gamma subunits / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / inward rectifier potassium channel activity / regulation of monoatomic ion transmembrane transport / parallel fiber to Purkinje cell synapse / monoatomic ion channel complex / neuronal cell body membrane / potassium ion import across plasma membrane ...G-protein activated inward rectifier potassium channel activity / Activation of G protein gated Potassium channels / Inhibition of voltage gated Ca2+ channels via Gbeta/gamma subunits / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / inward rectifier potassium channel activity / regulation of monoatomic ion transmembrane transport / parallel fiber to Purkinje cell synapse / monoatomic ion channel complex / neuronal cell body membrane / potassium ion import across plasma membrane / potassium channel activity / G-protein alpha-subunit binding / negative regulation of insulin secretion / presynapse / presynaptic membrane / postsynapse / axon / dendrite / cell surface / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Niu Y / Tao X | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Cryo-EM analysis of PIP regulation in mammalian GIRK channels. 著者: Yiming Niu / Xiao Tao / Kouki K Touhara / Roderick MacKinnon /  要旨: G-protein-gated inward rectifier potassium (GIRK) channels are regulated by G proteins and PIP. Here, using cryo-EM single particle analysis we describe the equilibrium ensemble of structures of ...G-protein-gated inward rectifier potassium (GIRK) channels are regulated by G proteins and PIP. Here, using cryo-EM single particle analysis we describe the equilibrium ensemble of structures of neuronal GIRK2 as a function of the C8-PIP concentration. We find that PIP shifts the equilibrium between two distinguishable structures of neuronal GIRK (GIRK2), extended and docked, towards the docked form. In the docked form the cytoplasmic domain, to which G binds, becomes accessible to the cytoplasmic membrane surface where G resides. Furthermore, PIP binding reshapes the G binding surface on the cytoplasmic domain, preparing it to receive G. We find that cardiac GIRK (GIRK1/4) can also exist in both extended and docked conformations. These findings lead us to conclude that PIP influences GIRK channels in a structurally similar manner to Kir2.2 channels. In Kir2.2 channels, the PIP-induced conformational changes open the pore. In GIRK channels, they prepare the channel for activation by G. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22200.map.gz emd_22200.map.gz | 229.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22200-v30.xml emd-22200-v30.xml emd-22200.xml emd-22200.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_22200.png emd_22200.png | 31.6 KB | ||
| Filedesc metadata |  emd-22200.cif.gz emd-22200.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22200 http://ftp.pdbj.org/pub/emdb/structures/EMD-22200 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22200 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22200 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22200_validation.pdf.gz emd_22200_validation.pdf.gz | 605.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22200_full_validation.pdf.gz emd_22200_full_validation.pdf.gz | 604.8 KB | 表示 | |
| XML形式データ |  emd_22200_validation.xml.gz emd_22200_validation.xml.gz | 5.7 KB | 表示 | |
| CIF形式データ |  emd_22200_validation.cif.gz emd_22200_validation.cif.gz | 6.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22200 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22200 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22200 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22200 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22200.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22200.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the G protein-gated inward rectifier K+ channel GIRK2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Homo-tetrameric assembly of G protein-activated inward rectifier ...
| 全体 | 名称: Homo-tetrameric assembly of G protein-activated inward rectifier potassium channel 2 |
|---|---|
| 要素 |
|
-超分子 #1: Homo-tetrameric assembly of G protein-activated inward rectifier ...
| 超分子 | 名称: Homo-tetrameric assembly of G protein-activated inward rectifier potassium channel 2 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: G protein-activated inward rectifier potassium channel 2
| 分子 | 名称: G protein-activated inward rectifier potassium channel 2 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 39.061957 KDa |
| 組換発現 | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: MAKRKIQRYV RKDGKCNVHH GNVRETYRYL TDIFTTLVDL KWRFNLLIFV MVYTVTWLFF GMIWWLIAYI RGDMDHIEDP SWTPCVTNL NGFVSAFLFS IETETTIGYG YRVITDKCPE GIILLLIQSV LGSIVNAFMV GCMFVKISQP KKRAETLVFS T HAVISMRD ...文字列: MAKRKIQRYV RKDGKCNVHH GNVRETYRYL TDIFTTLVDL KWRFNLLIFV MVYTVTWLFF GMIWWLIAYI RGDMDHIEDP SWTPCVTNL NGFVSAFLFS IETETTIGYG YRVITDKCPE GIILLLIQSV LGSIVNAFMV GCMFVKISQP KKRAETLVFS T HAVISMRD GKLCLMFRVG DLRNSHIVEA SIRAKLIKSK QTSEGEFIPL NQTDINVGYY TGDDRLFLVS PLIISHEINQ QS PFWEISK AQLPKEELEI VVILEGMVEA TGMTCQARSS YITSEILWGY RFTPVLTLED GFYEVDYNSF HETYETSTPS LSA KELAEL ANRAESNSLE VLFQ UniProtKB: G protein-activated inward rectifier potassium channel 2 |
-分子 #2: [(2R)-2-octanoyloxy-3-[oxidanyl-[(1R,2R,3S,4R,5R,6S)-2,3,6-tris(o...
| 分子 | 名称: [(2R)-2-octanoyloxy-3-[oxidanyl-[(1R,2R,3S,4R,5R,6S)-2,3,6-tris(oxidanyl)-4,5-diphosphonooxy-cyclohexyl]oxy-phosphoryl]oxy-propyl] octanoate タイプ: ligand / ID: 2 / コピー数: 4 / 式: PIO |
|---|---|
| 分子量 | 理論値: 746.566 Da |
| Chemical component information | 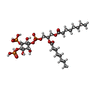 ChemComp-PIO: |
-分子 #3: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 3 / コピー数: 3 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM Tris-HCl pH 7.5, 150 mM KCl, 10 mM DTT, 1 mM DETA, 0.2% DM and 0.01% CHS |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 11349 / 平均露光時間: 10.0 sec. / 平均電子線量: 8.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















