+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2203 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Characterization of the insertase for beta-barrel proteins of the outer mitochondrial membrane. 3-D reconstruction of the TOB complex | |||||||||
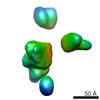
 マップデータ マップデータ | 3D reconstruction of Tob55 dimers isolated using the 9xHis tag on the Tob55 subunit. The Tob55 dimer is a complex identified in all outer membrane preparations but isolated only when the his-tag is on tob55. The dimer co-purifies with the TOB complex and was identified as a subclass of particles present in the his-Tob55 data sets. The mol. wgt. is 101 kDa. Two-fold symmetry has been applied | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TOB/SAM complex / beta-barrel proteins / Tob55/Sam50 / mitochondria outer membrane | |||||||||
| 生物種 |  Neurospora crassa (菌類) Neurospora crassa (菌類) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 13.4 Å | |||||||||
 データ登録者 データ登録者 | Klein A / Israel L / Lackey SWK / Nargang FE / Imhof A / Baumeister W / Neupert W / Thomas DR | |||||||||
 引用 引用 |  ジャーナル: J Cell Biol / 年: 2012 ジャーナル: J Cell Biol / 年: 2012タイトル: Characterization of the insertase for β-barrel proteins of the outer mitochondrial membrane. 著者: Astrid Klein / Lars Israel / Sebastian W K Lackey / Frank E Nargang / Axel Imhof / Wolfgang Baumeister / Walter Neupert / Dennis R Thomas /  要旨: The TOB-SAM complex is an essential component of the mitochondrial outer membrane that mediates the insertion of β-barrel precursor proteins into the membrane. We report here its isolation and ...The TOB-SAM complex is an essential component of the mitochondrial outer membrane that mediates the insertion of β-barrel precursor proteins into the membrane. We report here its isolation and determine its size, composition, and structural organization. The complex from Neurospora crassa was composed of Tob55-Sam50, Tob38-Sam35, and Tob37-Sam37 in a stoichiometry of 1:1:1 and had a molecular mass of 140 kD. A very minor fraction of the purified complex was associated with one Mdm10 protein. Using molecular homology modeling for Tob55 and cryoelectron microscopy reconstructions of the TOB complex, we present a model of the TOB-SAM complex that integrates biochemical and structural data. We discuss our results and the structural model in the context of a possible mechanism of the TOB insertase. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2203.map.gz emd_2203.map.gz | 9.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2203-v30.xml emd-2203-v30.xml emd-2203.xml emd-2203.xml | 10.3 KB 10.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-2203.jpg EMD-2203.jpg | 29.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2203 http://ftp.pdbj.org/pub/emdb/structures/EMD-2203 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2203 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2203 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2203_validation.pdf.gz emd_2203_validation.pdf.gz | 207 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2203_full_validation.pdf.gz emd_2203_full_validation.pdf.gz | 206.1 KB | 表示 | |
| XML形式データ |  emd_2203_validation.xml.gz emd_2203_validation.xml.gz | 5.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2203 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2203 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2203 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2203 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2203.map.gz / 形式: CCP4 / 大きさ: 10.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2203.map.gz / 形式: CCP4 / 大きさ: 10.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of Tob55 dimers isolated using the 9xHis tag on the Tob55 subunit. The Tob55 dimer is a complex identified in all outer membrane preparations but isolated only when the his-tag is on tob55. The dimer co-purifies with the TOB complex and was identified as a subclass of particles present in the his-Tob55 data sets. The mol. wgt. is 101 kDa. Two-fold symmetry has been applied | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.78 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Tob55 dimer
| 全体 | 名称: Tob55 dimer |
|---|---|
| 要素 |
|
-超分子 #1000: Tob55 dimer
| 超分子 | 名称: Tob55 dimer / タイプ: sample / ID: 1000 詳細: The complexes were monodisperse. A mitochondrial outer membrane complex which can be identified as a component of the outer mitochondrial membrane but whose function is unknown. 集合状態: dimeric / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 101 KDa / 理論値: 101 KDa 手法: Blue native gel electrophoresis and Isotope dilution mass spectroscopy analysis of bands isolated from BNGE gels. |
-分子 #1: Tob55 short
| 分子 | 名称: Tob55 short / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 集合状態: dimer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (菌類) / 株: Tob55 short HT / Organelle: mitochondria / 細胞中の位置: outer membrane Neurospora crassa (菌類) / 株: Tob55 short HT / Organelle: mitochondria / 細胞中の位置: outer membrane |
| 分子量 | 実験値: 50.7 KDa / 理論値: 50.7 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8.5 詳細: 1mM PMSF, 0.08%(v/v) Triton X-100, 50 mM HEPES pH 8.5 |
| グリッド | 詳細: lacey carbon films on 200 mesh Molybdenum grids |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER 手法: blot for 4-5 seconds before plunging with whatman filter paper #1 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected using the live FFT at imaging magnification. |
| 日付 | 2009年10月7日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: FEI EAGLE (4k x 4k) / 平均電子線量: 20 e/Å2 詳細: Images collected using TOM_acquisition software. 24800 dimers were identified in various his-Tob55 datasets. In the end 19500 were included in the final reconstruction. Two-fold symmetry was ...詳細: Images collected using TOM_acquisition software. 24800 dimers were identified in various his-Tob55 datasets. In the end 19500 were included in the final reconstruction. Two-fold symmetry was applied during the last 5 rounds of refinement. |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 84270 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): -3.7 µm / 最小 デフォーカス(公称値): -0.5 µm / 倍率(公称値): 62000 |
| 試料ステージ | 試料ホルダー: Gatan 656 side entry holder / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Phase and astigmatism correction applied to each micrograph |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 13.4 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER, TOM_toolbox 詳細: Final maps were reconstructed from images that had stable alignment parameters over the last 4 rounds of refinement. Stable was defined by absolute accumulated changes in theta and psi of the ...詳細: Final maps were reconstructed from images that had stable alignment parameters over the last 4 rounds of refinement. Stable was defined by absolute accumulated changes in theta and psi of the projection matched of less than 10 degrees. 24800 dimers were identified in various his-Tob55 datasets. In the end 19500 were included in the final reconstruction. Two-fold symmetry was applied during the last 5 rounds of refinement. 使用した粒子像数: 1 |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)