+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21172 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | De novo designed tetrahedral nanoparticle T33_dn10 | |||||||||
 マップデータ マップデータ | T33_dn10 Nanoparticle Map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | De novo / Nanoparticle / DE NOVO PROTEIN | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.86 Å | |||||||||
 データ登録者 データ登録者 | Antanasijevic A / Ward AB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Tailored design of protein nanoparticle scaffolds for multivalent presentation of viral glycoprotein antigens. 著者: George Ueda / Aleksandar Antanasijevic / Jorge A Fallas / William Sheffler / Jeffrey Copps / Daniel Ellis / Geoffrey B Hutchinson / Adam Moyer / Anila Yasmeen / Yaroslav Tsybovsky / Young-Jun ...著者: George Ueda / Aleksandar Antanasijevic / Jorge A Fallas / William Sheffler / Jeffrey Copps / Daniel Ellis / Geoffrey B Hutchinson / Adam Moyer / Anila Yasmeen / Yaroslav Tsybovsky / Young-Jun Park / Matthew J Bick / Banumathi Sankaran / Rebecca A Gillespie / Philip Jm Brouwer / Peter H Zwart / David Veesler / Masaru Kanekiyo / Barney S Graham / Rogier W Sanders / John P Moore / Per Johan Klasse / Andrew B Ward / Neil P King / David Baker /   要旨: Multivalent presentation of viral glycoproteins can substantially increase the elicitation of antigen-specific antibodies. To enable a new generation of anti-viral vaccines, we designed self- ...Multivalent presentation of viral glycoproteins can substantially increase the elicitation of antigen-specific antibodies. To enable a new generation of anti-viral vaccines, we designed self-assembling protein nanoparticles with geometries tailored to present the ectodomains of influenza, HIV, and RSV viral glycoprotein trimers. We first designed trimers tailored for antigen fusion, featuring N-terminal helices positioned to match the C termini of the viral glycoproteins. Trimers that experimentally adopted their designed configurations were incorporated as components of tetrahedral, octahedral, and icosahedral nanoparticles, which were characterized by cryo-electron microscopy and assessed for their ability to present viral glycoproteins. Electron microscopy and antibody binding experiments demonstrated that the designed nanoparticles presented antigenically intact prefusion HIV-1 Env, influenza hemagglutinin, and RSV F trimers in the predicted geometries. This work demonstrates that antigen-displaying protein nanoparticles can be designed from scratch, and provides a systematic way to investigate the influence of antigen presentation geometry on the immune response to vaccination. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21172.map.gz emd_21172.map.gz | 96.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21172-v30.xml emd-21172-v30.xml emd-21172.xml emd-21172.xml | 20.6 KB 20.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21172_fsc.xml emd_21172_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21172.png emd_21172.png | 190.6 KB | ||
| マスクデータ |  emd_21172_msk_1.map emd_21172_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-21172.cif.gz emd-21172.cif.gz | 6.2 KB | ||
| その他 |  emd_21172_half_map_1.map.gz emd_21172_half_map_1.map.gz emd_21172_half_map_2.map.gz emd_21172_half_map_2.map.gz | 80.7 MB 80.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21172 http://ftp.pdbj.org/pub/emdb/structures/EMD-21172 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21172 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21172 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21172.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21172.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T33_dn10 Nanoparticle Map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21172_msk_1.map emd_21172_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
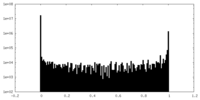
| 密度ヒストグラム |
-ハーフマップ: T33 dn10 Nanoparticle - Halfmap 1
| ファイル | emd_21172_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T33_dn10 Nanoparticle - Halfmap 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
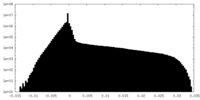
| 密度ヒストグラム |
-ハーフマップ: T33 dn10 Nanoparticle - Halfmap 2
| ファイル | emd_21172_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T33_dn10 Nanoparticle - Halfmap 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
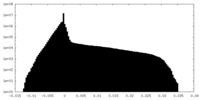
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : De novo designed tetrahedral nanoparticle T33_dn10
| 全体 | 名称: De novo designed tetrahedral nanoparticle T33_dn10 |
|---|---|
| 要素 |
|
-超分子 #1: De novo designed tetrahedral nanoparticle T33_dn10
| 超分子 | 名称: De novo designed tetrahedral nanoparticle T33_dn10 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Self-assembling nanoparticle with tetrahedral symmetry |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 546 KDa |
-分子 #1: T33_dn10A
| 分子 | 名称: T33_dn10A / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 14.082408 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGEEAELAYL LGELAYKLGE YRIAIRAYRI ALKRDPNNAE AWYNLGNAYY KQGDYDEAIE YYQKALELDP NNAEAWYNLG NAYYKQGDY DEAIEYYEKA LELDPENLEA LQNLLNAMDK QG |
-分子 #2: T33_dn10B
| 分子 | 名称: T33_dn10B / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 31.46683 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIEEVVAEMI DILAESSKKS IEELARAADN KTTEKAVAEA IEEIARLATA AIQLIEALAK NLASEEFMAR AISAIAELAK KAIEAIYRL ADNHTTDTFM ARAIAAIANL AVTAILAIAA LASNHTTEEF MARAISAIAE LAKKAIEAIY RLADNHTTDK F MAAAIEAI ...文字列: MIEEVVAEMI DILAESSKKS IEELARAADN KTTEKAVAEA IEEIARLATA AIQLIEALAK NLASEEFMAR AISAIAELAK KAIEAIYRL ADNHTTDTFM ARAIAAIANL AVTAILAIAA LASNHTTEEF MARAISAIAE LAKKAIEAIY RLADNHTTDK F MAAAIEAI ALLATLAILA IALLASNHTT EKFMARAIMA IAILAAKAIE AIYRLADNHT SPTYIEKAIE AIEKIARKAI KA IEMLAKN ITTEEYKEKA KKIIDIIRKL AKMAIKKLED NRTLEHHHHH H |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: TBS buffer, 0.2um filtered, 0.06mM DDM detergent added immediately before freezing | |||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 11 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV 詳細: 0.06mM DDM detergent (from an 8X stock) added immediately before freezing. | |||||||||
| 詳細 | Nanoparticles are generated by co-expression of two components (A and B) in E coli. Assembled particles are purified using a combination of Ni-affinity chromatography and gel-filtration chromatography. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 90.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 502 / 平均電子線量: 50.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Tetrahedral nanoparticle T33_dn10 model was docked into the reconstructed map using UCSF Chimera. The model was then relaxed using a combination of Rosetta relaxed refinement and manual refinement in Coot. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER |
| 得られたモデル |  PDB-6vfh: |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)