+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21012 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A small terminase protein from a thermophilic phage with a fixed helix-turn-helix geometry, symmetric | |||||||||
 マップデータ マップデータ | Cryo-EM reconstruction of the thermophilic bacteriophage P74-26 small terminase- symmetric | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | small terminase / bacteriophage / helix-turn-helix / motor / VIRAL PROTEIN | |||||||||
| 機能・相同性 | : / : / Phage G20C small terminase, N-terminal domain / Phage G20C small terminase C-terminal domain / Terminase small subunit 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Thermus virus P74-26 (ウイルス) Thermus virus P74-26 (ウイルス) | |||||||||
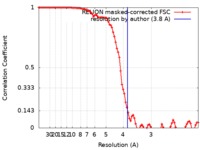
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Hayes JA / Hilbert BJ | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2020 ジャーナル: J Biol Chem / 年: 2020タイトル: A thermophilic phage uses a small terminase protein with a fixed helix-turn-helix geometry. 著者: Janelle A Hayes / Brendan J Hilbert / Christl Gaubitz / Nicholas P Stone / Brian A Kelch /  要旨: Tailed bacteriophages use a DNA-packaging motor to encapsulate their genome during viral particle assembly. The small terminase (TerS) component of this DNA-packaging machinery acts as a molecular ...Tailed bacteriophages use a DNA-packaging motor to encapsulate their genome during viral particle assembly. The small terminase (TerS) component of this DNA-packaging machinery acts as a molecular matchmaker that recognizes both the viral genome and the main motor component, the large terminase (TerL). However, how TerS binds DNA and the TerL protein remains unclear. Here we identified gp83 of the thermophilic bacteriophage P74-26 as the TerS protein. We found that TerS oligomerizes into a nonamer that binds DNA, stimulates TerL ATPase activity, and inhibits TerL nuclease activity. A cryo-EM structure of TerS revealed that it forms a ring with a wide central pore and radially arrayed helix-turn-helix domains. The structure further showed that these helix-turn-helix domains, which are thought to bind DNA by wrapping the double helix around the ring, are rigidly held in an orientation distinct from that seen in other TerS proteins. This rigid arrangement of the putative DNA-binding domain imposed strong constraints on how TerS can bind DNA. Finally, the TerS structure lacked the conserved C-terminal β-barrel domain used by other TerS proteins for binding TerL. This suggests that a well-ordered C-terminal β-barrel domain is not required for TerS to carry out its matchmaking function. Our work highlights a thermophilic system for studying the role of small terminase proteins in viral maturation and presents the structure of TerS, revealing key differences between this thermophilic phage and its mesophilic counterparts. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21012.map.gz emd_21012.map.gz | 5.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21012-v30.xml emd-21012-v30.xml emd-21012.xml emd-21012.xml | 18.4 KB 18.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21012_fsc.xml emd_21012_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21012.png emd_21012.png | 75.9 KB | ||
| Filedesc metadata |  emd-21012.cif.gz emd-21012.cif.gz | 5.7 KB | ||
| その他 |  emd_21012_half_map_1.map.gz emd_21012_half_map_1.map.gz emd_21012_half_map_2.map.gz emd_21012_half_map_2.map.gz | 49.6 MB 49.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21012 http://ftp.pdbj.org/pub/emdb/structures/EMD-21012 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21012 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21012 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21012_validation.pdf.gz emd_21012_validation.pdf.gz | 557.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21012_full_validation.pdf.gz emd_21012_full_validation.pdf.gz | 556.9 KB | 表示 | |
| XML形式データ |  emd_21012_validation.xml.gz emd_21012_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_21012_validation.cif.gz emd_21012_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21012 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21012 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21012 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21012 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21012.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21012.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the thermophilic bacteriophage P74-26 small terminase- symmetric | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.059 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
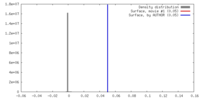
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map 1 of the 9-fold symmetric cryoEM...
| ファイル | emd_21012_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 of the 9-fold symmetric cryoEM reconstruction of the small terminase protein from the thermophilic bacteriophage P74-26. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
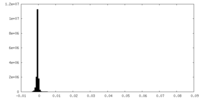
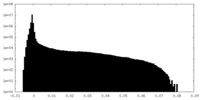
| 密度ヒストグラム |
-ハーフマップ: Half map 2 of the 9-fold symmetric cryoEM...
| ファイル | emd_21012_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 of the 9-fold symmetric cryoEM reconstruction of the small terminase protein from the thermophilic bacteriophage P74-26. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
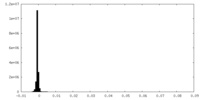
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Gene product 83 of the Thermus thermophilus bacteriophage P74-26
| 全体 | 名称: Gene product 83 of the Thermus thermophilus bacteriophage P74-26 |
|---|---|
| 要素 |
|
-超分子 #1: Gene product 83 of the Thermus thermophilus bacteriophage P74-26
| 超分子 | 名称: Gene product 83 of the Thermus thermophilus bacteriophage P74-26 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Thermus virus P74-26 (ウイルス) Thermus virus P74-26 (ウイルス) |
| 分子量 | 理論値: 171 KDa |
-分子 #1: Small terminase protein
| 分子 | 名称: Small terminase protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermus virus P74-26 (ウイルス) Thermus virus P74-26 (ウイルス) |
| 分子量 | 理論値: 19.080887 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPHMSVSFRD RVLKLYLLGF DPSEIAQTLS LDAKRKVTEE EVLHVLAEAR ELLSALPSLE DIRAEVGQAL ERARIFQKDL LAIYQNMLR NYNAMMEGLT EHPDGTPVIG VRPADIAAMA DRIMKIDQER ITALLNSLKV LGHVGSTTAG ALPSATELVS V EELVAEVA DETPKT UniProtKB: Terminase small subunit |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.4 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER / 詳細: 20 mA | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | This sample was monodisperse (PDI= 1.000) |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 2 / 実像数: 2822 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 2.6 µm 最小 デフォーカス(補正後): 1.4000000000000001 µm 倍率(補正後): 13000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)