+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20579 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
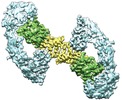
| タイトル | Cryo-EM structure of RET/GFRa3/ARTN extracellular complex. The 3D refinement was applied with C2 symmetry | |||||||||
 マップデータ マップデータ | Cryo-EM structure of RET/GFRa3/ARTN extracellular complex. The 3D refinement was applied with C2 symmetry. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RET / receptor tyrosine kinase / cryo-EM / SIGNALING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glial cell-derived neurotrophic factor receptor activity / glial cell-derived neurotrophic factor receptor binding / GDF15-GFRAL signaling pathway / Peyer's patch morphogenesis / positive regulation of metanephric glomerulus development / ureter maturation / embryonic epithelial tube formation / glial cell-derived neurotrophic factor receptor signaling pathway / lymphocyte migration into lymphoid organs / posterior midgut development ...glial cell-derived neurotrophic factor receptor activity / glial cell-derived neurotrophic factor receptor binding / GDF15-GFRAL signaling pathway / Peyer's patch morphogenesis / positive regulation of metanephric glomerulus development / ureter maturation / embryonic epithelial tube formation / glial cell-derived neurotrophic factor receptor signaling pathway / lymphocyte migration into lymphoid organs / posterior midgut development / SUMO is conjugated to E1 (UBA2:SAE1) / SUMOylation of nuclear envelope proteins / SUMO is transferred from E1 to E2 (UBE2I, UBC9) / SUMO is proteolytically processed / positive regulation of peptidyl-serine phosphorylation of STAT protein / membrane protein proteolysis / axon guidance receptor activity / Formation of the ureteric bud / SUMOylation of transcription factors / Postmitotic nuclear pore complex (NPC) reformation / SUMOylation of transcription cofactors / positive regulation of neuron maturation / neuron cell-cell adhesion / septin ring / Formation of the nephric duct / SUMOylation of DNA damage response and repair proteins / enteric nervous system development / SUMOylation of DNA replication proteins / peripheral nervous system development / sympathetic nervous system development / innervation / induction of positive chemotaxis / plasma membrane protein complex / SUMOylation of SUMOylation proteins / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / neuron maturation / positive regulation of extrinsic apoptotic signaling pathway in absence of ligand / SUMOylation of RNA binding proteins / NCAM1 interactions / positive regulation of cell adhesion mediated by integrin / SUMOylation of chromatin organization proteins / neural crest cell migration / ureteric bud development / extrinsic component of membrane / regulation of axonogenesis / response to pain / homophilic cell adhesion via plasma membrane adhesion molecules / ubiquitin-like protein ligase binding / protein sumoylation / RET signaling / positive regulation of cell size / neuroblast proliferation / regulation of cell adhesion / cellular response to retinoic acid / NPAS4 regulates expression of target genes / transmembrane receptor protein tyrosine kinase activity / condensed nuclear chromosome / axon guidance / growth factor activity / neuron migration / receptor protein-tyrosine kinase / receptor tyrosine kinase binding / : / positive regulation of neuron projection development / protein tag activity / MAPK cascade / signaling receptor activity / retina development in camera-type eye / nervous system development / RAF/MAP kinase cascade / protein tyrosine kinase activity / positive regulation of MAPK cascade / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / early endosome / receptor complex / endosome membrane / positive regulation of cell migration / response to xenobiotic stimulus / protein phosphorylation / axon / external side of plasma membrane / signaling receptor binding / neuronal cell body / dendrite / calcium ion binding / positive regulation of gene expression / positive regulation of DNA-templated transcription / signal transduction / extracellular space / extracellular region / ATP binding / identical protein binding / nucleus / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Li J / Shang GJ | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: Cryo-EM analyses reveal the common mechanism and diversification in the activation of RET by different ligands. 著者: Jie Li / Guijun Shang / Yu-Ju Chen / Chad A Brautigam / Jen Liou / Xuewu Zhang / Xiao-Chen Bai /  要旨: RET is a receptor tyrosine kinase (RTK) that plays essential roles in development and has been implicated in several human diseases. Different from most of RTKs, RET requires not only its cognate ...RET is a receptor tyrosine kinase (RTK) that plays essential roles in development and has been implicated in several human diseases. Different from most of RTKs, RET requires not only its cognate ligands but also co-receptors for activation, the mechanisms of which remain unclear due to lack of high-resolution structures of the ligand/co-receptor/receptor complexes. Here, we report cryo-EM structures of the extracellular region ternary complexes of GDF15/GFRAL/RET, GDNF/GFRα1/RET, NRTN/GFRα2/RET and ARTN/GFRα3/RET. These structures reveal that all the four ligand/co-receptor pairs, while using different atomic interactions, induce a specific dimerization mode of RET that is poised to bring the two kinase domains into close proximity for cross-phosphorylation. The NRTN/GFRα2/RET dimeric complex further pack into a tetrameric assembly, which is shown by our cell-based assays to regulate the endocytosis of RET. Our analyses therefore reveal both the common mechanism and diversification in the activation of RET by different ligands. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20579.map.gz emd_20579.map.gz | 49.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20579-v30.xml emd-20579-v30.xml emd-20579.xml emd-20579.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20579.png emd_20579.png | 240.6 KB | ||
| Filedesc metadata |  emd-20579.cif.gz emd-20579.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20579 http://ftp.pdbj.org/pub/emdb/structures/EMD-20579 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20579 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20579 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20579_validation.pdf.gz emd_20579_validation.pdf.gz | 580.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20579_full_validation.pdf.gz emd_20579_full_validation.pdf.gz | 580.5 KB | 表示 | |
| XML形式データ |  emd_20579_validation.xml.gz emd_20579_validation.xml.gz | 5.9 KB | 表示 | |
| CIF形式データ |  emd_20579_validation.cif.gz emd_20579_validation.cif.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20579 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20579 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20579 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20579 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20579.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20579.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of RET/GFRa3/ARTN extracellular complex. The 3D refinement was applied with C2 symmetry. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : RET, GFRAL and GDF15 extracellular complex
| 全体 | 名称: RET, GFRAL and GDF15 extracellular complex |
|---|---|
| 要素 |
|
-超分子 #1: RET, GFRAL and GDF15 extracellular complex
| 超分子 | 名称: RET, GFRAL and GDF15 extracellular complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 200 kDa/nm |
-分子 #1: Ubiquitin-like protein SMT3,Artemin
| 分子 | 名称: Ubiquitin-like protein SMT3,Artemin / タイプ: protein_or_peptide / ID: 1 詳細: A SUMO protein was fused to the N-terminal of NRTN for the protein expression コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.793287 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MASMSDSEVN QEAKPEVKPE VKPETHINLK VSDGSSEIFF KIKKTTPLRR LMEAFAKRQG KEMDSLRFL YDGIRIQADQ TPEDLDMEDN DIIEAHREQI GGSAGGPGSR ARAAGARGCR LRSQLVPVRA LGLGHRSDEL V RFRFCSGS ...文字列: MGSSHHHHHH SSGLVPRGSH MASMSDSEVN QEAKPEVKPE VKPETHINLK VSDGSSEIFF KIKKTTPLRR LMEAFAKRQG KEMDSLRFL YDGIRIQADQ TPEDLDMEDN DIIEAHREQI GGSAGGPGSR ARAAGARGCR LRSQLVPVRA LGLGHRSDEL V RFRFCSGS CRRARSPHDL SLASLLGAGA LRPPPGSRPV SQPCCRPTRY EAVSFMDVNS TWRTVDRLSA TACGCLG UniProtKB: Ubiquitin-like protein SMT3, Artemin |
-分子 #2: GDNF family receptor alpha-3
| 分子 | 名称: GDNF family receptor alpha-3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 38.370023 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DPLPTESRLM NSCLQARRKC QADPTCSAAY HHLDSCTSSI STPLPSEEPS VPADCLEAAQ QLRNSSLIGC MCHRRMKNQV ACLDIYWTV HRARSLGNYE LDVSPYEDTV TSKPWKMNLS KLNMLKPDSD LCLKFAMLCT LNDKCDRLRK AYGEACSGPH C QRHVCLRQ ...文字列: DPLPTESRLM NSCLQARRKC QADPTCSAAY HHLDSCTSSI STPLPSEEPS VPADCLEAAQ QLRNSSLIGC MCHRRMKNQV ACLDIYWTV HRARSLGNYE LDVSPYEDTV TSKPWKMNLS KLNMLKPDSD LCLKFAMLCT LNDKCDRLRK AYGEACSGPH C QRHVCLRQ LLTFFEKAAE PHAQGLLLCP CAPNDRGCGE RRRNTIAPNC ALPPVAPNCL ELRRLCFSDP LCRSRLVDFQ TH CHPMDIL GTCATEQSRC LRAYLGLIGT AMTPNFVSNV NTSVALSCTC RGSGNLQEEC EMLEGFFSHN PCLTEAIAAK MRF HSQLFS QDWPHPGTHH HHHHHH UniProtKB: GDNF family receptor alpha-3 |
-分子 #3: Proto-oncogene tyrosine-protein kinase receptor Ret
| 分子 | 名称: Proto-oncogene tyrosine-protein kinase receptor Ret / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO / EC番号: receptor protein-tyrosine kinase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 69.100812 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LYFSRDAYWE KLYVDQAAGT PLLYVHALRD APEEVPSFRL GQHLYGTYRT RLHENNWICI QEDTGLLYLN RSLDHSSWEK LSVRNHGFP LLTVYLKVFL SPTSLREGEC QWPGCARVYF SFFNTSFPAC SSLKPRELCF PETRPSFRIR ENRPPGTFHQ F RLLPVQFL ...文字列: LYFSRDAYWE KLYVDQAAGT PLLYVHALRD APEEVPSFRL GQHLYGTYRT RLHENNWICI QEDTGLLYLN RSLDHSSWEK LSVRNHGFP LLTVYLKVFL SPTSLREGEC QWPGCARVYF SFFNTSFPAC SSLKPRELCF PETRPSFRIR ENRPPGTFHQ F RLLPVQFL CPNISVAYRL LEGEGLPFRC APDSLEVSTR WALDREQREK YELVAVCTVH AGAREEVVMV PFPVTVYDED DS APTFPAG VDTASAVVEF KRKEDTVVAT LRVFDADVVP ASGELVRRYT STLLPGDTWA QQTFRVEHWP NETSVQANGS FVR ATVHDY RLVLNRNLSI SENRTMQLAV LVNDSDFQGP GAGVLLLHFN VSVLPVSLHL PSTYSLSVSR RARRFAQIGK VCVE NCQAF SGINVQYKLH SSGANCSTLG VVTSAEDTSG ILFVNDTKAL RRPKCAELHY MVVATDQQTS RQAQAQLLVT VEGSY VAEE AGCPLSCAVS KRRLECEECG GLGSPTGRCE WRQGDGKGIT RNFSTCSPST KTCPDGHCDV VETQDINICP QDCLRG SIV GGHEPGEPRG IKAGYGTCNC FPEEEKCFCE PEDIQDPLCD ELCRGTHHHH HHHH UniProtKB: Proto-oncogene tyrosine-protein kinase receptor Ret |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 14 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #5: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 5 / コピー数: 8 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-30 / 撮影したグリッド数: 1 / 平均露光時間: 15.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 46729 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 190 |
|---|---|
| 得られたモデル |  PDB-6q2s: |
 ムービー
ムービー コントローラー
コントローラー































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















