+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20076 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
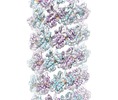
| タイトル | Cryo-EM structure of human TorsinA filament | |||||||||
 マップデータ マップデータ | Human TorsinA filament | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AAA+ ATPase / nucleotide binding / nuclear envelope / endoplasmic reticulum / membrane / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報synaptic vesicle membrane organization / nuclear membrane organization / regulation of dopamine uptake involved in synaptic transmission / nuclear envelope organization / intermediate filament cytoskeleton organization / protein deneddylation / regulation of protein localization to cell surface / positive regulation of synaptic vesicle endocytosis / misfolded protein binding / wound healing, spreading of cells ...synaptic vesicle membrane organization / nuclear membrane organization / regulation of dopamine uptake involved in synaptic transmission / nuclear envelope organization / intermediate filament cytoskeleton organization / protein deneddylation / regulation of protein localization to cell surface / positive regulation of synaptic vesicle endocytosis / misfolded protein binding / wound healing, spreading of cells / synaptic vesicle transport / : / kinesin binding / protein localization to nucleus / cytoskeletal protein binding / : / ERAD pathway / cytoplasmic vesicle membrane / secretory granule / ATP-dependent protein folding chaperone / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / neuron projection development / Cargo recognition for clathrin-mediated endocytosis / synaptic vesicle / unfolded protein binding / nuclear envelope / growth cone / response to oxidative stress / nuclear membrane / cytoskeleton / cell adhesion / endoplasmic reticulum lumen / intracellular membrane-bounded organelle / endoplasmic reticulum membrane / endoplasmic reticulum / ATP hydrolysis activity / extracellular exosome / ATP binding / identical protein binding / membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||
 データ登録者 データ登録者 | Zheng W / Demircioglu FE | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: The AAA + ATPase TorsinA polymerizes into hollow helical tubes with 8.5 subunits per turn. 著者: F Esra Demircioglu / Weili Zheng / Alexander J McQuown / Nolan K Maier / Nicki Watson / Iain M Cheeseman / Vladimir Denic / Edward H Egelman / Thomas U Schwartz /  要旨: TorsinA is an ER-resident AAA + ATPase, whose deletion of glutamate E303 results in the genetic neuromuscular disease primary dystonia. TorsinA is an unusual AAA + ATPase that needs an ...TorsinA is an ER-resident AAA + ATPase, whose deletion of glutamate E303 results in the genetic neuromuscular disease primary dystonia. TorsinA is an unusual AAA + ATPase that needs an external activator. Also, it likely does not thread a peptide substrate through a narrow central channel, in contrast to its closest structural homologs. Here, we examined the oligomerization of TorsinA to get closer to a molecular understanding of its still enigmatic function. We observe TorsinA to form helical filaments, which we analyzed by cryo-electron microscopy using helical reconstruction. The 4.4 Å structure reveals long hollow tubes with a helical periodicity of 8.5 subunits per turn, and an inner channel of ~ 4 nm diameter. We further show that the protein is able to induce tubulation of membranes in vitro, an observation that may reflect an entirely new characteristic of AAA + ATPases. We discuss the implications of these observations for TorsinA function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20076.map.gz emd_20076.map.gz | 12.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20076-v30.xml emd-20076-v30.xml emd-20076.xml emd-20076.xml | 11.3 KB 11.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20076.png emd_20076.png | 218 KB | ||
| Filedesc metadata |  emd-20076.cif.gz emd-20076.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20076 http://ftp.pdbj.org/pub/emdb/structures/EMD-20076 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20076 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20076 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20076_validation.pdf.gz emd_20076_validation.pdf.gz | 566.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20076_full_validation.pdf.gz emd_20076_full_validation.pdf.gz | 565.8 KB | 表示 | |
| XML形式データ |  emd_20076_validation.xml.gz emd_20076_validation.xml.gz | 5.9 KB | 表示 | |
| CIF形式データ |  emd_20076_validation.cif.gz emd_20076_validation.cif.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20076 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20076 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20076 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20076 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20076.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20076.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human TorsinA filament | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.169 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
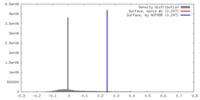
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TorsinA
| 全体 | 名称: TorsinA |
|---|---|
| 要素 |
|
-超分子 #1: TorsinA
| 超分子 | 名称: TorsinA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Torsin-1A
| 分子 | 名称: Torsin-1A / タイプ: protein_or_peptide / ID: 1 / コピー数: 25 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 32.590248 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GQKRSLSREA LQKDLDDNLF GQHLAKKIIL NAVFGFINNP KPKKPLTLSL HGWTGTGKNF VSKIIAENIY EGGLNSDYVH LFVATLHFP HASNITLYKD QLQLWIRGNV SACARSIFIF DEMDKMHAGL IDAIKPFLDY YDLVDGVSYQ KAMFIFLSNA G AERITDVA ...文字列: GQKRSLSREA LQKDLDDNLF GQHLAKKIIL NAVFGFINNP KPKKPLTLSL HGWTGTGKNF VSKIIAENIY EGGLNSDYVH LFVATLHFP HASNITLYKD QLQLWIRGNV SACARSIFIF DEMDKMHAGL IDAIKPFLDY YDLVDGVSYQ KAMFIFLSNA G AERITDVA LDFWRSGKQR EDIKLKDIEH ALSVSVFNNK NSGFWHSSLI DRNLIDYFVP FLPLEYKHLK MCIRVEMQSR GY EIDEDIV SRVAEEMTFF PKEERVFSDK GCKTVFTKLD YYYDD UniProtKB: Torsin-1A |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 25 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 5.53 Å 想定した対称性 - らせんパラメータ - ΔΦ: 42.51 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 4.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: SPIDER / 使用した粒子像数: 75909 |
|---|---|
| Segment selection | 選択した数: 75909 / ソフトウェア - 名称: EMAN2 / ソフトウェア - 詳細: EMAN2 e2helixboxer.py |
| 初期モデル | モデルのタイプ: NONE / 詳細: Featureless cylinder |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: SPIDER |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)