+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Proximal +1 nucleosome focused map | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mammalian PIC / +1 nucleosome / transcription initiation / TRANSCRIPTION | |||||||||
| 生物種 |  unidentified adenovirus (ウイルス) unidentified adenovirus (ウイルス) | |||||||||
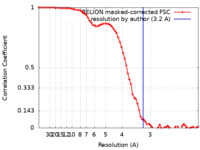
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Abril-Garrido J / Dienemann C / Grabbe F / Velychko T / Lidschreiber M / Wang H / Cramer P | |||||||||
| 資金援助 |  ドイツ, European Union, 2件 ドイツ, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: Structural basis of transcription reduction by a promoter-proximal +1 nucleosome. 著者: Julio Abril-Garrido / Christian Dienemann / Frauke Grabbe / Taras Velychko / Michael Lidschreiber / Haibo Wang / Patrick Cramer /  要旨: At active human genes, the +1 nucleosome is located downstream of the RNA polymerase II (RNA Pol II) pre-initiation complex (PIC). However, at inactive genes, the +1 nucleosome is found further ...At active human genes, the +1 nucleosome is located downstream of the RNA polymerase II (RNA Pol II) pre-initiation complex (PIC). However, at inactive genes, the +1 nucleosome is found further upstream, at a promoter-proximal location. Here, we establish a model system to show that a promoter-proximal +1 nucleosome can reduce RNA synthesis in vivo and in vitro, and we analyze its structural basis. We find that the PIC assembles normally when the edge of the +1 nucleosome is located 18 base pairs (bp) downstream of the transcription start site (TSS). However, when the nucleosome edge is located further upstream, only 10 bp downstream of the TSS, the PIC adopts an inhibited state. The transcription factor IIH (TFIIH) shows a closed conformation and its subunit XPB contacts DNA with only one of its two ATPase lobes, inconsistent with DNA opening. These results provide a mechanism for nucleosome-dependent regulation of transcription initiation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
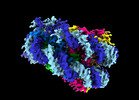
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16337.map.gz emd_16337.map.gz | 23.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16337-v30.xml emd-16337-v30.xml emd-16337.xml emd-16337.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16337_fsc.xml emd_16337_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16337.png emd_16337.png | 84 KB | ||
| マスクデータ |  emd_16337_msk_1.map emd_16337_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_16337_additional_1.map.gz emd_16337_additional_1.map.gz emd_16337_half_map_1.map.gz emd_16337_half_map_1.map.gz emd_16337_half_map_2.map.gz emd_16337_half_map_2.map.gz | 28.3 MB 23.3 MB 23.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16337 http://ftp.pdbj.org/pub/emdb/structures/EMD-16337 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16337 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16337 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16337_validation.pdf.gz emd_16337_validation.pdf.gz | 784.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16337_full_validation.pdf.gz emd_16337_full_validation.pdf.gz | 783.7 KB | 表示 | |
| XML形式データ |  emd_16337_validation.xml.gz emd_16337_validation.xml.gz | 12.5 KB | 表示 | |
| CIF形式データ |  emd_16337_validation.cif.gz emd_16337_validation.cif.gz | 17.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16337 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16337 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16337 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16337 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16337.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16337.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16337_msk_1.map emd_16337_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_16337_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16337_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16337_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RNA polymerase II core pre-initiation complex with the proximal +...
| 全体 | 名称: RNA polymerase II core pre-initiation complex with the proximal +1 nucleosome |
|---|---|
| 要素 |
|
-超分子 #1: RNA polymerase II core pre-initiation complex with the proximal +...
| 超分子 | 名称: RNA polymerase II core pre-initiation complex with the proximal +1 nucleosome タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 分子量 | 理論値: 240 KDa |
-超分子 #2: Nucleosome (protein)
| 超分子 | 名称: Nucleosome (protein) / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種: |
-超分子 #3: Nucleosome (DNA)
| 超分子 | 名称: Nucleosome (DNA) / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #5-#6 |
|---|---|
| 由来(天然) | 生物種:  unidentified adenovirus (ウイルス) unidentified adenovirus (ウイルス) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R3.5/1 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 3 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum SE / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 36478 / 平均露光時間: 3.0 sec. / 平均電子線量: 50.45 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.3 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)