+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human leptin in complex with the human LEP-R ectodomain fused to a C-terminal trimeric isoleucine GCN4 zipper (closed 3:3 model) | |||||||||
 マップデータ マップデータ | Sharpened cryo-EM map followin non-uniform refinement. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of lipoprotein lipid oxidation / cellular response to L-ascorbic acid / positive regulation of fat cell apoptotic process / negative regulation of glutamine transport /  leptin receptor activity / regulation of transport / negative regulation of appetite by leptin-mediated signaling pathway / negative regulation of glucagon secretion / regulation of endothelial cell proliferation / regulation of natural killer cell proliferation ...regulation of lipoprotein lipid oxidation / cellular response to L-ascorbic acid / positive regulation of fat cell apoptotic process / negative regulation of glutamine transport / leptin receptor activity / regulation of transport / negative regulation of appetite by leptin-mediated signaling pathway / negative regulation of glucagon secretion / regulation of endothelial cell proliferation / regulation of natural killer cell proliferation ...regulation of lipoprotein lipid oxidation / cellular response to L-ascorbic acid / positive regulation of fat cell apoptotic process / negative regulation of glutamine transport /  leptin receptor activity / regulation of transport / negative regulation of appetite by leptin-mediated signaling pathway / negative regulation of glucagon secretion / regulation of endothelial cell proliferation / regulation of natural killer cell proliferation / leptin receptor activity / regulation of transport / negative regulation of appetite by leptin-mediated signaling pathway / negative regulation of glucagon secretion / regulation of endothelial cell proliferation / regulation of natural killer cell proliferation /  leptin receptor binding / positive regulation of luteinizing hormone secretion / regulation of natural killer cell mediated cytotoxicity / leptin receptor binding / positive regulation of luteinizing hormone secretion / regulation of natural killer cell mediated cytotoxicity /  骨化 / regulation of natural killer cell activation / positive regulation of monoatomic ion transport / glycerol biosynthetic process / elastin metabolic process / leptin-mediated signaling pathway / positive regulation of follicle-stimulating hormone secretion / regulation of steroid biosynthetic process / regulation of intestinal cholesterol absorption / 骨化 / regulation of natural killer cell activation / positive regulation of monoatomic ion transport / glycerol biosynthetic process / elastin metabolic process / leptin-mediated signaling pathway / positive regulation of follicle-stimulating hormone secretion / regulation of steroid biosynthetic process / regulation of intestinal cholesterol absorption /  regulation of bone remodeling / regulation of brown fat cell differentiation / positive regulation of peroxisome proliferator activated receptor signaling pathway / adult feeding behavior / positive regulation of hepatic stellate cell activation / response to leptin / bone mineralization involved in bone maturation / regulation of bone remodeling / regulation of brown fat cell differentiation / positive regulation of peroxisome proliferator activated receptor signaling pathway / adult feeding behavior / positive regulation of hepatic stellate cell activation / response to leptin / bone mineralization involved in bone maturation /  regulation of feeding behavior / regulation of feeding behavior /  有性生殖 / activation of protein kinase C activity / negative regulation of cartilage development / multicellular organism development / 有性生殖 / activation of protein kinase C activity / negative regulation of cartilage development / multicellular organism development /  ovulation from ovarian follicle / negative regulation of appetite / positive regulation of developmental growth / leukocyte tethering or rolling / energy reserve metabolic process / prostaglandin secretion / negative regulation of glucose import / bile acid metabolic process / cellular response to leptin stimulus / hormone metabolic process / cardiac muscle hypertrophy / Signaling by Leptin / insulin secretion / intestinal absorption / aorta development / positive regulation of p38MAPK cascade / ovulation from ovarian follicle / negative regulation of appetite / positive regulation of developmental growth / leukocyte tethering or rolling / energy reserve metabolic process / prostaglandin secretion / negative regulation of glucose import / bile acid metabolic process / cellular response to leptin stimulus / hormone metabolic process / cardiac muscle hypertrophy / Signaling by Leptin / insulin secretion / intestinal absorption / aorta development / positive regulation of p38MAPK cascade /  cytokine receptor activity / peptide hormone receptor binding / negative regulation of vasoconstriction / cytokine receptor activity / peptide hormone receptor binding / negative regulation of vasoconstriction /  eating behavior / eating behavior /  regulation of gluconeogenesis / regulation of gluconeogenesis /  regulation of nitric-oxide synthase activity / fatty acid beta-oxidation / glycogen metabolic process / regulation of nitric-oxide synthase activity / fatty acid beta-oxidation / glycogen metabolic process /  central nervous system neuron development / central nervous system neuron development /  cytokine binding / regulation of cytokine production involved in inflammatory response / regulation of insulin secretion / response to dietary excess / negative regulation of lipid storage / transport across blood-brain barrier / T cell differentiation / positive regulation of TOR signaling / response to vitamin E / Synthesis, secretion, and deacylation of Ghrelin / glial cell proliferation / cytokine binding / regulation of cytokine production involved in inflammatory response / regulation of insulin secretion / response to dietary excess / negative regulation of lipid storage / transport across blood-brain barrier / T cell differentiation / positive regulation of TOR signaling / response to vitamin E / Synthesis, secretion, and deacylation of Ghrelin / glial cell proliferation /  regulation of angiogenesis / adipose tissue development / negative regulation of gluconeogenesis / regulation of angiogenesis / adipose tissue development / negative regulation of gluconeogenesis /  食作用 / 食作用 /  energy homeostasis / positive regulation of insulin receptor signaling pathway / cellular response to retinoic acid / positive regulation of T cell proliferation / positive regulation of tyrosine phosphorylation of STAT protein / positive regulation of interleukin-12 production / cholesterol metabolic process / negative regulation of autophagy / response to activity / female pregnancy / energy homeostasis / positive regulation of insulin receptor signaling pathway / cellular response to retinoic acid / positive regulation of T cell proliferation / positive regulation of tyrosine phosphorylation of STAT protein / positive regulation of interleukin-12 production / cholesterol metabolic process / negative regulation of autophagy / response to activity / female pregnancy /  糖新生 / determination of adult lifespan / positive regulation of interleukin-8 production / placenta development / positive regulation of receptor signaling pathway via JAK-STAT / response to insulin / lipid metabolic process / 糖新生 / determination of adult lifespan / positive regulation of interleukin-8 production / placenta development / positive regulation of receptor signaling pathway via JAK-STAT / response to insulin / lipid metabolic process /  hormone activity / Synthesis, secretion, and inactivation of Glucagon-like Peptide-1 (GLP-1) / hormone activity / Synthesis, secretion, and inactivation of Glucagon-like Peptide-1 (GLP-1) /  血圧 / cytokine-mediated signaling pathway / Transcriptional regulation of white adipocyte differentiation / positive regulation of protein import into nucleus / 血圧 / cytokine-mediated signaling pathway / Transcriptional regulation of white adipocyte differentiation / positive regulation of protein import into nucleus /  概日リズム / cellular response to insulin stimulus / positive regulation of interleukin-6 production 概日リズム / cellular response to insulin stimulus / positive regulation of interleukin-6 production類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
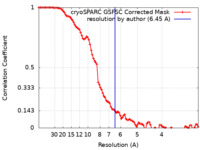
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.45 Å クライオ電子顕微鏡法 / 解像度: 6.45 Å | |||||||||
 データ登録者 データ登録者 | Verstraete K / Savvides SN / Verschueren KG / Tsirigotaki A | |||||||||
| 資金援助 |  ベルギー, 1件 ベルギー, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Mechanism of receptor assembly via the pleiotropic adipokine Leptin. 著者: Alexandra Tsirigotaki / Ann Dansercoer / Koen H G Verschueren / Iva Marković / Christoph Pollmann / Maximillian Hafer / Jan Felix / Catherine Birck / Wouter Van Putte / Dominiek Catteeuw / ...著者: Alexandra Tsirigotaki / Ann Dansercoer / Koen H G Verschueren / Iva Marković / Christoph Pollmann / Maximillian Hafer / Jan Felix / Catherine Birck / Wouter Van Putte / Dominiek Catteeuw / Jan Tavernier / J Fernando Bazan / Jacob Piehler / Savvas N Savvides / Kenneth Verstraete /     要旨: The adipokine Leptin activates its receptor LEP-R in the hypothalamus to regulate body weight and exerts additional pleiotropic functions in immunity, fertility and cancer. However, the structure and ...The adipokine Leptin activates its receptor LEP-R in the hypothalamus to regulate body weight and exerts additional pleiotropic functions in immunity, fertility and cancer. However, the structure and mechanism of Leptin-mediated LEP-R assemblies has remained unclear. Intriguingly, the signaling-competent isoform of LEP-R is only lowly abundant amid several inactive short LEP-R isoforms contributing to a mechanistic conundrum. Here we show by X-ray crystallography and cryo-EM that, in contrast to long-standing paradigms, Leptin induces type I cytokine receptor assemblies featuring 3:3 stoichiometry and demonstrate such Leptin-induced trimerization of LEP-R on living cells via single-molecule microscopy. In mediating these assemblies, Leptin undergoes drastic restructuring that activates its site III for binding to the Ig domain of an adjacent LEP-R. These interactions are abolished by mutations linked to obesity. Collectively, our study provides the structural and mechanistic framework for how evolutionarily conserved Leptin:LEP-R assemblies with 3:3 stoichiometry can engage distinct LEP-R isoforms to achieve signaling. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15681.map.gz emd_15681.map.gz | 79.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15681-v30.xml emd-15681-v30.xml emd-15681.xml emd-15681.xml | 25.4 KB 25.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15681_fsc.xml emd_15681_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15681.png emd_15681.png | 88.7 KB | ||
| マスクデータ |  emd_15681_msk_1.map emd_15681_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| その他 |  emd_15681_additional_1.map.gz emd_15681_additional_1.map.gz emd_15681_half_map_1.map.gz emd_15681_half_map_1.map.gz emd_15681_half_map_2.map.gz emd_15681_half_map_2.map.gz | 41.4 MB 77.7 MB 77.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15681 http://ftp.pdbj.org/pub/emdb/structures/EMD-15681 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15681 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15681 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8avfMC  7z3pC  7z3qC  7z3rC  8av2C  8avbC  8avcC  8avdC  8aveC  8avoC  8b7qC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15681.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15681.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened cryo-EM map followin non-uniform refinement. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.52 Å | ||||||||||||||||||||||||||||||||||||
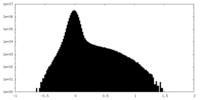
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15681_msk_1.map emd_15681_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
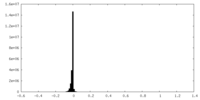
| 密度ヒストグラム |
-追加マップ: Non-sharpened cryo-EM map followin non-uniform refinement
| ファイル | emd_15681_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Non-sharpened cryo-EM map followin non-uniform refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
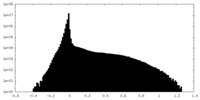
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_15681_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
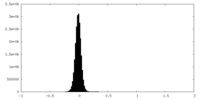
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_15681_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human leptin in complex with the human LEP-R ectodomain C-termina...
| 全体 | 名称: Human leptin in complex with the human LEP-R ectodomain C-terminally fused to a trimeric GCN4 isoleucine zipper tag |
|---|---|
| 要素 |
|
-超分子 #1: Human leptin in complex with the human LEP-R ectodomain C-termina...
| 超分子 | 名称: Human leptin in complex with the human LEP-R ectodomain C-terminally fused to a trimeric GCN4 isoleucine zipper tag タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all 詳細: Human leptin carrying an N-terminal His-tag and the human LEP-R ectodomain C-terminally fused to a trimeric GCN4 isoleucine zipper tag (without His-tag) were co-expressed in HEK293 FreeStyle ...詳細: Human leptin carrying an N-terminal His-tag and the human LEP-R ectodomain C-terminally fused to a trimeric GCN4 isoleucine zipper tag (without His-tag) were co-expressed in HEK293 FreeStyle cells in the presence of kifunensine. The resulting complex was purified from the conditioned medium by IMAC and SEC. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 350 KDa |
-分子 #1: Leptin
| 分子 | 名称: Leptin / タイプ: protein_or_peptide / ID: 1 詳細: N-terminally His-tagged human leptin was co-expressed with the human LEP-R ectodomain fused a trimeric GCN4 isoleucine zipper tag in HEK293 FreeStyle cells. コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 18.605061 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AHHHHHHPGG PGSENLYFQG GSTGGVPIQK VQDDTKTLIK TIVTRINDIS HTQSVSSKQK VTGLDFIPGL HPILTLSKMD QTLAVYQQI LTSMPSRNVI QISNDLENLR DLLHVLAFSK SCHLPWASGL ETLDSLGGVL EASGYSTEVV ALSRLQGSLQ D MLWQLDLS PGC |
-分子 #2: Leptin receptor
| 分子 | 名称: Leptin receptor / タイプ: protein_or_peptide / ID: 2 詳細: Human leptin carrying an N-terminal His-tag and the human LEP-R ectodomain C-terminally fused to a trimeric GCN4 isoleucine zipper tag (without His-tag) were co-expressed in HEK293 FreeStyle ...詳細: Human leptin carrying an N-terminal His-tag and the human LEP-R ectodomain C-terminally fused to a trimeric GCN4 isoleucine zipper tag (without His-tag) were co-expressed in HEK293 FreeStyle cells in the presence of kifunensine. The resulting complex was purified from the conditioned medium by IMAC and SEC. コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 98.822062 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: FNLSYPITPW RFKLSCMPPN STYDYFLLPA GLSKNTSNSN GHYETAVEPK FNSSGTHFSN LSKTTFHCCF RSEQDRNCSL CADNIEGKT FVSTVNSLVF QQIDANWNIQ CWLKGDLKLF ICYVESLFKN LFRNYNYKVH LLYVLPEVLE DSPLVPQKGS F QMVHCNCS ...文字列: FNLSYPITPW RFKLSCMPPN STYDYFLLPA GLSKNTSNSN GHYETAVEPK FNSSGTHFSN LSKTTFHCCF RSEQDRNCSL CADNIEGKT FVSTVNSLVF QQIDANWNIQ CWLKGDLKLF ICYVESLFKN LFRNYNYKVH LLYVLPEVLE DSPLVPQKGS F QMVHCNCS VHECCECLVP VPTAKLNDTL LMCLKITSGG VIFQSPLMSV QPINMVKPDP PLGLHMEITD DGNLKISWSS PP LVPFPLQ YQVKYSENST TVIREADKIV SATSLLVDSI LPGSSYEVQV RGKRLDGPGI WSDWSTPRVF TTQDVIYFPP KIL TSVGSN VSFHCIYKKE NKIVPSKEIV WWMNLAEKIP QSQYDVVSDH VSKVTFFNLN ETKPRGKFTY DAVYCCNEHE CHHR YAELY VIDVNINISC ETDGYLTKMT CRWSTSTIQS LAESTLQLRY HRSSLYCSDI PSIHPISEPK DCYLQSDGFY ECIFQ PIFL LSGYTMWIRI NHSLGSLDSP PTCVLPDSVV KPLPPSSVKA EITINIGLLK ISWEKPVFPE NNLQFQIRYG LSGKEV QWK MYEVYDAKSK SVSLPVPDLC AVYAVQVRCK RLDGLGYWSN WSNPAYTVVM DIKVPMRGPE FWRIINGDTM KKEKNVT LL WKPLMKNDSL CSVQRYVINH HTSCNGTWSE DVGNHTKFTF LWTEQAHTVT VLAINSIGAS VANFNLTFSW PMSKVNIV Q SLSAYPLNSS CVIVSWILSP SDYKLMYFII EWKNLNEDGE IKWLRISSSV KKYYIHDHFI PIEKYQFSLY PIFMEGVGK PKIINSFTQD DIEKHQSDST GGSGGSGGSG GSGGSRMKQI EDKIEEILSK IYHIENEIAR IKKLIGER |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: 20 mM HEPES, 150 mM NaCl, pH 7.4 | |||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 1 sec. / 前処理 - 雰囲気: AIR 詳細: 4 microliter of hLeptin:hLEP-RECD-tGCN4 complex at 0.3 mg.mL-1 was applied to a glow-discharged Quantifoil R 0.6/1 300 mesh golden grid coated with graphene (PUXANO), blotted for 1 s (blot ...詳細: 4 microliter of hLeptin:hLEP-RECD-tGCN4 complex at 0.3 mg.mL-1 was applied to a glow-discharged Quantifoil R 0.6/1 300 mesh golden grid coated with graphene (PUXANO), blotted for 1 s (blot force = 1) under 100% humidity at 295K and plunged into liquid ethane using an FEI Vitribot Mark IV | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: OTHER / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 13755 / 平均電子線量: 62.4 e/Å2 |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | (PDB ID: , ) |
|---|---|
| 詳細 | Atomic models for the 2:2 and 3:3 hLeptin:hLEP-R complexes were created based on the Alphafold2 predictions for hLEP-R and hLeptin, and the crystal structures for the mouse 3:3 Leptin:LEP-RIgCRH2 complex (pdb 7z3r) and the human Leptin:LEP-RCRH2 complex (pdb 7z3q) and fitted in the cryo-EM maps via Chimera. Atomic models were further refined via real space refinement in Phenix using rigid body refinement and coordinate refinement with reference restraints to the starting model. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-8avf: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)