登録情報 データベース : EMDB / ID : EMD-13159タイトル Structure of the V. vulnificus ExoY-G-actin-profilin complex Main map, was used to build VvExoY and Actin 複合体 : Structure of the V. vulnificus ExoY-G-actin-profilin complexタンパク質・ペプチド : Maltose/maltodextrin-binding periplasmic protein,RTX-toxinタンパク質・ペプチド : Actin, cytoplasmic 1タンパク質・ペプチド : Profilin-1リガンド : CALCIUM IONリガンド : ADENOSINE-5'-TRIPHOSPHATE機能・相同性 分子機能 ドメイン・相同性 構成要素
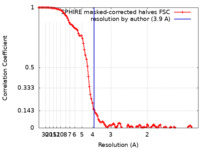
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Vibrio vulnificus (バクテリア) / Homo sapiens (ヒト)手法 / / 解像度 : 3.9 Å Belyy A / Merino F / Raunser S 資金援助 Organization Grant number 国 Max Planck Society
ジャーナル : Nat Commun / 年 : 2021タイトル : Mechanism of actin-dependent activation of nucleotidyl cyclase toxins from bacterial human pathogens.著者 : Alexander Belyy / Felipe Merino / Undine Mechold / Stefan Raunser / 要旨 : Bacterial human pathogens secrete initially inactive nucleotidyl cyclases that become potent enzymes by binding to actin inside eukaryotic host cells. The underlying molecular mechanism of this ... Bacterial human pathogens secrete initially inactive nucleotidyl cyclases that become potent enzymes by binding to actin inside eukaryotic host cells. The underlying molecular mechanism of this activation is, however, unclear. Here, we report structures of ExoY from Pseudomonas aeruginosa and Vibrio vulnificus bound to their corresponding activators F-actin and profilin-G-actin. The structures reveal that in contrast to the apo-state, two flexible regions become ordered and interact strongly with actin. The specific stabilization of these regions results in an allosteric stabilization of the nucleotide binding pocket and thereby to an activation of the enzyme. Differences in the sequence and conformation of the actin-binding regions are responsible for the selective binding to either F- or G-actin. Other nucleotidyl cyclase toxins that bind to calmodulin rather than actin undergo a similar disordered-to-ordered transition during activation, suggesting that the allosteric activation-by-stabilization mechanism of ExoY is conserved in these enzymes, albeit the different activator. 履歴 登録 2021年7月1日 - ヘッダ(付随情報) 公開 2021年11月17日 - マップ公開 2021年11月17日 - 更新 2021年12月1日 - 現状 2021年12月1日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 Vibrio vulnificus (バクテリア) /
Vibrio vulnificus (バクテリア) /  Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 ドイツ, 1件
ドイツ, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2021
ジャーナル: Nat Commun / 年: 2021

 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_13159.map.gz
emd_13159.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-13159-v30.xml
emd-13159-v30.xml emd-13159.xml
emd-13159.xml EMDBヘッダ
EMDBヘッダ emd_13159_fsc.xml
emd_13159_fsc.xml FSCデータファイル
FSCデータファイル emd_13159.png
emd_13159.png emd_13159_additional_1.map.gz
emd_13159_additional_1.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-13159
http://ftp.pdbj.org/pub/emdb/structures/EMD-13159 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13159
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13159 emd_13159_validation.pdf.gz
emd_13159_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_13159_full_validation.pdf.gz
emd_13159_full_validation.pdf.gz emd_13159_validation.xml.gz
emd_13159_validation.xml.gz emd_13159_validation.cif.gz
emd_13159_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13159
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13159 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13159
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13159 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_13159.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_13159.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 Vibrio vulnificus (バクテリア)
Vibrio vulnificus (バクテリア)
 Homo sapiens (ヒト)
Homo sapiens (ヒト) Trichoplusia ni (イラクサキンウワバ)
Trichoplusia ni (イラクサキンウワバ) Homo sapiens (ヒト)
Homo sapiens (ヒト)

 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー




































 Z
Z Y
Y X
X