+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12706 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Murine CIII2 focus-refined from supercomplex CICIII2 | |||||||||
 マップデータ マップデータ | murine CIII2 focus-refined from CICIII2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | mitochondria / respiratory chain / supercomplex / OXIDOREDUCTASE / complex III / complex IV / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Complex III assembly / subthalamus development / pons development / pyramidal neuron development / cerebellar Purkinje cell layer development / Respiratory electron transport / response to mercury ion / mitochondrial respiratory chain complex III assembly / thalamus development / respiratory chain complex III ...Complex III assembly / subthalamus development / pons development / pyramidal neuron development / cerebellar Purkinje cell layer development / Respiratory electron transport / response to mercury ion / mitochondrial respiratory chain complex III assembly / thalamus development / respiratory chain complex III / Mitochondrial protein degradation / : / : / response to alkaloid / quinol-cytochrome-c reductase / response to copper ion / ubiquinol-cytochrome-c reductase activity / response to glucagon / cellular respiration / midbrain development / hypothalamus development / mitochondrial electron transport, ubiquinol to cytochrome c / response to cobalamin / electron transport coupled proton transport / response to hyperoxia / animal organ regeneration / response to cadmium ion / response to hormone / respiratory electron transport chain / hippocampus development / response to activity / mitochondrial membrane / metalloendopeptidase activity / response to toxic substance / 2 iron, 2 sulfur cluster binding / response to calcium ion / myelin sheath / response to ethanol / mitochondrial inner membrane / response to hypoxia / oxidoreductase activity / response to xenobiotic stimulus / ubiquitin protein ligase binding / heme binding / protein-containing complex binding / protein-containing complex / mitochondrion / proteolysis / nucleoplasm / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
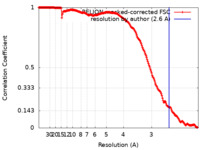
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Vercellino I / Sazanov LA | |||||||||
| 資金援助 |  オーストリア, 1件 オーストリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Structure and assembly of the mammalian mitochondrial supercomplex CIIICIV. 著者: Irene Vercellino / Leonid A Sazanov /  要旨: The enzymes of the mitochondrial electron transport chain are key players of cell metabolism. Despite being active when isolated, in vivo they associate into supercomplexes, whose precise role is ...The enzymes of the mitochondrial electron transport chain are key players of cell metabolism. Despite being active when isolated, in vivo they associate into supercomplexes, whose precise role is debated. Supercomplexes CIIICIV (refs. ), CICIII (ref. ) and CICIIICIV (respirasome) exist in mammals, but in contrast to CICIII and the respirasome, to date the only known eukaryotic structures of CIIICIV come from Saccharomyces cerevisiae and plants, which have different organization. Here we present the first, to our knowledge, structures of mammalian (mouse and ovine) CIIICIV and its assembly intermediates, in different conformations. We describe the assembly of CIIICIV from the CIII precursor to the final CIIICIV conformation, driven by the insertion of the N terminus of the assembly factor SCAF1 (ref. ) deep into CIII, while its C terminus is integrated into CIV. Our structures (which include CICIII and the respirasome) also confirm that SCAF1 is exclusively required for the assembly of CIIICIV and has no role in the assembly of the respirasome. We show that CIII is asymmetric due to the presence of only one copy of subunit 9, which straddles both monomers and prevents the attachment of a second copy of SCAF1 to CIII, explaining the presence of one copy of CIV in CIIICIV in mammals. Finally, we show that CIII and CIV gain catalytic advantage when assembled into the supercomplex and propose a role for CIIICIV in fine tuning the efficiency of electron transfer in the electron transport chain. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12706.map.gz emd_12706.map.gz | 71.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12706-v30.xml emd-12706-v30.xml emd-12706.xml emd-12706.xml | 28.8 KB 28.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12706_fsc.xml emd_12706_fsc.xml | 18.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12706.png emd_12706.png | 156.1 KB | ||
| Filedesc metadata |  emd-12706.cif.gz emd-12706.cif.gz | 8.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12706 http://ftp.pdbj.org/pub/emdb/structures/EMD-12706 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12706 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12706 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12706_validation.pdf.gz emd_12706_validation.pdf.gz | 587.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12706_full_validation.pdf.gz emd_12706_full_validation.pdf.gz | 586.6 KB | 表示 | |
| XML形式データ |  emd_12706_validation.xml.gz emd_12706_validation.xml.gz | 4.3 KB | 表示 | |
| CIF形式データ |  emd_12706_validation.cif.gz emd_12706_validation.cif.gz | 4.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12706 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12706 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12706 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12706 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12706.map.gz / 形式: CCP4 / 大きさ: 77.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12706.map.gz / 形式: CCP4 / 大きさ: 77.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | murine CIII2 focus-refined from CICIII2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.49981 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
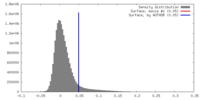
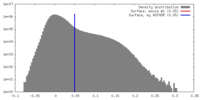
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Murine complex CIII2 focus-refined from supercomplex CICIII2
+超分子 #1: Murine complex CIII2 focus-refined from supercomplex CICIII2
+分子 #1: Cytochrome b-c1 complex subunit 1, mitochondrial
+分子 #2: Cytochrome b-c1 complex subunit 2, mitochondrial
+分子 #3: Cytochrome b
+分子 #4: Cytochrome c1, heme protein, mitochondrial
+分子 #5: Cytochrome b-c1 complex subunit Rieske, mitochondrial
+分子 #6: Cytochrome b-c1 complex subunit 7
+分子 #7: Cytochrome b-c1 complex subunit 8
+分子 #8: Cytochrome b-c1 complex subunit 6, mitochondrial
+分子 #9: Cytochrome b-c1 complex subunit 9
+分子 #10: Cytochrome b-c1 complex subunit 10
+分子 #11: Cytochrome b-c1 complex subunit 9
+分子 #12: 1,2-Distearoyl-sn-glycerophosphoethanolamine
+分子 #13: CARDIOLIPIN
+分子 #14: PROTOPORPHYRIN IX CONTAINING FE
+分子 #15: HEME C
+分子 #16: FE2/S2 (INORGANIC) CLUSTER
+分子 #17: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOCHOLINE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.7 構成要素:
詳細: CHAPS was added only upon freezing | |||||||||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 1 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 5 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 7245 / 平均露光時間: 1.17 sec. / 平均電子線量: 90.66 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.7 µm / 最小 デフォーカス(補正後): 0.2 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.4 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


























 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)