登録情報 データベース : EMDB / ID : EMD-11838タイトル Structure of the core MTA1/HDAC1/MBD2 NURD deacetylase complex Core NuRD deacetylase complex 複合体 : Core NuRD deacetylase complex containing two copies of MTA1 and HDAC1 and a single copy of MBD2タンパク質・ペプチド : Methyl-CpG-binding domain protein 2タンパク質・ペプチド : Metastasis-associated protein MTA1タンパク質・ペプチド : Histone deacetylase 1リガンド : INOSITOL HEXAKISPHOSPHATEリガンド : ZINC IONリガンド : POTASSIUM ION / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
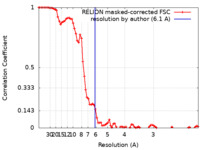
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 6.1 Å Millard CJ / Fairall L 資金援助 Organization Grant number 国 Wellcome Trust WT100237/Z/12/Z Medical Research Council (MRC, United Kingdom) MC_PC_17136 Wolfson Foundation
ジャーナル : Nucleic Acids Res / 年 : 2020タイトル : The topology of chromatin-binding domains in the NuRD deacetylase complex.著者 : Christopher J Millard / Louise Fairall / Timothy J Ragan / Christos G Savva / John W R Schwabe / 要旨 : Class I histone deacetylase complexes play essential roles in many nuclear processes. Whilst they contain a common catalytic subunit, they have diverse modes of action determined by associated ... Class I histone deacetylase complexes play essential roles in many nuclear processes. Whilst they contain a common catalytic subunit, they have diverse modes of action determined by associated factors in the distinct complexes. The deacetylase module from the NuRD complex contains three protein domains that control the recruitment of chromatin to the deacetylase enzyme, HDAC1/2. Using biochemical approaches and cryo-electron microscopy, we have determined how three chromatin-binding domains (MTA1-BAH, MBD2/3 and RBBP4/7) are assembled in relation to the core complex so as to facilitate interaction of the complex with the genome. We observe a striking arrangement of the BAH domains suggesting a potential mechanism for binding to di-nucleosomes. We also find that the WD40 domains from RBBP4 are linked to the core with surprising flexibility that is likely important for chromatin engagement. A single MBD2 protein binds asymmetrically to the dimerisation interface of the complex. This symmetry mismatch explains the stoichiometry of the complex. Finally, our structures suggest how the holo-NuRD might assemble on a di-nucleosome substrate. 履歴 登録 2020年10月14日 - ヘッダ(付随情報) 公開 2020年11月11日 - マップ公開 2020年11月11日 - 更新 2024年5月1日 - 現状 2024年5月1日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 英国, 3件
英国, 3件  引用
引用 ジャーナル: Nucleic Acids Res / 年: 2020
ジャーナル: Nucleic Acids Res / 年: 2020
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_11838.map.gz
emd_11838.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-11838-v30.xml
emd-11838-v30.xml emd-11838.xml
emd-11838.xml EMDBヘッダ
EMDBヘッダ emd_11838_fsc.xml
emd_11838_fsc.xml FSCデータファイル
FSCデータファイル emd_11838.png
emd_11838.png emd_11838_msk_1.map
emd_11838_msk_1.map マスクマップ
マスクマップ emd-11838.cif.gz
emd-11838.cif.gz emd_11838_half_map_1.map.gz
emd_11838_half_map_1.map.gz emd_11838_half_map_2.map.gz
emd_11838_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-11838
http://ftp.pdbj.org/pub/emdb/structures/EMD-11838 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11838
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11838 emd_11838_validation.pdf.gz
emd_11838_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_11838_full_validation.pdf.gz
emd_11838_full_validation.pdf.gz emd_11838_validation.xml.gz
emd_11838_validation.xml.gz emd_11838_validation.cif.gz
emd_11838_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11838
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11838 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11838
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11838 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_11838.map.gz / 形式: CCP4 / 大きさ: 46.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_11838.map.gz / 形式: CCP4 / 大きさ: 46.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_11838_msk_1.map
emd_11838_msk_1.map 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー





















































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)