+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10676 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human Coronavirus HKU1 Haemagglutinin-Esterase | |||||||||
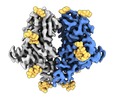
 マップデータ マップデータ | Human coronavirus-HKU1 haemagglutinin esterase (local resolution filtered map). | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sialate 9-O-acetylesterase activity / sialate 4-O-acetylesterase activity / sialate O-acetylesterase activity / sialate O-acetylesterase / membrane => GO:0016020 / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / viral envelope / host cell plasma membrane / virion membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human coronavirus HKU1 (ウイルス) Human coronavirus HKU1 (ウイルス) | |||||||||
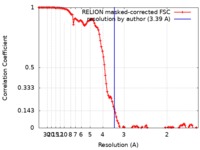
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.39 Å | |||||||||
 データ登録者 データ登録者 | Hurdiss DL / Drulyte I | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Cryo-EM structure of coronavirus-HKU1 haemagglutinin esterase reveals architectural changes arising from prolonged circulation in humans. 著者: Daniel L Hurdiss / Ieva Drulyte / Yifei Lang / Tatiana M Shamorkina / Matti F Pronker / Frank J M van Kuppeveld / Joost Snijder / Raoul J de Groot /  要旨: The human betacoronaviruses HKU1 and OC43 (subgenus Embecovirus) arose from separate zoonotic introductions, OC43 relatively recently and HKU1 apparently much longer ago. Embecovirus particles ...The human betacoronaviruses HKU1 and OC43 (subgenus Embecovirus) arose from separate zoonotic introductions, OC43 relatively recently and HKU1 apparently much longer ago. Embecovirus particles contain two surface projections called spike (S) and haemagglutinin-esterase (HE), with S mediating receptor binding and membrane fusion, and HE acting as a receptor-destroying enzyme. Together, they promote dynamic virion attachment to glycan-based receptors, specifically 9-O-acetylated sialic acid. Here we present the cryo-EM structure of the ~80 kDa, heavily glycosylated HKU1 HE at 3.4 Å resolution. Comparison with existing HE structures reveals a drastically truncated lectin domain, incompatible with sialic acid binding, but with the structure and function of the esterase domain left intact. Cryo-EM and mass spectrometry analysis reveals a putative glycan shield on the now redundant lectin domain. The findings further our insight into the evolution and host adaptation of human embecoviruses, and demonstrate the utility of cryo-EM for studying small, heavily glycosylated proteins. #1:  ジャーナル: Biorxiv / 年: 2020 ジャーナル: Biorxiv / 年: 2020タイトル: Cryo-EM structure of coronavirus-HKU1 haemagglutinin esterase reveals architectural changes arising from prolonged circulation in humans 著者: Hurdiss DL / Drulyte I / Lang Y / Shamorkina TM / Pronker MF / van Kuppeveld FJM / Snijder J / de Groot RJ | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10676.map.gz emd_10676.map.gz | 80.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10676-v30.xml emd-10676-v30.xml emd-10676.xml emd-10676.xml | 27.2 KB 27.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10676_fsc.xml emd_10676_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10676.png emd_10676.png | 161.6 KB | ||
| マスクデータ |  emd_10676_msk_1.map emd_10676_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10676_additional_1.map.gz emd_10676_additional_1.map.gz emd_10676_additional_2.map.gz emd_10676_additional_2.map.gz emd_10676_additional_3.map.gz emd_10676_additional_3.map.gz emd_10676_half_map_1.map.gz emd_10676_half_map_1.map.gz emd_10676_half_map_2.map.gz emd_10676_half_map_2.map.gz | 114.2 MB 96.3 MB 7.3 MB 96.9 MB 96.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10676 http://ftp.pdbj.org/pub/emdb/structures/EMD-10676 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10676 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10676 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10676_validation.pdf.gz emd_10676_validation.pdf.gz | 356.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10676_full_validation.pdf.gz emd_10676_full_validation.pdf.gz | 355.9 KB | 表示 | |
| XML形式データ |  emd_10676_validation.xml.gz emd_10676_validation.xml.gz | 17.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10676 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10676 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10676 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10676 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6y3yMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10390 (タイトル: Cryo-EM structure of coronavirus-HKU1 haemagglutinin esterase EMPIAR-10390 (タイトル: Cryo-EM structure of coronavirus-HKU1 haemagglutinin esteraseData size: 5.0 TB Data #1: Unaligned multi-frame gain-normalised movies of HCoV-HKU1 HE [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10676.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10676.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human coronavirus-HKU1 haemagglutinin esterase (local resolution filtered map). | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.805 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
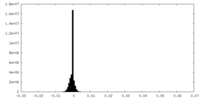
| ファイル |  emd_10676_msk_1.map emd_10676_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
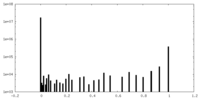
| 密度ヒストグラム |
-追加マップ: Human coronavirus-HKU1 haemagglutinin esterase (Gaussian filtered N-glycan difference...
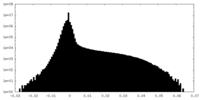
| ファイル | emd_10676_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human coronavirus-HKU1 haemagglutinin esterase (Gaussian filtered N-glycan difference map). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
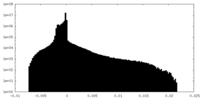
| 密度ヒストグラム |
-追加マップ: Human coronavirus-HKU1 haemagglutinin esterase (unsharpened map).
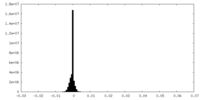
| ファイル | emd_10676_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human coronavirus-HKU1 haemagglutinin esterase (unsharpened map). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
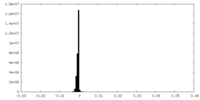
| 密度ヒストグラム |
-追加マップ: Human coronavirus-HKU1 haemagglutinin esterase (sharpened map).
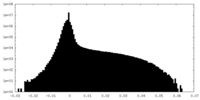
| ファイル | emd_10676_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human coronavirus-HKU1 haemagglutinin esterase (sharpened map). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
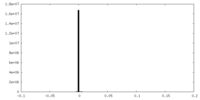
| 密度ヒストグラム |
-ハーフマップ: Human coronavirus-HKU1 haemagglutinin esterase (half map 2).
| ファイル | emd_10676_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human coronavirus-HKU1 haemagglutinin esterase (half map 2). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Human coronavirus-HKU1 haemagglutinin esterase (half map 1).
| ファイル | emd_10676_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human coronavirus-HKU1 haemagglutinin esterase (half map 1). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human Coronavirus HKU1 Haemagglutinin-Esterase
| 全体 | 名称: Human Coronavirus HKU1 Haemagglutinin-Esterase |
|---|---|
| 要素 |
|
-超分子 #1: Human Coronavirus HKU1 Haemagglutinin-Esterase
| 超分子 | 名称: Human Coronavirus HKU1 Haemagglutinin-Esterase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: Dimeric complex |
|---|---|
| 由来(天然) | 生物種:  Human coronavirus HKU1 (ウイルス) Human coronavirus HKU1 (ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 実験値: 30.777 KDa |
-分子 #1: Hemagglutinin-esterase
| 分子 | 名称: Hemagglutinin-esterase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: sialate O-acetylesterase |
|---|---|
| 由来(天然) | 生物種:  Human coronavirus HKU1 (ウイルス) Human coronavirus HKU1 (ウイルス) |
| 分子量 | 理論値: 40.432648 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LAFNEPLNVV SHLNHDWFLF GDSRSDCNHI NNLKIKNFDY LDIHPSLCNN GKISSSAGDS IFKSFHFTRF YNYTGEGDQI IFYEGVNFN PYHRFKCFPN GSNDVWLLNK VRFYRALYSN MAFFRYLTFV DIPYNVSLSK FNSCKSDILS LNNPIFINYS K EVYFTLLG ...文字列: LAFNEPLNVV SHLNHDWFLF GDSRSDCNHI NNLKIKNFDY LDIHPSLCNN GKISSSAGDS IFKSFHFTRF YNYTGEGDQI IFYEGVNFN PYHRFKCFPN GSNDVWLLNK VRFYRALYSN MAFFRYLTFV DIPYNVSLSK FNSCKSDILS LNNPIFINYS K EVYFTLLG CSLYLVPLCL FKSNFSQYYY NIDTGSVYGF SNVVYPDLDC IYISLKPGSY KVSTTAPFLS LPTKALCFDK SK QFVPVQV VDSRWNNERA SDISLSVACQ LPYCYFRNSS ANYVGKYDIN HGDSGFISIL SGLLYNVSCI SYYGVFLYDN FTS IWPYYS FGRCPTSSII KHPICVYDSD PLVPR |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 詳細: 20 mA | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot force = 1 Blot time = 5.5 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 詳細 | Titan Krios G4 was used with fringe-free imaging and aberration-free image shift. |
| 撮影 | フィルム・検出器のモデル: OTHER / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / 撮影したグリッド数: 1 / 実像数: 6029 / 平均露光時間: 5.6 sec. / 平均電子線量: 40.0 e/Å2 / 詳細: Falcon 4 Direct Electron Detector |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.5 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | A homology model of HCOV-HKU1 HE (uniprot ID: Q5MQD1) was generated using the phyre2 server |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
| 得られたモデル |  PDB-6y3y: |
 ムービー
ムービー コントローラー
コントローラー



















 Z
Z Y
Y X
X