+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0476 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of NLRP3 bound to NEK7 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Inflammasome / Activator / Biological process Immunity / Inflammatory response / Innate immunity / Transcription / Transcription regulation Ligand / ATP binding / Nucleotide binding / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報NEK6-subfamily protein kinase / Inhibition of PKR / regulation of NLRP3 inflammasome complex assembly / detection of biotic stimulus / molecular sensor activity / eukaryotic translation initiation factor 2alpha kinase activity / response to interferon-alpha / phosphatidylinositol phosphate binding / negative regulation of osteoblast proliferation / positive regulation of T-helper 2 cell differentiation ...NEK6-subfamily protein kinase / Inhibition of PKR / regulation of NLRP3 inflammasome complex assembly / detection of biotic stimulus / molecular sensor activity / eukaryotic translation initiation factor 2alpha kinase activity / response to interferon-alpha / phosphatidylinositol phosphate binding / negative regulation of osteoblast proliferation / positive regulation of T-helper 2 cell differentiation / Activation of NIMA Kinases NEK9, NEK6, NEK7 / positive regulation of T-helper 2 cell cytokine production / regulation of hematopoietic progenitor cell differentiation / interphase microtubule organizing center / positive regulation of stress-activated MAPK cascade / positive regulation of type 2 immune response / NLRP3 inflammasome complex assembly / NLRP3 inflammasome complex / Nuclear Pore Complex (NPC) Disassembly / peptidoglycan binding / protein phosphatase regulator activity / cysteine-type endopeptidase activator activity / SUMOylation of immune response proteins / osmosensory signaling pathway / regulation of hematopoietic stem cell proliferation / phosphatidylinositol-4-phosphate binding / cellular response to potassium ion / negative regulation of non-canonical NF-kappaB signal transduction / regulation of hematopoietic stem cell differentiation / pattern recognition receptor signaling pathway / regulation of translational initiation / negative regulation of interleukin-1 beta production / negative regulation of viral genome replication / positive regulation of interleukin-4 production / negative regulation of acute inflammatory response / positive regulation of NLRP3 inflammasome complex assembly / positive regulation of telomere maintenance / microtubule organizing center / pyroptotic inflammatory response / The NLRP3 inflammasome / endoplasmic reticulum unfolded protein response / Purinergic signaling in leishmaniasis infection / positive regulation of chemokine production / spindle assembly / signaling adaptor activity / antiviral innate immune response / EML4 and NUDC in mitotic spindle formation / regulation of mitotic cell cycle / molecular function activator activity / cellular response to amino acid starvation / positive regulation of cytokine production / positive regulation of interleukin-1 beta production / non-membrane spanning protein tyrosine kinase activity / protein maturation / non-specific protein-tyrosine kinase / molecular condensate scaffold activity / defense response / Cytoprotection by HMOX1 / positive regulation of non-canonical NF-kappaB signal transduction / : / ADP binding / PKR-mediated signaling / protein homooligomerization / cellular response to virus / Evasion by RSV of host interferon responses / negative regulation of inflammatory response / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / ISG15 antiviral mechanism / response to virus / Metalloprotease DUBs / spindle pole / positive regulation of inflammatory response / SARS-CoV-1 activates/modulates innate immune responses / kinase activity / Interferon alpha/beta signaling / double-stranded RNA binding / protein autophosphorylation / regulation of inflammatory response / cellular response to lipopolysaccharide / protein-macromolecule adaptor activity / defense response to virus / sequence-specific DNA binding / DNA-binding transcription factor binding / molecular adaptor activity / microtubule / protein phosphorylation / protein kinase activity / non-specific serine/threonine protein kinase / positive regulation of MAPK cascade / negative regulation of translation / ribosome / translation / inflammatory response / Golgi membrane / negative regulation of cell population proliferation / innate immune response / protein serine kinase activity / protein serine/threonine kinase activity / apoptotic process / centrosome 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
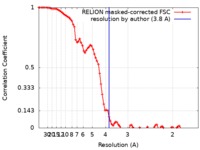
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Sharif H / Wang L / Wu H | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: Structural mechanism for NEK7-licensed activation of NLRP3 inflammasome. 著者: Humayun Sharif / Li Wang / Wei Li Wang / Venkat Giri Magupalli / Liudmila Andreeva / Qi Qiao / Arthur V Hauenstein / Zhaolong Wu / Gabriel Núñez / Youdong Mao / Hao Wu /   要旨: The NLRP3 inflammasome can be activated by stimuli that include nigericin, uric acid crystals, amyloid-β fibrils and extracellular ATP. The mitotic kinase NEK7 licenses the assembly and activation ...The NLRP3 inflammasome can be activated by stimuli that include nigericin, uric acid crystals, amyloid-β fibrils and extracellular ATP. The mitotic kinase NEK7 licenses the assembly and activation of the NLRP3 inflammasome in interphase. Here we report a cryo-electron microscopy structure of inactive human NLRP3 in complex with NEK7, at a resolution of 3.8 Å. The earring-shaped NLRP3 consists of curved leucine-rich-repeat and globular NACHT domains, and the C-terminal lobe of NEK7 nestles against both NLRP3 domains. Structural recognition between NLRP3 and NEK7 is confirmed by mutagenesis both in vitro and in cells. Modelling of an active NLRP3-NEK7 conformation based on the NLRC4 inflammasome predicts an additional contact between an NLRP3-bound NEK7 and a neighbouring NLRP3. Mutations to this interface abolish the ability of NEK7 or NLRP3 to rescue NLRP3 activation in NEK7-knockout or NLRP3-knockout cells. These data suggest that NEK7 bridges adjacent NLRP3 subunits with bipartite interactions to mediate the activation of the NLRP3 inflammasome. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0476.map.gz emd_0476.map.gz | 48.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0476-v30.xml emd-0476-v30.xml emd-0476.xml emd-0476.xml | 34.1 KB 34.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0476_fsc.xml emd_0476_fsc.xml | 8.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0476.png emd_0476.png | 150.3 KB | ||
| Filedesc metadata |  emd-0476.cif.gz emd-0476.cif.gz | 9.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0476 http://ftp.pdbj.org/pub/emdb/structures/EMD-0476 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0476 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0476 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0476.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0476.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : NL3-NEK7
| 全体 | 名称: NL3-NEK7 |
|---|---|
| 要素 |
|
-超分子 #1: NL3-NEK7
| 超分子 | 名称: NL3-NEK7 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|
-超分子 #2: NLRP3
| 超分子 | 名称: NLRP3 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2 詳細: The sequence is according to NP_001230062.1 isoform which lacks 2 residues from N-terminus. Total residues are 1034aa as compared to 1036 in UNP Q96P20. There are mutations in YRKKYRKY ...詳細: The sequence is according to NP_001230062.1 isoform which lacks 2 residues from N-terminus. Total residues are 1034aa as compared to 1036 in UNP Q96P20. There are mutations in YRKKYRKY instead its IYCAKYRAY mutations were intended to introduce so that the class of MBP and NLRP3 is avoided |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: NEK7
| 超分子 | 名称: NEK7 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Interferon-induced, double-stranded RNA-activated protein kinase,...
| 分子 | 名称: Interferon-induced, double-stranded RNA-activated protein kinase,Serine/threonine-protein kinase Nek7 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: non-specific serine/threonine protein kinase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 32.01515 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSTDKRFGMD FRIEKKIGRG QFSEVYRAAC RLDGKTVALK KVQIFDLMDA KARADCIKEI DLLAQLNHPN VIKYYVCFIT GNELNIVLE LADAGDLSRM IKHFKKQKRL IPERTVWKYF VQLCSALEHM HSRRVMHRDI KPANVFITAT GVVKLGDLGL G RFFSSKTT ...文字列: GSTDKRFGMD FRIEKKIGRG QFSEVYRAAC RLDGKTVALK KVQIFDLMDA KARADCIKEI DLLAQLNHPN VIKYYVCFIT GNELNIVLE LADAGDLSRM IKHFKKQKRL IPERTVWKYF VQLCSALEHM HSRRVMHRDI KPANVFITAT GVVKLGDLGL G RFFSSKTT AAHSLVGTPY YMSPERIHEN GYNFKSDIWS LGCLLYEMAA LQSPFYGDKM NLYSLCKKIE QCDYPPLPSD HY SEELRQL VNMCINPDPE KRPDVTYVYD VAKRMHACTA SS UniProtKB: Interferon-induced, double-stranded RNA-activated protein kinase, Serine/threonine-protein kinase Nek7 |
-分子 #2: Isoform 2 of NACHT, LRR and PYD domains-containing protein 3
| 分子 | 名称: Isoform 2 of NACHT, LRR and PYD domains-containing protein 3 タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 117.890984 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASTRCKLAR YLEDLEDVDL KKFKMHLEDY PPQKGCIPLP RGQTEKADHV DLATLMIDFN GEEKAWAMAV WIFAAINRRD LYEKAKRDE PKWGSDNARV SNPTVICQED SIEEEWMGLL EYLSRISICK MKKIYCAKYR AYVRSRFQCI EDRNARLGES V SLNKRYTR ...文字列: MASTRCKLAR YLEDLEDVDL KKFKMHLEDY PPQKGCIPLP RGQTEKADHV DLATLMIDFN GEEKAWAMAV WIFAAINRRD LYEKAKRDE PKWGSDNARV SNPTVICQED SIEEEWMGLL EYLSRISICK MKKIYCAKYR AYVRSRFQCI EDRNARLGES V SLNKRYTR LRLIKEHRSQ QEREQELLAI GKTKTCESPV SPIKMELLFD PDDEHSEPVH TVVFQGAAGI GKTILARKMM LD WASGTLY QDRFDYLFYI HCREVSLVTQ RSLGDLIMSC CPDPNPPIHK IVRKPSRILF LMDGFDELQG AFDEHIGPLC TDW QKAERG DILLSSLIRK KLLPEASLLI TTRPVALEKL QHLLDHPRHV EILGFSEAKR KEYFFKYFSD EAQARAAFSL IQEN EVLFT MCFIPLVCWI VCTGLKQQME SGKSLAQTSK TTTAVYVFFL SSLLQPRGGS QEHGLCAHLW GLCSLAADGI WNQKI LFEE SDLRNHGLQK ADVSAFLRMN LFQKEVDCEK FYSFIHMTFQ EFFAAMYYLL EEEKEGRTNV PGSRLKLPSR DVTVLL ENY GKFEKGYLIF VVRFLFGLVN QERTSYLEKK LSCKISQQIR LELLKWIEVK AKAKKLQIQP SQLELFYCLY EMQEEDF VQ RAMDYFPKIE INLSTRMDHM VSSFCIENCH RVESLSLGFL HNMPKEEEEE EKEGRHLDMV QCVLPSSSHA ACSHGLVN S HLTSSFCRGL FSVLSTSQSL TELDLSDNSL GDPGMRVLCE TLQHPGCNIR RLWLGRCGLS HECCFDISLV LSSNQKLVE LDLSDNALGD FGIRLLCVGL KHLLCNLKKL WLVSCCLTSA CCQDLASVLS TSHSLTRLYV GENALGDSGV AILCEKAKNP QCNLQKLGL VNSGLTSVCC SALSSVLSTN QNLTHLYLRG NTLGDKGIKL LCEGLLHPDC KLQVLELDNC NLTSHCCWDL S TLLTSSQS LRKLSLGNND LGDLGVMMFC EVLKQQSCLL QNLGLSEMYF NYETKSALET LQEEKPELTV VFEPSW UniProtKB: NACHT, LRR and PYD domains-containing protein 3 |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.1 K / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TITAN THEMIS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-6npy: |
 ムービー
ムービー コントローラー
コントローラー




























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)