+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0345 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Metabotropic Glutamate Receptor 5 bound to L-quisqualate and Nb43 | |||||||||
 マップデータ マップデータ | Metabotropic Glutamate Receptor 5 bound to L-quisqualate and Nb43, primary map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報A2A adenosine receptor binding / phospholipase C-activating G protein-coupled glutamate receptor signaling pathway / G protein-coupled receptor activity involved in regulation of postsynaptic membrane potential / trans-synaptic signaling by endocannabinoid, modulating synaptic transmission / adenylate cyclase inhibiting G protein-coupled glutamate receptor activity / positive regulation of long-term neuronal synaptic plasticity / desensitization of G protein-coupled receptor signaling pathway / neurotransmitter receptor activity involved in regulation of postsynaptic cytosolic calcium ion concentration / G protein-coupled glutamate receptor signaling pathway / astrocyte projection ...A2A adenosine receptor binding / phospholipase C-activating G protein-coupled glutamate receptor signaling pathway / G protein-coupled receptor activity involved in regulation of postsynaptic membrane potential / trans-synaptic signaling by endocannabinoid, modulating synaptic transmission / adenylate cyclase inhibiting G protein-coupled glutamate receptor activity / positive regulation of long-term neuronal synaptic plasticity / desensitization of G protein-coupled receptor signaling pathway / neurotransmitter receptor activity involved in regulation of postsynaptic cytosolic calcium ion concentration / G protein-coupled glutamate receptor signaling pathway / astrocyte projection / Class C/3 (Metabotropic glutamate/pheromone receptors) / : / glutamate receptor activity / Neurexins and neuroligins / protein tyrosine kinase activator activity / : / regulation of synaptic transmission, glutamatergic / protein tyrosine kinase binding / dendritic shaft / learning / locomotory behavior / G protein-coupled receptor activity / postsynaptic density membrane / synapse organization / Schaffer collateral - CA1 synapse / cognition / cellular response to amyloid-beta / G alpha (q) signalling events / chemical synaptic transmission / positive regulation of MAPK cascade / dendritic spine / learning or memory / glutamatergic synapse / dendrite / regulation of DNA-templated transcription / identical protein binding / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Koehl A / Hu H / Feng D / Sun B / Weis WI / Skiniotis GS / Mathiesen JM / Kobilka BK | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: Structural insights into the activation of metabotropic glutamate receptors. 著者: Antoine Koehl / Hongli Hu / Dan Feng / Bingfa Sun / Yan Zhang / Michael J Robertson / Matthew Chu / Tong Sun Kobilka / Toon Laeremans / Jan Steyaert / Jeffrey Tarrasch / Somnath Dutta / ...著者: Antoine Koehl / Hongli Hu / Dan Feng / Bingfa Sun / Yan Zhang / Michael J Robertson / Matthew Chu / Tong Sun Kobilka / Toon Laeremans / Jan Steyaert / Jeffrey Tarrasch / Somnath Dutta / Rasmus Fonseca / William I Weis / Jesper M Mathiesen / Georgios Skiniotis / Brian K Kobilka /     要旨: Metabotropic glutamate receptors are family C G-protein-coupled receptors. They form obligate dimers and possess extracellular ligand-binding Venus flytrap domains, which are linked by cysteine-rich ...Metabotropic glutamate receptors are family C G-protein-coupled receptors. They form obligate dimers and possess extracellular ligand-binding Venus flytrap domains, which are linked by cysteine-rich domains to their 7-transmembrane domains. Spectroscopic studies show that signalling is a dynamic process, in which large-scale conformational changes underlie the transmission of signals from the extracellular Venus flytraps to the G protein-coupling domains-the 7-transmembrane domains-in the membrane. Here, using a combination of X-ray crystallography, cryo-electron microscopy and signalling studies, we present a structural framework for the activation mechanism of metabotropic glutamate receptor subtype 5. Our results show that agonist binding at the Venus flytraps leads to a compaction of the intersubunit dimer interface, thereby bringing the cysteine-rich domains into close proximity. Interactions between the cysteine-rich domains and the second extracellular loops of the receptor enable the rigid-body repositioning of the 7-transmembrane domains, which come into contact with each other to initiate signalling. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0345.map.gz emd_0345.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0345-v30.xml emd-0345-v30.xml emd-0345.xml emd-0345.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0345.png emd_0345.png | 90.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0345 http://ftp.pdbj.org/pub/emdb/structures/EMD-0345 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0345 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0345 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0345_validation.pdf.gz emd_0345_validation.pdf.gz | 472.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0345_full_validation.pdf.gz emd_0345_full_validation.pdf.gz | 472.3 KB | 表示 | |
| XML形式データ |  emd_0345_validation.xml.gz emd_0345_validation.xml.gz | 6 KB | 表示 | |
| CIF形式データ |  emd_0345_validation.cif.gz emd_0345_validation.cif.gz | 6.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0345 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0345 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0345 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0345 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0345.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0345.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Metabotropic Glutamate Receptor 5 bound to L-quisqualate and Nb43, primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex between metabotropic glutamate receptor 5 bound to L-quis...
| 全体 | 名称: Complex between metabotropic glutamate receptor 5 bound to L-quisqualate and Nb43 |
|---|---|
| 要素 |
|
-超分子 #1: Complex between metabotropic glutamate receptor 5 bound to L-quis...
| 超分子 | 名称: Complex between metabotropic glutamate receptor 5 bound to L-quisqualate and Nb43 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 220 KDa |
-分子 #1: Metabotropic glutamate receptor 5
| 分子 | 名称: Metabotropic glutamate receptor 5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 90.484805 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SERRVVAHMP GDIIIGALFS VHHQPTVDKV HERKCGAVRE QYGIQRVEAM LHTLERINSD PTLLPNITLG CEIRDSCWHS AVALEQSIE FIRDSLISSE EEEGLVRCVD GSSSSFRSKK PIVGVIGPGS SSVAIQVQNL LQLFNIPQIA YSATSMDLSD K TLFKYFMR ...文字列: SERRVVAHMP GDIIIGALFS VHHQPTVDKV HERKCGAVRE QYGIQRVEAM LHTLERINSD PTLLPNITLG CEIRDSCWHS AVALEQSIE FIRDSLISSE EEEGLVRCVD GSSSSFRSKK PIVGVIGPGS SSVAIQVQNL LQLFNIPQIA YSATSMDLSD K TLFKYFMR VVPSDAQQAR AMVDIVKRYN WTYVSAVHTE GNYGESGMEA FKDMSAKEGI CIAHSYKIYS NAGEQSFDKL LK KLTSHLP KARVVACFCE GMTVRGLLMA MRRLGLAGEF LLLGSDGWAD RYDVTDGYQR EAVGGITIKL QSPDVKWFDD YYL KLRPET NHRNPWFQEF WQHRFQCRLE GFPQENSKYN KTCNSSLTLK THHVQDSKMG FVINAIYSMA YGLHNMQMSL CPGY AGLCD AMKPIDGRKL LESLMKTNFT GVSGDTILFD ENGDSPGRYE IMNFKEMGKD YFDYINVGSW DNGELKMDDD EVWSK KSNI IRSVCSEPCE KGQIKVIRKG EVSCCWTCTP CKENEYVFDE YTCKACQLGS WPTDDLTGCD LIPVQYLRWG DPEPIA AVV FACLGLLATL FVTVVFIIYR DTPVVKSSSR ELCYIILAGI CLGYLCTFCL IAKPKQIYCY LQRIGIGLSP AMSYSAL VT KTNRIARILA GSKKKICTKK PRFMSACAQL VIAFILICIQ LGIIVALFIM EPPDIMHDYP SIREVYLICN TTNLGVVT P LGYNGLLILS CTFYAFKTRN VPANFNEAKY IAFTMYTTCI IWLAFVPIYF GSNYKIITMC FSVSLSATVA LGCMFVPKV YIIL |
-分子 #2: Nanobody 43
| 分子 | 名称: Nanobody 43 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.354672 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLVESGGG LVQAGGSLRL SCAASGRTFT SYAMGWFRQA PGKERESVAA ISSSGGSTHY ADSVKGRFTI SRDNSKNTVY LQMNSLKPE DTAVYYCAAA MYGSRWPDWE YDYWGQGTQV TVSS |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 4 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #4: (S)-2-AMINO-3-(3,5-DIOXO-[1,2,4]OXADIAZOLIDIN-2-YL)-PROPIONIC ACID
| 分子 | 名称: (S)-2-AMINO-3-(3,5-DIOXO-[1,2,4]OXADIAZOLIDIN-2-YL)-PROPIONIC ACID タイプ: ligand / ID: 4 / コピー数: 2 / 式: QUS |
|---|---|
| 分子量 | 理論値: 189.126 Da |
| Chemical component information | 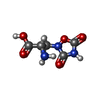 ChemComp-QUS: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 12 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 390.0 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV 詳細: additional 0.1%OG was added to the sample right before applying sample to grid; 3.5ul sample was applied; blot for 1 second before plunging;. | |||||||||||||||
| 詳細 | Sample was mono disperse as assayed by size exclusion chromatography |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 8.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 47169 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Geometry |
|---|---|
| 得られたモデル |  PDB-6n51: |
 ムービー
ムービー コントローラー
コントローラー



















