+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0172 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the archaeal extremophilic internal membrane containing Haloarcula hispanica icosahedral virus 2 (HHIV-2) at 3.78 Angstroms resolution. | |||||||||||||||
 マップデータ マップデータ | Map derived from RELION postprocessed map. The map was then centred at 0,0,0 and finally used for model refinement. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | single vertical beta-barrel virus / archaeal / membrane-containing / quasi-atomic resolution / VIRUS | |||||||||||||||
| 機能・相同性 | VP7 / VP4 / Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Haloarcula hispanica icosahedral virus 2 (ウイルス) Haloarcula hispanica icosahedral virus 2 (ウイルス) | |||||||||||||||
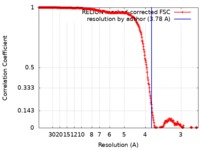
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.78 Å | |||||||||||||||
 データ登録者 データ登録者 | Abrescia NG / Santos-Perez I | |||||||||||||||
| 資金援助 |  スペイン, スペイン,  フィンランド, 4件 フィンランド, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2015 ジャーナル: Structure / 年: 2015タイトル: Insight into the Assembly of Viruses with Vertical Single β-barrel Major Capsid Proteins. 著者: David Gil-Carton / Salla T Jaakkola / Diego Charro / Bibiana Peralta / Daniel Castaño-Díez / Hanna M Oksanen / Dennis H Bamford / Nicola G A Abrescia /    要旨: Archaeal viruses constitute the least explored niche within the virosphere. Structure-based approaches have revealed close relationships between viruses infecting organisms from different domains of ...Archaeal viruses constitute the least explored niche within the virosphere. Structure-based approaches have revealed close relationships between viruses infecting organisms from different domains of life. Here, using biochemical and cryo-electron microscopy techniques, we solved the structure of euryarchaeal, halophilic, internal membrane-containing Haloarcula hispanica icosahedral virus 2 (HHIV-2). We show that the density of the two major capsid proteins (MCPs) recapitulates vertical single β-barrel proteins and that disulfide bridges stabilize the capsid. Below, ordered density is visible close to the membrane and at the five-fold vertices underneath the host-interacting vertex complex underpinning membrane-protein interactions. The HHIV-2 structure exemplifies the division of conserved architectural elements of a virion, such as the capsid, from those that evolve rapidly due to selective environmental pressure such as host-recognizing structures. We propose that in viruses with two vertical single β-barrel MCPs the vesicle is indispensable, and membrane-protein interactions serve as protein-railings for guiding the assembly. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0172.map.gz emd_0172.map.gz | 1.5 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0172-v30.xml emd-0172-v30.xml emd-0172.xml emd-0172.xml | 29.9 KB 29.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0172_fsc.xml emd_0172_fsc.xml | 26.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0172.png emd_0172.png | 219.2 KB | ||
| マスクデータ |  emd_0172_msk_1.map emd_0172_msk_1.map | 1.6 GB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-0172.cif.gz emd-0172.cif.gz | 6.8 KB | ||
| その他 |  emd_0172_additional.map.gz emd_0172_additional.map.gz emd_0172_half_map_1.map.gz emd_0172_half_map_1.map.gz emd_0172_half_map_2.map.gz emd_0172_half_map_2.map.gz | 1.5 GB 1.3 GB 1.3 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0172 http://ftp.pdbj.org/pub/emdb/structures/EMD-0172 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0172 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0172 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0172_validation.pdf.gz emd_0172_validation.pdf.gz | 433.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0172_full_validation.pdf.gz emd_0172_full_validation.pdf.gz | 432.2 KB | 表示 | |
| XML形式データ |  emd_0172_validation.xml.gz emd_0172_validation.xml.gz | 29 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0172 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0172 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0172 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0172 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0172.map.gz / 形式: CCP4 / 大きさ: 1.6 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0172.map.gz / 形式: CCP4 / 大きさ: 1.6 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map derived from RELION postprocessed map. The map was then centred at 0,0,0 and finally used for model refinement. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0172_msk_1.map emd_0172_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
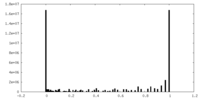
| 密度ヒストグラム |
-追加マップ: Relion post-processed map bfactor -40
| ファイル | emd_0172_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion post-processed map bfactor -40 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
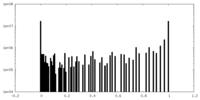
| 密度ヒストグラム |
-ハーフマップ: Half maps for gold-standard FSC and generation of...
| ファイル | emd_0172_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half maps for gold-standard FSC and generation of both submitted postprocessed maps. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
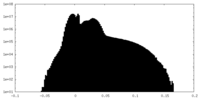
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_0172_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
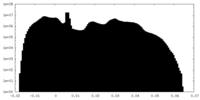
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Haloarcula hispanica icosahedral virus 2
| 全体 | 名称:  Haloarcula hispanica icosahedral virus 2 (ウイルス) Haloarcula hispanica icosahedral virus 2 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Haloarcula hispanica icosahedral virus 2
| 超分子 | 名称: Haloarcula hispanica icosahedral virus 2 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 1154689 / 生物種: Haloarcula hispanica icosahedral virus 2 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Haloarcula hispanica ATCC 33960 (好塩性) Haloarcula hispanica ATCC 33960 (好塩性) |
-分子 #1: VP4
| 分子 | 名称: VP4 / タイプ: protein_or_peptide / ID: 1 / 詳細: http://mit.cicbiogune.int:39000/projects/P2/W1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica icosahedral virus 2 (ウイルス) Haloarcula hispanica icosahedral virus 2 (ウイルス) |
| 分子量 | 理論値: 25.585746 KDa |
| 配列 | 文字列: QTQEYTINHT GGVLGDSYVT TASNQTSPQR ETAVLSFECP RKFEEINYVG QRDATRFVPR TTESITGSAN DDTVVDLTAN IQPVAGEEV IAEQDYPVAV AYNVTQGVEV DVVDADYAAD TVTLGTNPAD GDEVKVWPIM SDGDVQFRLI NQFGQEEGRV Y PWSTPLYR ...文字列: QTQEYTINHT GGVLGDSYVT TASNQTSPQR ETAVLSFECP RKFEEINYVG QRDATRFVPR TTESITGSAN DDTVVDLTAN IQPVAGEEV IAEQDYPVAV AYNVTQGVEV DVVDADYAAD TVTLGTNPAD GDEVKVWPIM SDGDVQFRLI NQFGQEEGRV Y PWSTPLYR WHDFPQLKRG REINLHGSAS WSENETLEIL LDAPQALTWE DSDYPRGQYV TTLEQDVEIT L UniProtKB: VP4 |
-分子 #2: VP7
| 分子 | 名称: VP7 / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica icosahedral virus 2 (ウイルス) Haloarcula hispanica icosahedral virus 2 (ウイルス) |
| 分子量 | 理論値: 18.473355 KDa |
| 配列 | 文字列: PEIGNNGAEK QISLHKGQPF IDTQDVGAAD PNTPAVTIEG PSDYVIAIDA GTPVAPEFRD ANGDKLDPST RVTIQKCDKQ GNPLGDGIV FSDTLGRFEY SKMRSDPDYM RKTTTSLMID EREIVKIFVE VPPNANGMDA DNSRITIGDD TSDYGKAVGI V EHGDLSPA ESKA UniProtKB: VP7 |
-分子 #3: VP7
| 分子 | 名称: VP7 / タイプ: protein_or_peptide / ID: 3 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica icosahedral virus 2 (ウイルス) Haloarcula hispanica icosahedral virus 2 (ウイルス) |
| 分子量 | 理論値: 18.857811 KDa |
| 配列 | 文字列: PEIGNNGAEK QISLHKGQPF IDTQDVGAAD PNTPAVTIEG PSDYVIAIDA GTPVAPEFRD ANGDKLDPST RVTIQKCDKQ GNPLGDGIV FSDTLGRFEY SKMRSDPDYM RKTTTSLMID EREIVKIFVE VPPNANGMDA DNSRITIGDD TSDYGKAVGI V EHGDLSPA ESKAVRQ UniProtKB: VP7 |
-分子 #4: VP7
| 分子 | 名称: VP7 / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica icosahedral virus 2 (ウイルス) Haloarcula hispanica icosahedral virus 2 (ウイルス) |
| 分子量 | 理論値: 17.347154 KDa |
| 配列 | 文字列: IGNNGAEKQI SLHKGQPFID TQDVGAADPN TPAVTIEGPS DYVIAIDAGT PVAPEFRDAN GDKLDPSTRV TIQKCDKQGN PLGDGIVFS DTLGRFEYSK MRSDPDYMRK TTTSLMIDER EIVKIFVEVP PNANGMDADN SRITIGDDTS DYGKAVGIVE H G UniProtKB: VP7 |
-分子 #5: Uncharacterized protein
| 分子 | 名称: Uncharacterized protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica icosahedral virus 2 (ウイルス) Haloarcula hispanica icosahedral virus 2 (ウイルス) |
| 分子量 | 理論値: 14.206529 KDa |
| 配列 | 文字列: QTADGRVGLV PVNSYVTLET DDLDTDEHPV TDAGTVALEP GESAPIVRYD LGQPAAVYAV GATDEANVEY ELKVNNSKTV GGRTNSPLG VLNTPFSFVE KLGGAIPCET AATYWAHYSS DATGTVELAG RMHIEV UniProtKB: Uncharacterized protein |
-分子 #6: VP16 (vertex complex)
| 分子 | 名称: VP16 (vertex complex) / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica icosahedral virus 2 (ウイルス) Haloarcula hispanica icosahedral virus 2 (ウイルス) |
| 分子量 | 理論値: 18.485723 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #7: GPS III
| 分子 | 名称: GPS III / タイプ: protein_or_peptide / ID: 7 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica icosahedral virus 2 (ウイルス) Haloarcula hispanica icosahedral virus 2 (ウイルス) |
| 分子量 | 理論値: 8.954028 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) |
-分子 #8: polypeptide stretch (vertex complex)
| 分子 | 名称: polypeptide stretch (vertex complex) / タイプ: protein_or_peptide / ID: 8 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloarcula hispanica icosahedral virus 2 (ウイルス) Haloarcula hispanica icosahedral virus 2 (ウイルス) |
| 分子量 | 理論値: 2.145636 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.2 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 | |||||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X