+検索条件
-Structure paper
| タイトル | Tubulin code eraser CCP5 binds branch glutamates by substrate deformation |
|---|---|
| ジャーナル・号・ページ | Nature, Year 2024 |
| 掲載日 | 2023年11月28日 (構造データの登録日) |
 著者 著者 | Chen J / Zehr EA / Gruschus JM / Szyk A / Liu Y / Tanner ME / Tjandra N / Roll-Mecak A |
 リンク リンク |  Nature / PubMedで検索 Nature / PubMedで検索 |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 1.8 - 3.6 Å |
| 構造データ | EMDB-42950, PDB-8v3q: EMDB-42951, PDB-8v3r: EMDB-42952, PDB-8v3s: EMDB-42971, PDB-8v4k: EMDB-42972, PDB-8v4l: EMDB-42973, PDB-8v4m:  PDB-8v3m:  PDB-8v3n:  PDB-8v3o:  PDB-8v3p: |
| 化合物 | 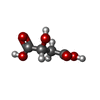 ChemComp-MLT:  ChemComp-IMD:  ChemComp-ZN:  ChemComp-HOH:  PDB-1aag:  ChemComp-K:  ChemComp-GLU:  ChemComp-MG:  ChemComp-GTP: 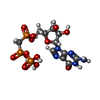 ChemComp-G2P: |
| 由来 |
|
 キーワード キーワード | HYDROLASE / carboxypeptidase deglutamylation branch glutamate removal microtubule / HYDROLASE/INHIBITOR / HYDROLASE-INHIBITOR complex / carboxypeptidase / deglutamylation / branch glutamate removal / microtubule / HYDROLASE/SUBSTRATE / HYDROLASE-SUBSTRATE complex / STRUCTURAL PROTEIN / HYDROLASE-SUBSTRATE / STRUCTURAL PROTEIN complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について















 homo sapiens (ヒト)
homo sapiens (ヒト)
