+検索条件
-Structure paper
| タイトル | In transcription antitermination by Qλ, NusA induces refolding of Qλ to form a nozzle that extends the RNA polymerase RNA-exit channel. |
|---|---|
| ジャーナル・号・ページ | Proc Natl Acad Sci U S A, Vol. 119, Issue 33, Page e2205278119, Year 2022 |
| 掲載日 | 2022年8月16日 |
 著者 著者 | Zhou Yin / Jeremy G Bird / Jason T Kaelber / Bryce E Nickels / Richard H Ebright /  |
| PubMed 要旨 | Lambdoid bacteriophage Q proteins are transcription antipausing and antitermination factors that enable RNA polymerase (RNAP) to read through pause and termination sites. Q proteins load onto RNAP ...Lambdoid bacteriophage Q proteins are transcription antipausing and antitermination factors that enable RNA polymerase (RNAP) to read through pause and termination sites. Q proteins load onto RNAP engaged in promoter-proximal pausing at a Q binding element (QBE) and adjacent sigma-dependent pause element to yield a Q-loading complex, and they translocate with RNAP as a pausing-deficient, termination-deficient Q-loaded complex. In previous work, we showed that the Q protein of bacteriophage 21 (Q21) functions by forming a nozzle that narrows and extends the RNAP RNA-exit channel, preventing formation of pause and termination RNA hairpins. Here, we report atomic structures of four states on the pathway of antitermination by the Q protein of bacteriophage λ (Qλ), a Q protein that shows no sequence similarity to Q21 and that, unlike Q21, requires the transcription elongation factor NusA for efficient antipausing and antitermination. We report structures of Qλ, the Qλ-QBE complex, the NusA-free pre-engaged Qλ-loading complex, and the NusA-containing engaged Qλ-loading complex. The results show that Qλ, like Q21, forms a nozzle that narrows and extends the RNAP RNA-exit channel, preventing formation of RNA hairpins. However, the results show that Qλ has no three-dimensional structural similarity to Q21, employs a different mechanism of QBE recognition than Q21, and employs a more complex process for loading onto RNAP than Q21, involving recruitment of Qλ to form a pre-engaged loading complex, followed by NusA-facilitated refolding of Qλ to form an engaged loading complex. The results establish that Qλ and Q21 are not structural homologs and are solely functional analogs. |
 リンク リンク |  Proc Natl Acad Sci U S A / Proc Natl Acad Sci U S A /  PubMed:35951650 / PubMed:35951650 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 1.46 - 3.36 Å |
| 構造データ | EMDB-26438, PDB-7ubm: EMDB-26439, PDB-7ubn:  PDB-7ubj:  PDB-7ubk: 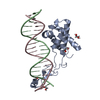 PDB-7ubl: |
| 化合物 |  ChemComp-ZN:  ChemComp-CL:  ChemComp-GOL:  ChemComp-EDO:  ChemComp-PEG:  ChemComp-PO4:  ChemComp-HOH:  ChemComp-MG: |
| 由来 |
|
 キーワード キーワード | GENE REGULATION / RNA polymerase / DNA Binding / transcription / Q-dependent antitermination / Q antitermination factor / GENE REGULATION/DNA / GENE REGULATION-DNA complex / TRANSFERASE/DNA/RNA / TRANSFERASE-DNA-RNA complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について








 escherichia phage lambda (λファージ)
escherichia phage lambda (λファージ)