+検索条件
-Structure paper
| タイトル | Structural insights into dimerization and activation of the mGlu2-mGlu3 and mGlu2-mGlu4 heterodimers. |
|---|---|
| ジャーナル・号・ページ | Cell Res, Vol. 33, Issue 10, Page 762-774, Year 2023 |
| 掲載日 | 2023年6月8日 |
 著者 著者 | Xinwei Wang / Mu Wang / Tuo Xu / Ye Feng / Qiang Shao / Shuo Han / Xiaojing Chu / Yechun Xu / Shuling Lin / Qiang Zhao / Beili Wu /  |
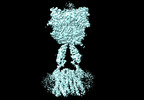
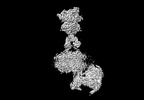
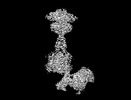
| PubMed 要旨 | Heterodimerization of the metabotropic glutamate receptors (mGlus) has shown importance in the functional modulation of the receptors and offers potential drug targets for treating central nervous ...Heterodimerization of the metabotropic glutamate receptors (mGlus) has shown importance in the functional modulation of the receptors and offers potential drug targets for treating central nervous system diseases. However, due to a lack of molecular details of the mGlu heterodimers, understanding of the mechanisms underlying mGlu heterodimerization and activation is limited. Here we report twelve cryo-electron microscopy (cryo-EM) structures of the mGlu2-mGlu3 and mGlu2-mGlu4 heterodimers in different conformational states, including inactive, intermediate inactive, intermediate active and fully active conformations. These structures provide a full picture of conformational rearrangement of mGlu2-mGlu3 upon activation. The Venus flytrap domains undergo a sequential conformational change, while the transmembrane domains exhibit a substantial rearrangement from an inactive, symmetric dimer with diverse dimerization patterns to an active, asymmetric dimer in a conserved dimerization mode. Combined with functional data, these structures reveal that stability of the inactive conformations of the subunits and the subunit-G protein interaction pattern are determinants of asymmetric signal transduction of the heterodimers. Furthermore, a novel binding site for two mGlu4 positive allosteric modulators was observed in the asymmetric dimer interfaces of the mGlu2-mGlu4 heterodimer and mGlu4 homodimer, and may serve as a drug recognition site. These findings greatly extend our knowledge about signal transduction of the mGlus. |
 リンク リンク |  Cell Res / Cell Res /  PubMed:37286794 / PubMed:37286794 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.8 - 3.7 Å |
| 構造データ | EMDB-36165, PDB-8jcu: EMDB-36166, PDB-8jcv: EMDB-36167, PDB-8jcw: EMDB-36168, PDB-8jcx: EMDB-36169, PDB-8jcy: EMDB-36170, PDB-8jcz: EMDB-36171, PDB-8jd0: EMDB-36172, PDB-8jd1: EMDB-36173, PDB-8jd2: EMDB-36174, PDB-8jd3: EMDB-36175, PDB-8jd4: EMDB-36176, PDB-8jd5: EMDB-36177, PDB-8jd6: 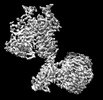 EMDB-36221: Cryo-EM map of Gi-bound metabotropic glutamate receptor mGlu4 focused on TMD and G protein 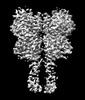 EMDB-36222: Cryo-EM map of Gi1-bound metabotropic glutamate receptor mGlu4 focused on ECD 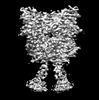 EMDB-36226: Cryo-EM map of Gi1-bound mGlu2-mGlu4 heterodimer focused on ECD  EMDB-36227: Cryo-EM map of Gi1-bound mGlu2-mGlu4 heterodimer focused on TMD and G protein  EMDB-36286: The consensus Cryo-EM map of Gi1-bound mGlu2-mGlu4 heterodimer  EMDB-36287: The consensus Cryo-EM map of Gi-bound metabotropic glutamate receptor mGlu4 |
| 化合物 |  ChemComp-Z99:  ChemComp-NAG:  ChemComp-CLR: 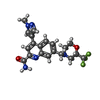 ChemComp-J9R:  ChemComp-GLU:  ChemComp-HZR:  ChemComp-PEF:  ChemComp-BQI:  ChemComp-BK0: 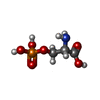 ChemComp-SEP: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / Complex structure / mGlu2-3 heterodimer / mGlu2-3 heterodimer in presence of NAM563 / mGlu2-mGlu3 heterodimer with Gi protein / mGlu2-mGlu4 heterodimer / Gi1-bound mGlu2-mGlu4 heterodimer / Gi-bound metabotropic glutamate receptor mGlu4 |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について























 homo sapiens (ヒト)
homo sapiens (ヒト)
