+検索条件
-Structure paper
| タイトル | Fragment screening libraries for the identification of protein hot spots and their minimal binding pharmacophores. |
|---|---|
| ジャーナル・号・ページ | Rsc Med Chem, Vol. 14, Page 135-143, Year 2023 |
| 掲載日 | 2022年5月20日 (構造データの登録日) |
 著者 著者 | Whitehouse, R.L. / Alwan, W.S. / Ilyichova, O.V. / Taylor, A.J. / Chandrashekaran, I.R. / Mohanty, B. / Doak, B.C. / Scanlon, M.J. |
 リンク リンク |  Rsc Med Chem / Rsc Med Chem /  PubMed:36760747 PubMed:36760747 |
| 手法 | X線回折 |
| 解像度 | 1.47 - 2.5 Å |
| 構造データ |  PDB-8cxd:  PDB-8cxe:  PDB-8czm:  PDB-8czn:  PDB-8d10:  PDB-8d11:  PDB-8d12:  PDB-8dg0:  PDB-8dg1:  PDB-8dg2: |
| 化合物 |  ChemComp-CU:  ChemComp-010:  ChemComp-HOH:  ChemComp-IMD:  ChemComp-BYZ:  ChemComp-PGE:  ChemComp-PKN: 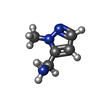 ChemComp-Q2I: 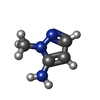 ChemComp-Q2O: 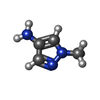 ChemComp-Q3F: 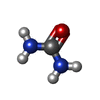 ChemComp-URE:  ChemComp-DMS:  ChemComp-GOL: |
| 由来 |
|
 キーワード キーワード | OXIDOREDUCTASE / DISULFIDE OXIDOREDUCTASE / REDOX PROTEIN / Microfrag screening / FBDD / Fragment / ANTIMICROBIAL PROTEIN |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




