+検索条件
-Structure paper
| タイトル | Atomic structure of the KEOPS complex: an ancient protein kinase-containing molecular machine. |
|---|---|
| ジャーナル・号・ページ | Mol. Cell, Vol. 32, Page 259-275, Year 2008 |
| 掲載日 | 2008年9月25日 (構造データの登録日) |
 著者 著者 | Mao, D.Y. / Neculai, D. / Downey, M. / Orlicky, S. / Haffani, Y.Z. / Ceccarelli, D.F. / Ho, J.S. / Szilard, R.K. / Zhang, W. / Ho, C.S. ...Mao, D.Y. / Neculai, D. / Downey, M. / Orlicky, S. / Haffani, Y.Z. / Ceccarelli, D.F. / Ho, J.S. / Szilard, R.K. / Zhang, W. / Ho, C.S. / Wan, L. / Fares, C. / Rumpel, S. / Kurinov, I. / Arrowsmith, C.H. / Durocher, D. / Sicheri, F. |
 リンク リンク |  Mol. Cell / Mol. Cell /  PubMed:18951093 PubMed:18951093 |
| 手法 | NMR (溶液) / X線回折 |
| 解像度 | 2.48 - 3.6 Å |
| 構造データ |  PDB-2k8y:  PDB-3en9:  PDB-3enc:  PDB-3enh:  PDB-3eno:  PDB-3enp: |
| 化合物 |  ChemComp-MG: 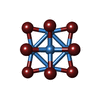 ChemComp-TBR:  ChemComp-HOH: |
| 由来 |
|
 キーワード キーワード | structural genomics / unknown function / regulatory subunit of the Bud32 kinase / component of the KEOPS complex / PSI-2 / Protein Structure Initiative / Northeast Structural Genomics Consortium / NESG / HYDROLASE / endopeptidase activity / protein kinase activity / protein serine/threonine kinase activity / ATP binding / metallopeptidase activity / hydrolase activity / metal ion binding / Metal-binding / Metalloprotease / Protease / Zinc / dimerization domain / KEOPS / telomere / hydrolase/unknown function / transcription / hydrolase-unknown function COMPLEX / KEOPS complex / ATPase / dimerization module / KEOPS complex telomere kinase regulator / Nucleus |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




 methanocaldococcus jannaschii (メタン生成菌)
methanocaldococcus jannaschii (メタン生成菌) homo sapiens (ヒト)
homo sapiens (ヒト)