-検索条件
-検索結果
検索 (著者・登録者: walker & s)の結果416件中、1から50件目までを表示しています
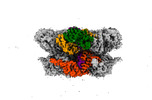
EMDB-42603: 
Human p97/VCP structure with a triazole inhibitor (NSC799462/hexamer)
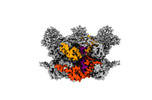
EMDB-42625: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC804515)
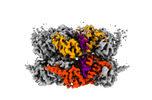
EMDB-42626: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC819701/up)
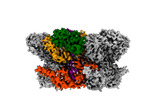
EMDB-42627: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC819701/down)
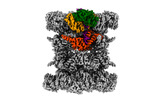
EMDB-44748: 
Human p97/VCP structure with a triazole inhibitor (NSC799462/dodecamer)

EMDB-50503: 
Omicron BA.1 Spike protein with neutralizing NTD specific mAb K501SP6

PDB-9fjk: 
Omicron BA.1 Spike protein with neutralizing NTD specific mAb K501SP6

EMDB-43551: 
CCHFV GP38 bound with ADI-46143 and ADI-46158 Fabs

EMDB-43552: 
CCHFV GP38 bound with ADI-58062 and ADI-63530 Fabs

EMDB-43553: 
CCHFV GP38 bound with ADI-58026 and ADI-63547 Fabs

EMDB-43604: 
CCHFV GP38 bound to ADI-46152 and ADI-58048 Fabs

PDB-8vww: 
CCHFV GP38 bound to ADI-46152 and ADI-58048 Fabs
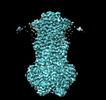
EMDB-40180: 
MsbA bound to cerastecin C

PDB-8gk7: 
MsbA bound to cerastecin C
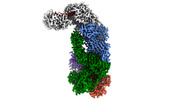
EMDB-40248: 
CRISPR-Cas type III-D effector complex
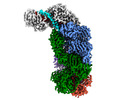
EMDB-40250: 
CRISPR-Cas type III-D effector complex bound to a self-target RNA in the pre-cleavage state
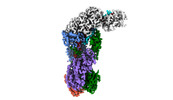
EMDB-40251: 
CRISPR-Cas type III-D effector complex bound to self-target RNA in a post-cleavage state

EMDB-40276: 
CRISPR-Cas type III-D effector complex consensus map

EMDB-40296: 
CRISPR-Cas type III-D effector complex local refinement map

EMDB-40297: 
CRISPR-Cas type III-D effector complex bound to a target RNA local refinement map

EMDB-40298: 
CRISPR-Cas type III-D effector complex bound to a target RNA consensus map

PDB-8s9t: 
CRISPR-Cas type III-D effector complex

PDB-8s9v: 
CRISPR-Cas type III-D effector complex bound to a self-target RNA in the pre-cleavage state

PDB-8s9x: 
CRISPR-Cas type III-D effector complex bound to self-target RNA in a post-cleavage state

EMDB-16441: 
Omicron B.1.1.529 2 RBD up conformation

PDB-8c5r: 
Omicron B.1.1.529 2 RBD up conformation

EMDB-29930: 
T. cruzi topoisomerase II alpha bound to dsDNA and the covalent inhibitor CT1

PDB-8gcc: 
T. cruzi topoisomerase II alpha bound to dsDNA and the covalent inhibitor CT1

EMDB-29790: 
30S focus refined map of WT E.coli ribosome complexed with A-site ortho-aminobenzoic acid charged tRNA-Phe

EMDB-27538: 
Lymphocytic choriomeningitis virus glycoprotein in complex with neutralizing antibody M28

EMDB-27539: 
Lymphocytic choriomeningitis virus glycoprotein

PDB-8dmh: 
Lymphocytic choriomeningitis virus glycoprotein in complex with neutralizing antibody M28

PDB-8dmi: 
Lymphocytic choriomeningitis virus glycoprotein

EMDB-29786: 
Structure of WT E.coli 70S ribosome complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site ortho-aminobenzoic acid charged NH-tRNAPhe

EMDB-29788: 
Structure of WT E.coli ribosome 50S subunit with complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site 3-aminopyridine-4-carboxylic acid charged NH-tRNAPhe

PDB-8g6w: 
Structure of WT E.coli 70S ribosome complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site ortho-aminobenzoic acid charged NH-tRNAPhe

PDB-8g6y: 
Structure of WT E.coli ribosome 50S subunit with complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site 3-aminopyridine-4-carboxylic acid charged NH-tRNAPhe

EMDB-29787: 
Structure of WT E.coli ribosome 50S subunit with complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site meta-aminobenzoic acid charged NH-tRNAPhe

PDB-8g6x: 
Structure of WT E.coli ribosome 50S subunit with complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site meta-aminobenzoic acid charged NH-tRNAPhe

EMDB-27232: 
Subtomogram of Fluad vaccine hemagglutinin spiked nanodisc

EMDB-27233: 
Tomogram of Flublok vaccine hemagglutinin starfish complexes

EMDB-25699: 
VFLIP Spike Trimer with GAR03

EMDB-25700: 
VFLIP Spike Trimer with GAR05 FAB

PDB-7t5o: 
VFLIP Spike Trimer with GAR03

EMDB-26653: 
Native Lassa glycoprotein in complex with neutralizing antibodies 8.9F and 37.2D

EMDB-26655: 
Native Lassa glycoprotein in complex with neutralizing antibodies 12.1F and 37.2D

PDB-7uot: 
Native Lassa glycoprotein in complex with neutralizing antibodies 8.9F and 37.2D

PDB-7uov: 
Native Lassa glycoprotein in complex with neutralizing antibodies 12.1F and 37.2D

EMDB-26443: 
Structure of the DU422 SOSIP.664 trimer in complex with neutralizing antibody Fab fragments 10-1074 and BG24

PDB-7ucg: 
Structure of the DU422 SOSIP.664 trimer in complex with neutralizing antibody Fab fragments 10-1074 and BG24
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
