-検索条件
-検索結果
検索 (著者・登録者: tan & cw)の結果全47件を表示しています

EMDB-16110: 
Human Urea Transporter UT-A (N-Terminal Domain Model)

EMDB-16111: 
Map of Human Urea Transporter UT-A Collected with 0 and 30 Degree Tilts

EMDB-16112: 
Human Urea Transporter UT-B/UT1 in Complex with Inhibitor UTBinh-14

EMDB-33650: 
SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment of anti-RBD antibody E7

EMDB-33651: 
SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment of anti-RBD antibody E7 (focused refinement on Fab-RBD interface)

EMDB-28189: 
SARS-CoV-2 Spike in complex with biparatopic nanobody BP10
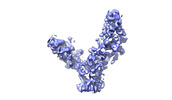
EMDB-28190: 
SARS-CoV-2 RBD in complex with biparatopic nanobody BP10 local refinement

EMDB-26383: 
Cryo-EM structure of the core human NADPH oxidase NOX2

EMDB-15526: 
In situ cryo-electron tomogram of a bulk autophagy phagophore in S. cerevisiae

EMDB-15545: 
In situ cryo-electron tomogram of a bulk autophagy phagophore in S. cerevisiae #2

EMDB-15546: 
In situ cryo-electron tomogram of a bulk autophagy autophagosome with END cargo in S. cerevisiae #1

EMDB-15547: 
In situ cryo-electron tomogram of a bulk autophagy autophagosome fusing with the vacuole in S. cerevisiae #1

EMDB-15548: 
In situ cryo-electron tomogram of a bulk autophagy phagophore in S. cerevisiae #3

EMDB-15549: 
In situ cryo-electron tomogram of a bulk autophagy phagophore in S. cerevisiae #4

EMDB-25817: 
Cryo-EM map of protomer of the cytoplasmic ring of the nuclear pore complex from Xenopus laevis

EMDB-11804: 
Structure of Wild-Type Human Potassium Chloride Transporter KCC3 in NaCl (LMNG/CHS)

EMDB-11805: 
Structure of Wild-type Human Potassium Chloride Transporter KCC3 in NaCl (MSP E3D1)

EMDB-22995: 
Spike protein trimer

EMDB-12311: 
Structure of Wild-Type Human Potassium Chloride Transporter KCC3 in NaCl (LMNG/CHS)

EMDB-11799: 
Structure of Human Potassium Chloride Transporter KCC3 S45D/T940D/T997D in NaCl (Reference Map)

EMDB-11800: 
Structure of Human Potassium Chloride Transporter KCC3 S45D/T940D/T997D in NaCl (Subclass)

EMDB-22993: 
SARS-CoV-2 spike glycoprotein:Fab 5A6 complex I

EMDB-22994: 
SARS-CoV-2 Spike protein in complex with Fab 2H4

EMDB-22997: 
SARS-CoV-2 spike glycoprotein:Fab 3D11 complex

EMDB-23707: 
SARS-CoV-2 Spike:5A6 Fab complex I focused refinement

EMDB-23709: 
SARS-CoV-2 Spike:Fab 3D11 complex focused refinement

EMDB-23717: 
SARS-CoV-2 S-NTD + Fab CM25

EMDB-10704: 
Structure of Human Potassium Chloride Transporter KCC3b (S45D/T940D/T997D) in KCl

EMDB-11128: 
Cryo-electron tomogram of human Uromodulin filaments

EMDB-11129: 
Subtomogram average of human Uromodulin filaments

EMDB-11130: 
Cryo-electron tomogram of elastase treated human Uromodulin filaments (eUmod)

EMDB-11131: 
Subtomogram average of elastase treated human Uromodulin filaments (eUmod)

EMDB-11132: 
Cryo-electron tomogram of human Uromodulin filaments incubated with FimH lectin domain
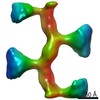
EMDB-11133: 
Subtomogram average of human Uromodulin filaments incubated with FimH lectin domain

EMDB-11134: 
Cryo-electron tomogram of elastase treated human Uromodulin filaments incubated with FimH lectin domain

EMDB-11135: 
Subtomogram average of elastase treated human Uromodulin fibers incubated with FimH lectin domain

EMDB-11136: 
Cryo-electron tomogram of E. coli AAEC [pSH2] incubated with human Uromodulin filaments

EMDB-11137: 
Cryo-electron tomogram of non-piliated E. coli AAEC189 incubated with human Uromodulin filaments

EMDB-11138: 
Cryo-electron tomogram of FIB milled E.coli - Uromodulin aggregates

EMDB-11139: 
Cryo-electron tomogram of E. coli AAEC189 [pSH2] incubated with elastase treated human Uromodulin filaments

EMDB-11140: 
Cryo-electron tomogram of urine from patient with E. coli urinary tract infection

EMDB-11141: 
Cryo-electron tomogram of urine from patient with K. pneumoniae urinary tract infection

EMDB-11142: 
Cryo-electron tomogram of urine from patient with P. aeruginosa urinary tract infection

EMDB-11143: 
Cryo-electron tomogram of urine from patient with S. mitis urinary tract infection

EMDB-4746: 
CryoEM structure of calcium-bound human TMEM16K / Anoctamin 10 in detergent (2mM Ca2+, closed form)

EMDB-4747: 
CryoEM structure of calcium-bound human TMEM16K / Anoctamin 10 in detergent (low Ca2+, closed form)

EMDB-4748: 
CryoEM structure of calcium-free human TMEM16K / Anoctamin 10 in detergent (closed form)
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
