-検索条件
-検索結果
検索 (著者・登録者: shao & f)の結果875件中、1から50件目までを表示しています

EMDB-37546: 
Spike Trimer of BA.2.86 in complex with one hACE2

EMDB-37548: 
Spike Trimer of BA.2.86 in complex with two hACE2s

EMDB-37549: 
Spike Trimer of BA.2.86 with three RBDs down

EMDB-37550: 
Spike Trimer of BA.2.86 with single RBD up

EMDB-38049: 
SARS-CoV-2 JN.1 Spike

EMDB-38072: 
SARS-CoV-2 BA.2.75 Spike with K356T mutation (3 RBD down)

EMDB-38073: 
SARS-CoV-2 BA.2.75 Spike with K356T mutation (1 RBD up)

EMDB-38681: 
BA.2.86 Spike Trimer in complex with heparan sulfate

EMDB-38682: 
JN.1 Spike Trimer in complex with heparan sulfate

EMDB-38683: 
XBB.1.5 Spike Trimer in complex with heparan sulfate

EMDB-38684: 
BA.2.86-T356K Spike Trimer in complex with heparan sulfate (Local refinement)

EMDB-37553: 
BA.2.86 RBD in complex with hACE2 (local refinement)

EMDB-38056: 
BA.2.86 Spike Trimer with ins483V mutation (3 RBD down)

EMDB-38057: 
BA.2.86 Spike Trimer with ins483V mutation (1 RBD up)

EMDB-38063: 
BA.2.86 Spike Trimer with T356K mutation (3 RBD down)

EMDB-38064: 
BA.2.86 Spike Trimer with T356K mutation (1 RBD up)

EMDB-38700: 
XBB.1.5-K356T S-trimer (1 RBD up)

EMDB-38701: 
XBB.1.5-K356T S-trimer (3 RBDs down)

EMDB-36721: 
Structure of TbAQP2 in complex with anti-trypanosomatid drug melarsoprol

EMDB-36722: 
Structure of the TbAQP2 in the apo conformation

EMDB-36723: 
Structure of TbAQP2 in complex with anti-trypanosomatid drug pentamidine

PDB-8jy6: 
Structure of TbAQP2 in complex with anti-trypanosomatid drug melarsoprol

PDB-8jy7: 
Structure of the TbAQP2 in the apo conformation

PDB-8jy8: 
Structure of TbAQP2 in complex with anti-trypanosomatid drug pentamidine

EMDB-36732: 
Cryo-EM structure of the gasdermin pore from Trichoplax adhaerens

EMDB-36733: 
Cryo-EM structure of the gasdermin pore from Trichoplax adhaerens

EMDB-36734: 
Cryo-EM structure of RCD-1 pore from Neurospora crassa

PDB-8jyw: 
Cryo-EM structure of the gasdermin pore from Trichoplax adhaerens

PDB-8jyz: 
Cryo-EM structure of RCD-1 pore from Neurospora crassa

EMDB-35832: 
Cryo-EM structure of the GPR34 receptor in complex with the antagonist YL-365

PDB-8iyx: 
Cryo-EM structure of the GPR34 receptor in complex with the antagonist YL-365

EMDB-35492: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with ceramide

EMDB-35493: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with diacylglycerol/phosphoethanolamine

EMDB-37383: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with ceramide/phosphoethanolamine

EMDB-37385: 
The cryo-EM structure of human sphingomyelin synthase-related protein

PDB-8ijq: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with ceramide

PDB-8ijr: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with diacylglycerol/phosphoethanolamine

PDB-8w9w: 
The cryo-EM structure of human sphingomyelin synthase-related protein in complex with ceramide/phosphoethanolamine

PDB-8w9y: 
The cryo-EM structure of human sphingomyelin synthase-related protein

EMDB-33990: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAbs 3E8 and 5E3 (localized refinement)
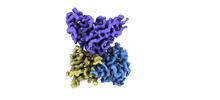
EMDB-33992: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAb 10E4 (localized refinement)
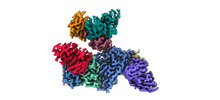
EMDB-33993: 
Cryo-EM density map of EBV gHgL-gp42 in complex with four mAbs 5E3, 3E8, 6H2 and 10E4
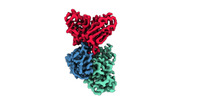
EMDB-33994: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAb 6H2 (localized refinement)

PDB-7yoy: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAbs 3E8 and 5E3 (localized refinement)

PDB-7yp1: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAb 10E4 (localized refinement)

PDB-7yp2: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAb 6H2 (localized refinement)

EMDB-42044: 
H2HA A/Japan/305/1957 complexed with mouse polyclonal Fab - wk 12, reoriented immunogen

EMDB-42045: 
H2HA A/Japan/305/1957 complexed with mouse polyclonal Fab - wk 12, control immunogen

EMDB-42046: 
H7N9 A/Shanghai/2/2013 complexed with mouse polyclonal Fab - wk 12, control immunogen

EMDB-42048: 
H3N2 A/Victoria/3/1975 complexed with mouse polyclonal Fab - wk 7, control immunogen
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
