-検索条件
-検索結果
検索 (著者・登録者: seitz & i)の結果51件中、1から50件目までを表示しています

EMDB-16076: 
Helical shell of CCMV capsid protein on DNA origami 6HB-2k

EMDB-16077: 
Inner helical shell of CCMV capsid protein on DNA origami 6HB-10k

EMDB-16078: 
Outer helical shell of CCMV capsid protein on DNA origami 6HB-10k
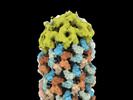
EMDB-16079: 
Helical shell cap of CCMV capsid protein on DNA origami 6HB-2k

EMDB-16080: 
Helical shell of CCMV capsid protein on DNA origami 24HB-2.5k

PDB-8bi4: 
Helical shell of CCMV capsid protein on DNA origami 6HB-2k

EMDB-15295: 
African cichlid nackednavirus capsid at pH 7.5

EMDB-16371: 
African cichlid nackednavirus capsid at pH 5.5

PDB-8aac: 
African cichlid nackednavirus capsid at pH 7.5

PDB-8c0o: 
African cichlid nackednavirus capsid at pH 5.5

EMDB-25183: 
P. chlororaphis 70S ribosome in situ subtomogram average

EMDB-25220: 
In situ subtomogram average of the 201phi2-1 phage nucleus major shell protein, chimallin (concave class)

EMDB-25221: 
In situ consensus subtomogram average of the 201phi2-1 chimallin

EMDB-25222: 
In situ subtomogram average of 201phi2-1 phage nucleus major shell protein, chimallin (intermediate/flat class)

EMDB-25223: 
In situ subtomogram average of the 201phi2-1 phage nucleus major shell protein, chimallin (convex class)

EMDB-25229: 
In situ subtomogram average of the Goslar major phage nucleus shell protein, chimallin (consensus class)

EMDB-25262: 
In situ subtomogram average of Goslar phage nucleus major shell protein, chimallin (concave class)

EMDB-25358: 
In situ subtomogram average of the major Goslar phage nucleus shell protein, chimallin (convex class)

EMDB-25359: 
In situ subtomogram average of the APEC2248 70S ribosome

EMDB-25360: 
In situ subtomogram average of the APEC2248 50S ribosome

EMDB-25390: 
201Phi2-1 Chimallin Cubic (O, 24mer) assembly

EMDB-25391: 
201phi2-1 Chimallin localized tetramer reconstruction

EMDB-25392: 
201phi2-1 Chimallin C1 localized reconstruction

EMDB-25393: 
201phi2-1 chimallin rectangular (D4,40mer) assembly

EMDB-25394: 
Goslar chimallin cubic (O, 24mer) assembly

EMDB-25395: 
Goslar chimallin C4 tetramer localized reconstruction

EMDB-25396: 
Goslar chimallin C1 localized reconstruction

PDB-7sqq: 
201Phi2-1 Chimallin Cubic (O, 24mer) assembly

PDB-7sqr: 
201phi2-1 Chimallin localized tetramer reconstruction

PDB-7sqs: 
201phi2-1 Chimallin C1 localized reconstruction

PDB-7sqt: 
Goslar chimallin cubic (O, 24mer) assembly

PDB-7squ: 
Goslar chimallin C4 tetramer localized reconstruction

PDB-7sqv: 
Goslar chimallin C1 localized reconstruction

EMDB-11993: 
Structure of elongating SARS-CoV-2 RNA-dependent RNA polymerase with Remdesivir at position -3 (structure 1)

EMDB-11994: 
Structure of elongating SARS-CoV-2 RNA-dependent RNA polymerase with Remdesivir at position -4 (structure 2)

EMDB-11995: 
Structure of elongating SARS-CoV-2 RNA-dependent RNA polymerase with AMP at position -4 (structure 3)

PDB-7b3b: 
Structure of elongating SARS-CoV-2 RNA-dependent RNA polymerase with Remdesivir at position -3 (structure 1)

PDB-7b3c: 
Structure of elongating SARS-CoV-2 RNA-dependent RNA polymerase with Remdesivir at position -4 (structure 2)

PDB-7b3d: 
Structure of elongating SARS-CoV-2 RNA-dependent RNA polymerase with AMP at position -4 (structure 3)

EMDB-3822: 
Structure of the full-length African cichlid nackednavirus icosahedral capsid

EMDB-3823: 
Structure of the truncated African cichlid nackednavirus icosahedral capsid

EMDB-1399: 
Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions.

EMDB-1400: 
Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions.

EMDB-1401: 
Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions.

EMDB-1402: 
Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions.

EMDB-1403: 
Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions.

EMDB-1404: 
Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions.

EMDB-1405: 
Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions.

EMDB-1406: 
Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions.

EMDB-1407: 
Cryo-electron microscopy of hepatitis B virions reveals variability in envelope capsid interactions.
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
