-検索条件
-検索結果
検索 (著者・登録者: schultz & c)の結果119件中、1から50件目までを表示しています

EMDB-40825: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

PDB-8sx3: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

EMDB-41024: 
MD65 N332-GT5 SOSIP in complex with RM_N332_03 Fab and RM20A3 Fab

EMDB-41025: 
MD65 N332-GT5 SOSIP in complex with RM_N332_36 Fab and RM20A3 Fab

EMDB-41026: 
MD65 N332-GT5 SOSIP in complex with RM_N332_32 Fab and RM20A3

EMDB-41027: 
MD65 N332-GT5 SOSIP in complex with RM_N332_08 Fab and RM20A3 Fab

EMDB-41034: 
MD64 N332-GT5 SOSIP

EMDB-41035: 
MD65 N332-GT5 SOSIP in complex with RM_N332_07 Fab and RM20A3 Fab

PDB-8t49: 
MD65 N332-GT5 SOSIP in complex with RM_N332_03 Fab and RM20A3 Fab

PDB-8t4a: 
MD65 N332-GT5 SOSIP in complex with RM_N332_36 Fab and RM20A3 Fab

PDB-8t4b: 
MD65 N332-GT5 SOSIP in complex with RM_N332_32 Fab and RM20A3

PDB-8t4d: 
MD65 N332-GT5 SOSIP in complex with RM_N332_08 Fab and RM20A3 Fab

PDB-8t4k: 
MD64 N332-GT5 SOSIP

PDB-8t4l: 
MD65 N332-GT5 SOSIP in complex with RM_N332_07 Fab and RM20A3 Fab
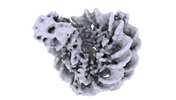
EMDB-18497: 
Human Sirtuin 6 in complex with nucleosome - structure showing H2A tail path

EMDB-42853: 
Cryo-EM structure of the unliganded hexameric prenyltransferase in bifunctional copalyl diphosphate synthase from Penicillium fellutanum with an open conformation
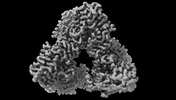
EMDB-42855: 
Cryo-EM structure of the unliganded hexameric prenyltransferase in bifunctional copalyl diphosphate synthase from Penicillium fellutanum with an open conformation, reconstruction in C1

PDB-8v0f: 
Cryo-EM structure of the unliganded hexameric prenyltransferase in bifunctional copalyl diphosphate synthase from Penicillium fellutanum with an open conformation

EMDB-16842: 
SIRT6 bound nucleosome

EMDB-16843: 
Nucleosome bound human SIRT6

EMDB-16845: 
Nucleosome Bound human SIRT6 (composite structure)

EMDB-16861: 
Sirt6 bound nucleosome - consensus map

PDB-8of4: 
Nucleosome Bound human SIRT6 (Composite)

EMDB-41158: 
CS2it1p2_F7K local refinement for HCMV Trimer in complex with CS2it1p2_F7K Fab and CS4tt1p1_E3K Fab

EMDB-41180: 
Global reconstruction for HCMV Pentamer in complex with CS2pt1p2_A10L Fab and CS3pt1p4_C1L Fab

EMDB-15869: 
NuA4 Histone Acetyltransferase Complex Epl1 full length

EMDB-15881: 
NuA4 Histone Acetyltransferase Complex Full length Epl1 Core

EMDB-15896: 
NuA4 Histone Acetyltransferase Complex Shortened Epl1 Loop 1 (SL1)

EMDB-15897: 
NuA4 Histone Acetyltransferase Complex Epl1 Shortened Loop 1 (SL1) core
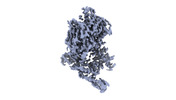
EMDB-15066: 
NuA4 Histone Acetyltransferase complex - Upper lobe

EMDB-15067: 
NuA4 Histone Acetyltransferase complex - Tra1 part 1
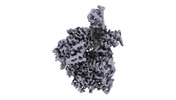
EMDB-15070: 
NuA4 Histone Acetyltransferase complex - Tra1 Part 2

EMDB-14989: 
NuA4 Histone Acetyltransferase Complex (Composite)

PDB-7zvw: 
NuA4 Histone Acetyltransferase Complex

EMDB-14980: 
NuA4 Histone Acetyltransferase complex

EMDB-24479: 
Structure of ACLY-D1026A-substrates

EMDB-24511: 
Structure of ACLY D1026A-substrates-asym-int

EMDB-24577: 
Structure of ACLY D1026A - substrates-asym

EMDB-29668: 
Structure of ACLY-D1026A-products-asym

EMDB-29669: 
Structure of ACLY-D1026A-products

EMDB-29739: 
Structure of ACLY-D1026A-substrates, local refinement of ASH domain

EMDB-29740: 
Structure of ACLY-D1026A-products, local refinement of ASH domain

PDB-7rig: 
Structure of ACLY-D1026A-substrates

PDB-7rkz: 
Structure of ACLY D1026A-substrates-asym-int

PDB-7rmp: 
Structure of ACLY D1026A - substrates-asym

PDB-8g1e: 
Structure of ACLY-D1026A-products-asym

PDB-8g1f: 
Structure of ACLY-D1026A-products

PDB-8g5c: 
Structure of ACLY-D1026A-substrates, local refinement of ASH domain

PDB-8g5d: 
Structure of ACLY-D1026A-products, local refinement of ASH domain

EMDB-29657: 
Semi-synthetic CoA-alpha-Synuclein Constructs Trap N-terminal Acetyltransferase NatB for Binding Mechanism Studies
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
