-検索条件
-検索結果
検索 (著者・登録者: pestova & tv)の結果60件中、1から50件目までを表示しています

EMDB-25535: 
Structure of the HCV IRES bound to the 40S ribosomal subunit, head open. Structure 9(delta dII)

EMDB-25536: 
Structure of the wt IRES and 40S ribosome binary complex, open conformation. Structure 10(wt)

EMDB-25537: 
Structure of the wt IRES and 40S ribosome ternary complex, open conformation. Structure 11(wt)

EMDB-25538: 
Structure of the wt IRES eIF2-containing 48S initiation complex, closed conformation. Structure 12(wt).

EMDB-25539: 
Structure of the delta dII IRES eIF2-containing 48S initiation complex, closed conformation. Structure 12(delta dII).

EMDB-25542: 
Structure of the wt IRES eIF5B-containing pre-48S initiation complex, open conformation. Structure 14(wt)
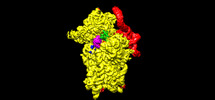
EMDB-25545: 
Map of wt IRES eIF2-containing 48S initiation complex with IRES domain II in position 1. Structure 16(wt).
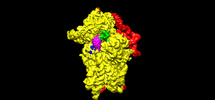
EMDB-25546: 
Map of wt IRES eIF2-containing 48S initiation complex with IRES domain II in position 2. Structure 17(wt).
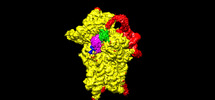
EMDB-25547: 
Map of wt IRES eIF2-containing 48S initiation complex with IRES domain II in position 3. Structure 18(wt)
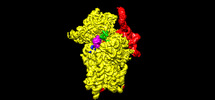
EMDB-25548: 
Map of wt IRES eIF2-containing 48S initiation complex with IRES domain II in position 4. Structure 19(wt)
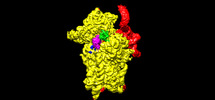
EMDB-25549: 
Map of wt IRES eIF2-containing 48S initiation complex with IRES domain II in position 5. Structure 20(wt)
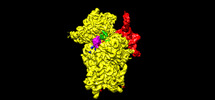
EMDB-25550: 
Map of wt IRES eIF2-containing 48S initiation complex with IRES domain II in position 6. Structure 21(wt)
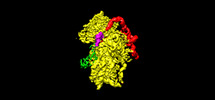
EMDB-25551: 
Map of wt IRES eIF5B-containing 48S initiation complex with IRES domain II in position 7. Structure 22(wt)

EMDB-25552: 
Map of wt IRES eIF5B-containing 48S initiation complex with IRES domain II in position 8. Structure 23(wt)
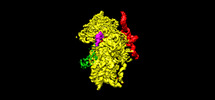
EMDB-25553: 
Map of wt IRES eIF5B-containing 48S initiation complex with IRES domain II in position 9. Structure 24(wt)
EMDB-25554: 
Map of wt IRES eIF5B-containing 48S initiation complex with IRES domain II in position 10. Structure 25(wt)

EMDB-25555: 
Map of wt IRES eIF5B-containing 48S initiation complex with IRES domain II in position 11. Structure 26(wt)
EMDB-25556: 
Map of wt IRES eIF5B-containing 48S initiation complex with IRES domain II in position 12. Structure 27(wt)
EMDB-25557: 
Map of wt IRES eIF5B-containing 48S initiation complex with IRES domain II in position 13. Structure 28(wt)
EMDB-25597: 
Consensus map of open 40S ribosome from wt IRES eIF5B-containing sample
EMDB-25598: 
Consensus map of closed 40S ribosome from wt IRES eIF5B-containing sample
EMDB-25599: 
Consensus map of closed 40S ribosome from delta dII IRES eIF5B-containing sample

EMDB-25529: 
Structure of the HCV IRES binding to the 40S ribosomal subunit, closed conformation. Structure 3(delta dII)

EMDB-25530: 
Structure of the HCV IRES binding to the 40S ribosomal subunit, closed conformation. Structure 4(delta dII)

EMDB-25531: 
Structure of the HCV IRES binding to the 40S ribosomal subunit, closed conformation. Structure 5(delta dII)

EMDB-25532: 
Structure of the HCV IRES bound to the 40S ribosomal subunit, closed conformation. Structure 6(delta dII)
EMDB-25540: 
Structure of the wt IRES w/o eIF2 48S initiation complex, closed conformation. Structure 13(wt)

EMDB-25541: 
Structure of the delta dII IRES w/o eIF2 48S initiation complex, closed conformation. Structure 13(delta dII)

EMDB-25543: 
Structure of the wt IRES eIF5B-containing 48S initiation complex, closed conformation. Structure 15(wt)

EMDB-25544: 
Structure of the delta dII IRES eIF5B-containing 48S initiation complex, closed conformation. Structure 15(delta dII)

EMDB-25588: 
Consensus map of open 40S ribosome from wt IRES eIF2-containing sample
EMDB-25589: 
Consensus map of open 40S ribosome from wt IRES eIF5B-containing sample

EMDB-25594: 
Consensus map of open 40S ribosome from wt IRES eIF5B-, and eIF2-containing sample
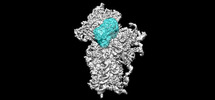
EMDB-25595: 
Consensus map of closed 40S ribosome from wt IRES eIF2-containing sample
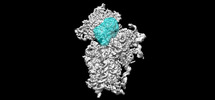
EMDB-25596: 
Consensus map of closed 40S ribosome from delta dII IRES eIF2-containing sample

EMDB-25602: 
Map of the wt IRES eIF5B-containing 48S initiation complex, closed conformation

EMDB-25603: 
Map of the wt IRES eIF5B-containing pre-48S initiation complex, open conformation

EMDB-25604: 
Map of the wt IRES eIF2-containing 48S initiation complex, closed conformation

EMDB-25620: 
Map of the delta IRES eIF2-containing 48S initiation complex w/o eIF2, closed conformation

EMDB-25533: 
Structure of the HCV IRES bound to the 40S ribosomal subunit, head opening. Structure 7(delta dII)
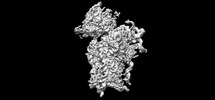
EMDB-25590: 
Consensus map of 40S ribosome with head motion from wt IRES eIF2-containing sample
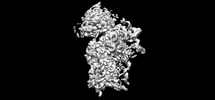
EMDB-25591: 
Consensus map of 40S ribosome with head motion from wt IRES eIF5B-containing sample
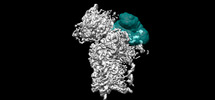
EMDB-25592: 
Consensus map of 40S ribosome showing IRES binding
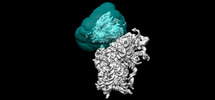
EMDB-25593: 
Consensus map of 40S ribosome with head motion from wt IRES eIF5B-, and eIF2-containing sample

EMDB-25527: 
Structure of the HCV IRES binding to the 40S ribosomal subunit, closed conformation. Structure 1(delta dII)

EMDB-25528: 
Structure of the HCV IRES binding to the 40S ribosomal subunit, closed conformation. Structure 2(delta dII)

EMDB-25534: 
Structure of the HCV IRES bound to the 40S ribosomal subunit, head opening. Structure 8(delta dII)

EMDB-25600: 
Map of the wt IRES eIF5B-containing 48S initiation complex, closed conformation. eIF5B in position 1

EMDB-25601: 
Map of the wt IRES eIF5B-containing 48S initiation complex, closed conformation. eIF5B in position 2

EMDB-11590: 
The Halastavi arva virus intergenic region IRES promotes translation by the simplest possible initiation mechanism
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
