-検索条件
-検索結果
検索 (著者・登録者: perry & k)の結果57件中、1から50件目までを表示しています

EMDB-37827: 
Cryo-EM structure of the IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the pre-strand exchange state

EMDB-37828: 
Cryo-EM structure of the IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the pre-strand exchange locked state

EMDB-37829: 
Cryo-EM structure of the IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the post-strand exchange state (Holliday junction intermediate)

EMDB-37830: 
Cryo-EM structure of the IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the post-strand exchange state (Holliday junction resolution)

PDB-8wt6: 
Cryo-EM structure of the IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the pre-strand exchange state

PDB-8wt7: 
Cryo-EM structure of the IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the pre-strand exchange locked state

PDB-8wt8: 
Cryo-EM structure of the IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the post-strand exchange state (Holliday junction intermediate)

PDB-8wt9: 
Cryo-EM structure of the IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the post-strand exchange state (Holliday junction resolution)

EMDB-15525: 
Cryo-EM structure of the RecA postsynaptic filament from S. pneumoniae

PDB-8amf: 
Cryo-EM structure of the RecA postsynaptic filament from S. pneumoniae

EMDB-41075: 
SARS-CoV-2 spike in complex with Fab 71281-33

EMDB-41076: 
SARS-CoV-2 spike in complex with Fab 71281-33 (2)

EMDB-15524: 
Cryo-EM structure of the RecA presynaptic filament from S.pneumoniae

PDB-8amd: 
Cryo-EM structure of the RecA presynaptic filament from S.pneumoniae

EMDB-26639: 
SARS-CoV-2 replication-transcription complex bound to Remdesivir triphosphate, in a pre-catalytic state

EMDB-26641: 
SARS-CoV-2 replication-transcription complex bound to ATP, in a pre-catalytic state

EMDB-26642: 
SARS-CoV-2 replication-transcription complex bound to UTP, in a pre-catalytic state.

EMDB-26645: 
SARS-CoV-2 replication-transcription complex bound to GTP, in a pre-catalytic state

EMDB-26646: 
SARS-CoV-2 replication-transcription complex bound to CTP, in a pre-catalytic state

PDB-7uo4: 
SARS-CoV-2 replication-transcription complex bound to Remdesivir triphosphate, in a pre-catalytic state

PDB-7uo7: 
SARS-CoV-2 replication-transcription complex bound to ATP, in a pre-catalytic state

PDB-7uo9: 
SARS-CoV-2 replication-transcription complex bound to UTP, in a pre-catalytic state

PDB-7uob: 
SARS-CoV-2 replication-transcription complex bound to GTP, in a pre-catalytic state

PDB-7uoe: 
SARS-CoV-2 replication-transcription complex bound to CTP, in a pre-catalytic state
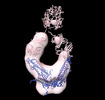
EMDB-26217: 
Negative stain EM map of COVA1-07 mAb bound to the S2 domain of SARS-CoV-2 S
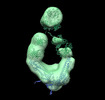
EMDB-26218: 
Negative stain EM map of COVA2-14 mAb bound to the S2 domain of SARS-CoV-2 S

EMDB-26219: 
Negative stain EM map of COVA2-18 mAb bound to the S2 domain of SARS-CoV-2 S
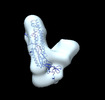
EMDB-26220: 
Negative stain EM map of the S2 domain of SARS-CoV-2 S

EMDB-26667: 
Cryo-EM structure of SHOC2-PP1c-MRAS holophosphatase complex

PDB-7upi: 
Cryo-EM structure of SHOC2-PP1c-MRAS holophosphatase complex

EMDB-24427: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC - engaged class

EMDB-24429: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC - swiveled class

EMDB-24430: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC (composite)

EMDB-24431: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(1)-RTC

PDB-7rdy: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC - engaged class

PDB-7re0: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC - swiveled class

PDB-7re1: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC (composite)

PDB-7re2: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(1)-RTC

EMDB-24426: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC - open class

EMDB-24428: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC - apo class

EMDB-24432: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC dimer

PDB-7rdx: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC - open class

PDB-7rdz: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC - apo class

PDB-7re3: 
SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC dimer

EMDB-12693: 
Cryo-EM structure of an Escherichia coli TnaC-ribosome complex stalled in response to L-tryptophan

EMDB-12694: 
Cryo-EM structure of an Escherichia coli TnaC(R23F)-ribosome complex stalled in response to L-tryptophan

EMDB-12695: 
Cryo-EM structure of an Escherichia coli TnaC(R23F)-ribosome-RF2 complex stalled in response to L-tryptophan

PDB-7o19: 
Cryo-EM structure of an Escherichia coli TnaC-ribosome complex stalled in response to L-tryptophan

PDB-7o1a: 
Cryo-EM structure of an Escherichia coli TnaC(R23F)-ribosome complex stalled in response to L-tryptophan

PDB-7o1c: 
Cryo-EM structure of an Escherichia coli TnaC(R23F)-ribosome-RF2 complex stalled in response to L-tryptophan
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
