-検索条件
-検索結果
検索 (著者・登録者: lutz & m)の結果59件中、1から50件目までを表示しています

EMDB-41158: 
CS2it1p2_F7K local refinement for HCMV Trimer in complex with CS2it1p2_F7K Fab and CS4tt1p1_E3K Fab

EMDB-41180: 
Global reconstruction for HCMV Pentamer in complex with CS2pt1p2_A10L Fab and CS3pt1p4_C1L Fab

EMDB-28858: 
Top-down design of protein architectures with reinforcement learning

EMDB-28859: 
Top-down design of protein architectures with reinforcement learning
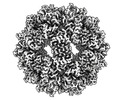
EMDB-28860: 
Top-down design of protein architectures with reinforcement learning

PDB-8f4x: 
Top-down design of protein architectures with reinforcement learning

PDB-8f53: 
Top-down design of protein architectures with reinforcement learning

PDB-8f54: 
Top-down design of protein architectures with reinforcement learning

EMDB-12684: 
ABC transporter NosFY, nucleotide-free in GDN

EMDB-12688: 
ABC transporter NosDFY, nucleotide-free in lipid nanodisc

PDB-7o0z: 
ABC transporter NosFY, nucleotide-free in GDN

PDB-7o13: 
ABC transporter NosDFY, nucleotide-free in lipid nanodisc

EMDB-13885: 
CryoEM structure of the ABC transporter NosDFY complexed with nitrous oxide reductase NosZ

EMDB-14813: 
ABC transporter complex NosDFYL in GDN

PDB-7qba: 
CryoEM structure of the ABC transporter NosDFY complexed with nitrous oxide reductase NosZ

PDB-7znq: 
ABC transporter complex NosDFYL in GDN

EMDB-13049: 
ABC Transporter complex NosDFYL, R-domain 1

EMDB-13050: 
ABC Transporter complex NosDFYL, consensus refinement

EMDB-13051: 
ABC Transporter complex NosDFYL, R-domain 2

EMDB-13052: 
ABC Transporter complex NosDFYL, R-domain 3
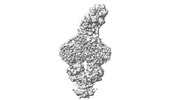
EMDB-13053: 
ABC Transporter complex NosDFYL, membrane anchor

PDB-7osf: 
ABC Transporter complex NosDFYL, R-domain 1

PDB-7osg: 
ABC Transporter complex NosDFYL, consensus refinement

PDB-7osh: 
ABC Transporter complex NosDFYL, R-domain 2

PDB-7osi: 
ABC Transporter complex NosDFYL, R-domain 3

PDB-7osj: 
ABC Transporter complex NosDFYL, membrane anchor

EMDB-12683: 
ABC transporter NosDFY, nucleotide-free in GDN

EMDB-12685: 
ABC transporter NosDFY, nucleotide-free in GDN, R-domain 2
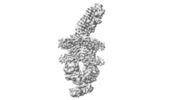
EMDB-12686: 
ABC transporter NosDFY, nucleotide-free in GDN, R-domain 1

EMDB-12687: 
ABC transporter NosDFY, AMPPNP-bound in GDN

EMDB-12689: 
ABC transporter NosDFY, nucleotide-free in lipid nanodisc, R-domain 1

EMDB-12690: 
ABC transporter NosDFY, nucleotide-free in lipid nanodisc, R-domain 2
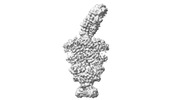
EMDB-12691: 
ABC transporter NosDFY, nucleotide-free in lipid nanodisc, R-domain 3

EMDB-12692: 
ABC transporter NosDFY E154Q, ATP-bound in lipid nanodisc

PDB-7o0y: 
ABC transporter NosDFY, nucleotide-free in GDN

PDB-7o10: 
ABC transporter NosDFY, nucleotide-free in GDN, R-domain 2

PDB-7o11: 
ABC transporter NosDFY, nucleotide-free in GDN, R-domain 1

PDB-7o12: 
ABC transporter NosDFY, AMPPNP-bound in GDN

PDB-7o14: 
ABC transporter NosDFY, nucleotide-free in lipid nanodisc, R-domain 1

PDB-7o15: 
ABC transporter NosDFY, nucleotide-free in lipid nanodisc, R-domain 2

PDB-7o16: 
ABC transporter NosDFY, nucleotide-free in lipid nanodisc, R-domain 3

PDB-7o17: 
ABC transporter NosDFY E154Q, ATP-bound in lipid nanodisc

EMDB-25008: 
Structure of the SARS-CoV-2 S 6P trimer in complex with the human neutralizing antibody Fab fragment, R40-1G8

PDB-7sc1: 
Structure of the SARS-CoV-2 S 6P trimer in complex with the human neutralizing antibody Fab fragment, R40-1G8

EMDB-13142: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation without nucleotides

EMDB-13143: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation with ADP/ATP

EMDB-13144: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation with ADP/ATP and rhodamine 6G

EMDB-13145: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in outward-facing conformation with ADP-orthovanadate/ATP

PDB-7p03: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation without nucleotides

PDB-7p04: 
Cryo-EM structure of Pdr5 from Saccharomyces cerevisiae in inward-facing conformation with ADP/ATP
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
