-検索条件
-検索結果
検索 (著者・登録者: kishi & ke)の結果104件中、1から50件目までを表示しています

EMDB-36905: 
SARS-CoV-2 BA.1 RBD with UT28-RD

EMDB-36906: 
SARS-CoV-2 BA.1 spike with UT28-RD

PDB-8k5g: 
Structure of the SARS-CoV-2 BA.1 RBD with UT28-RD

PDB-8k5h: 
Structure of the SARS-CoV-2 BA.1 spike with UT28-RD

EMDB-35029: 
SARS-CoV2 spike protein with ACE2, no ACE2 binding.

EMDB-35030: 
SARS-CoV2 spike protein with ACE2. 1 ACE2 bound form.

EMDB-35031: 
SARS-CoV2 spike protein with ACE2. 2 ACE2 bound form.

EMDB-35032: 
SARS-CoV2 spike protein with ACE2. 3 ACE2 bound form.

EMDB-35036: 
SARS-CoV2 spike protein with ACE2 decoy.no ACE2 decoy binding

EMDB-35037: 
SARS-CoV2 spike protein with ACE2 decoy. 1 ACE2 decoy bound form.

EMDB-35038: 
SARS-CoV2 spike protein with ACE2 decoy. 1 ACE2 decoy bound and 2 RBD up form.

EMDB-35039: 
SARS-CoV2 spike protein with ACE2 decoy. 2 ACE2 decoy bound form.

EMDB-35040: 
SARS-CoV2 spike protein with ACE2 decoy. 3 ACE2 decoy bound form.

EMDB-36345: 
RBD of SARS-CoV2 spike protein with ACE2 decoy

PDB-8jje: 
RBD of SARS-CoV2 spike protein with ACE2 decoy

EMDB-33785: 
Cryo-EM structure of the histamine-bound histamine H4 receptor and Gq complex

EMDB-33786: 
Cryo-EM structure of the imetit-bound histamine H4 receptor and Gq complex

PDB-7yfc: 
Cryo-EM structure of the histamine-bound histamine H4 receptor and Gq complex

PDB-7yfd: 
Cryo-EM structure of the imetit-bound histamine H4 receptor and Gq complex

EMDB-34530: 
Membrane protein A

EMDB-34531: 
Membrane protein B

EMDB-35713: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR1 H225F mutant in lipid nanodisc

PDB-8h86: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR1 in lipid nanodisc

PDB-8h87: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR2 in lipid nanodisc

PDB-8iu0: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR1 H225F mutant in lipid nanodisc

EMDB-34106: 
GroEL on EG-grid stored for 3 months after graphene oxidation
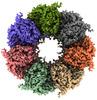
EMDB-34743: 
GroEL on Quantifoil grid

EMDB-34362: 
1 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus

EMDB-34363: 
2 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus

EMDB-34364: 
3 nucleotide-bound V1EG of V/A-ATPase from Thermus thermophilus.

EMDB-34365: 
2 sulfate-bound V1EG of V/A-ATPase from Thermus thermophilus.

EMDB-34366: 
1 sulfate and 1 ATP bound V1EG of V/A-ATPase from Thermus thermophilus.

PDB-8gxu: 
1 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus

PDB-8gxw: 
2 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus

PDB-8gxx: 
3 nucleotide-bound V1EG of V/A-ATPase from Thermus thermophilus.

PDB-8gxy: 
2 sulfate-bound V1EG of V/A-ATPase from Thermus thermophilus.

PDB-8gxz: 
1 sulfate and 1 ATP bound V1EG of V/A-ATPase from Thermus thermophilus.

EMDB-32159: 
GroEL on hydrophilized graphene grid (high particle density)
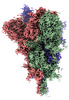
EMDB-32160: 
SARS-CoV-2 spike protein (1-up RBD) on EG-grid
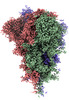
EMDB-32161: 
SARS-CoV-2 spike protein (1-up RBD) on Quantifoil grid
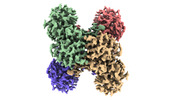
EMDB-32162: 
GAPDH on EG-grid

EMDB-34203: 
CryoEM structure of pentameric MotA from Aquifex aeolicus

PDB-8gqy: 
CryoEM structure of pentameric MotA from Aquifex aeolicus

EMDB-33251: 
Nucleotide-depleted F1 domain of FoF1-ATPase from Bacillus PS3, state1

EMDB-33252: 
Nucleotide-depleted F1 domain of FoF1-ATPase from Bacillus PS3, , state2

EMDB-33253: 
Nucleotide-depleted FoF1-ATPase from Bacillus PS3, state3

EMDB-33258: 
F1 domain of epsilon C-terminal domain deleted FoF1 from Bacillus PS3,state1,nucleotide depeleted

EMDB-33259: 
F1 domain of epsilon C-terminal domain deleted FoF1 from Bacillus PS3,state1,unisite condition

EMDB-33260: 
F1 domain of epsilon C-terminal domain deleted FoF1 from Bacillus PS3,state1,unisite condition

EMDB-33261: 
F1 domain of epsilon C-terminal domain deleted FoF1 from Bacillus PS3,state2,nucleotide depleted
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
