-検索条件
-検索結果
検索 (著者・登録者: jiang & hl)の結果全36件を表示しています

EMDB-44635: 
Inactive mu opioid receptor bound to Nb6, naloxone and NAM

PDB-9bjk: 
Inactive mu opioid receptor bound to Nb6, naloxone and NAM

EMDB-43011: 
Phosphorylated, ATP-bound, E1371Q human cystic fibrosis transmembrane conductance regulator (E1371Q-CFTR)

EMDB-43014: 
Phosphorylated, ATP-bound, inhibitor 172-bound E1371Q human cystic fibrosis transmembrane conductance regulator

EMDB-31824: 
Cryo-EM structure of the SV1-Gs complex.

EMDB-0744: 
Cryo-EM structure of a class A GPCR with G protein complex

EMDB-0745: 
Cryo-EM structure of CB1-G protein complex

PDB-6kpf: 
Cryo-EM structure of a class A GPCR with G protein complex

PDB-6kpg: 
Cryo-EM structure of CB1-G protein complex

EMDB-10213: 
Structure of the human DDB1-DDA1-DCAF15 E3 ubiquitin ligase bound to RBM39 and Indisulam

PDB-6sj7: 
Structure of the human DDB1-DDA1-DCAF15 E3 ubiquitin ligase bound to RBM39 and Indisulam

EMDB-20817: 
Cryo-EM structure of HIV-1 neutralizing antibody DH270 UCA3 in complex with CH848 10.17DT Env

EMDB-20818: 
Cryo-EM structure of HIV-1 neutralizing antibody DH270.6 in complex with CH848 10.17DT Env

EMDB-20819: 
Cryo-EM structure of vaccine-elicited HIV-1 neutralizing antibody DH270.mu1 in complex with CH848 10.17DT Env

EMDB-6442: 
CryoEM structure of endogenously assembled Tetrahymena telomerase holoenzyme at 9.4 Angstrom resolution

EMDB-6443: 
CryoEM structure of endogenously assembled Tetrahymena telomerase holoenzyme at 8.9 Angstrom resolution

EMDB-2503: 
Cryo-EM Structure of Isomeric Molluscan Hemocyanin Type 1 triggered by viral infection
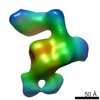
EMDB-5807: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (Teb1-f, conformation II)

EMDB-5808: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (Teb1-f, conformation III)
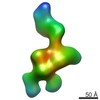
EMDB-5809: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (Teb1-f, conformation IV)

EMDB-5810: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (Teb1-f, conformation V)

EMDB-5811: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (Teb1-f, conformation VI)

EMDB-5812: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (Teb1-f, conformation VII)

EMDB-5813: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (Teb1-f, conformation VIII)

EMDB-5814: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (Teb1-f, conformation IX)

EMDB-5815: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (Teb1-f, conformation X)
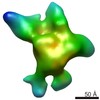
EMDB-5816: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (TERT-f, stable conformation)

EMDB-5817: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (TERT-f, subcomplex lacking p65 N-terminus)

EMDB-5818: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (TERT-f, subcomplex lacking Teb1)

EMDB-5819: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (TERT-f, subcomplex lacking p75-p19-p45)

EMDB-5820: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (f-Teb1C, stable conformation)

EMDB-5821: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (TERT-f, affinity labeled by anti-FLAG Fab)

EMDB-5822: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (Teb1-f, affinity labeled by anti-FLAG Fab)
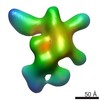
EMDB-5823: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (f-p50, affinity labeled by two copies of anti-FLAG Fab)

EMDB-5824: 
Negative-stain electron microscopy reconstruction of Tetrahymena telomerase (f-p50, affinity labeled by one copy of anti-FLAG Fab)

EMDB-5804: 
Three-dimensional structure of endogenously assembled Tetrahymena telomerase holoenzyme determined by negative-stain electron microscopy
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
