-検索条件
-検索結果
検索 (著者・登録者: jenkins & d)の結果102件中、1から50件目までを表示しています

EMDB-18990: 
CryoEM map of tau PHF sarkosyl-extracted from a human AD patient (associated with in situ tomography)

EMDB-50148: 
Tau PHF subtomogram average relating to CS1 extended data Figure 9A

EMDB-50152: 
Tau PHF subtomogram average relating to CS2 Figure 3i-j.

EMDB-50153: 
Tau PHF subtomogram average relating to CS3 extended data Figure 9c

EMDB-50155: 
Tau PHF subtomogram average relating to CS4 extended data Figure 9d

EMDB-50156: 
Tau PHF subtomogram average relating to CS5 extended data Figure 9b

EMDB-50157: 
Tau PHF subtomogram average relating to CS6 extended data Figure 9e

EMDB-50159: 
Tau PHF subtomogram average relating to CS7 extended data Figure 9f

EMDB-50160: 
Tau PHF subtomogram average relating to LOL1_PHF Figure 4g-h

EMDB-50161: 
Tau SF subtomogram average relating to LOL1_SF Figure 4g-h

EMDB-50162: 
Tau SF subtomogram average relating to LOL2_SF Figure 4i-j

EMDB-50358: 
In vitro-induced genome-releasing intermediate of Rhodobacter microvirus Ebor computed with C5 symmetry

EMDB-50356: 
Empty capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-50357: 
Native capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-50359: 
Rhodobacter microvirus Ebor attached to B10 host cell reconstructed by single particle analysis with applied C5 symmetry

EMDB-50360: 
Rhodobacter microvirus Ebor attached to the outer membrane vesicle

EMDB-50361: 
Rhodobacter microvirus Ebor attached to the host cell reconstructed by subtomogram averaging

PDB-9ffg: 
Empty capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

PDB-9ffh: 
Native capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-40954: 
ADP-bound Bcs1 (C7 symmetrized)

EMDB-41061: 
ATP-1 state of Bcs1 (C7 symmetrized)

EMDB-41095: 
ADP-bound Bcs1 (unsymmetrized)

EMDB-41148: 
Apo Bcs1, unsymmetrized

EMDB-41276: 
ATP-1 state of Bcs1 (unsymmetrized)
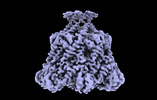
EMDB-41462: 
ATP-2 state of Bcs1 (C7 symmetrized)
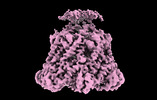
EMDB-41476: 
ATP-2 state of Bcs1 (unsymmetrized)

EMDB-41609: 
Bcs1 bound with ISP-ED

PDB-8t14: 
ADP-bound Bcs1 (C7 symmetrized)

PDB-8t5u: 
ATP-1 state of Bcs1 (C7 symmetrized)

PDB-8t7u: 
ADP-bound Bcs1 (unsymmetrized)

PDB-8tby: 
Apo Bcs1, unsymmetrized

PDB-8ti0: 
ATP-1 state of Bcs1 (unsymmetrized)

PDB-8tp1: 
ATP-2 state of Bcs1 (C7 symmetrized)

PDB-8tpl: 
ATP-2 state of Bcs1 (unsymmetrized)

EMDB-19132: 
Structure of dynein-2 intermediate chain DYNC2I2 (WDR34) in complex with dynein-2 heavy chain DYNC2H1.

EMDB-19133: 
Structure of dynein-2 intermediate chain DYNC2I1 (WDR60) in complex with the dynein-2 heavy chain DYNC2H1.

PDB-8rgg: 
Structure of dynein-2 intermediate chain DYNC2I2 (WDR34) in complex with dynein-2 heavy chain DYNC2H1.

PDB-8rgh: 
Structure of dynein-2 intermediate chain DYNC2I1 (WDR60) in complex with the dynein-2 heavy chain DYNC2H1.

EMDB-41008: 
Cryo-EM structure of the DHA bound FFA4-Gq complex

EMDB-41010: 
Cryo-EM structure of the Butyrate bound FFA2-Gq complex

EMDB-41013: 
Cryo-EM structure of the DHA bound FFA1-Gq complex

PDB-8t3q: 
Cryo-EM structure of the DHA bound FFA4-Gq complex

PDB-8t3s: 
Cryo-EM structure of the Butyrate bound FFA2-Gq complex

PDB-8t3v: 
Cryo-EM structure of the DHA bound FFA1-Gq complex

EMDB-43013: 
Myxococcus xanthus HEnc-K417N(A) protein shell with icosahedral T=1 symmetry

EMDB-43016: 
Myxococcus xanthus HEnc-K417N(A) protein shell with tetrahedral symmetry (12 pentamers, 4 hexamers)

EMDB-43037: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D3 symmetry (12 pentamers, 3 hexamers)

EMDB-43038: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D6 symmetry (12 pentamers, 8 hexamers)

EMDB-43039: 
Myxococcus xanthus HEnc-K417N(A) protein shell with C2 symmetry (12 pentamers, 9 hexamers)

EMDB-43040: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D3 symmetry (12 pentamers, 11 hexamers)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
